تحميل سريع للبيانات على دفعات لملفات BigWig التي تحتوي على بيانات المسار اللاجيني والتسلسلات المقابلة المدعومة بواسطة وحدة معالجة الرسومات لتطبيقات التعلم العميق.
يعتمد برنامج Bigwig-loader بشكل أساسي على مكتبة Rapidsai kvikio وcupy، وكلاهما من الأفضل تثبيتهما باستخدام conda/mamba. يمكن الآن أيضًا تثبيت Bigwig-loader باستخدام conda/mamba. لإنشاء بيئة جديدة مع تثبيت برنامج bigwig-loader:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderأو أضف هذا إلى ملف Environment.yml الخاص بك:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderوالتحديث:
mamba env update -f environment.ymlيمكن أيضًا تثبيت Bigwig-loader باستخدام النقطة في بيئة تم تثبيت مكتبة Rapidsai kvikio وcupy عليها بالفعل:
pip install bigwig-loaderلقد قمنا بتغليف BigWigDataset في مجموعة بيانات PyTorch القابلة للتكرار والتي يمكنك استخدامها مباشرة:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () يمكن استيراد كائن Dataset غير محدد الإطار من bigwig_loader.dataset . يقوم كائن مجموعة البيانات هذا بإرجاع موتر كوبي. تلتزم موترات Cupy بواجهة صفيف cuda ويمكن تحويلها بدون نسخة إلى JAX أو Tensorflow.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)راجع دليل الأمثلة لمزيد من الأمثلة.
تهدف هذه المكتبة إلى تحميل دفعات من البيانات بنفس الأبعاد، مما يسمح ببعض الافتراضات التي يمكنها تسريع عملية التحميل. كما يتبين من المخطط أدناه، عند تحميل كمية صغيرة من البيانات، يكون pyBigWig سريعًا جدًا، ولكنه لا يستغل الطبيعة المجمعة لتحميل البيانات للتعلم الآلي.
في المعيار أدناه، أنشأنا أيضًا أدوات تحميل بيانات PyTorch (باستخدام set_start_method('spawn')) باستخدام pyBigWig للمقارنة بالسيناريو الواقعي حيث سيتم استخدام وحدات المعالجة المركزية المتعددة لكل وحدة معالجة رسومات. نرى أن إنتاجية محمل بيانات وحدة المعالجة المركزية لا ترتفع خطيًا مع عدد وحدات المعالجة المركزية، وبالتالي يصبح من الصعب الحصول على الإنتاجية المطلوبة للحفاظ على وحدة معالجة الرسومات، وتدريب الشبكة العصبية، مشبعة أثناء خطوات التعلم.
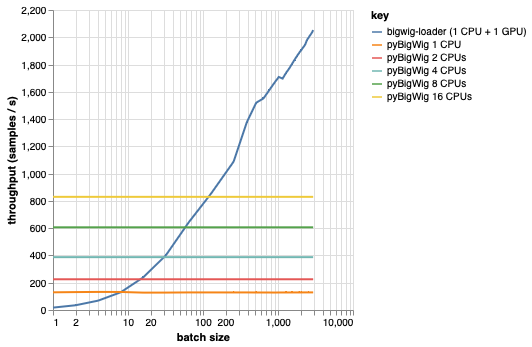
هذه هي المشكلة التي يحلها المحمل الكبير. هذا مثال لكيفية استخدام bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml في هذه البيئة، يجب أن تكون قادرًا على تشغيل pytest -v ورؤية نجاح الاختبارات. ملاحظة: أنت بحاجة إلى وحدة معالجة الرسومات (GPU) لاستخدام أداة التحميل الكبيرة!
يرشدك هذا القسم خلال الخطوات اللازمة لإضافة وظائف جديدة. إذا كان هناك أي شيء غير واضح، يرجى فتح موضوع.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install لتثبيت خطافات الالتزام المسبقالاختبارات موجودة في دليل الاختبارات. أحد أهم الاختبارات هو test_against_pybigwig الذي يتأكد من أنه إذا كان هناك خطأ في pyBigWIg، فهو موجود أيضًا في bigwig-loader.
pytest -vv .عندما تصبح برامج تشغيل github المزودة بوحدة معالجة الرسومات متاحة، نود أيضًا إجراء هذه الاختبارات في CI. لكن في الوقت الحالي، يمكنك تشغيلها محليًا.
إذا كنت تستخدم هذه المكتبة، فكر في الاستشهاد بما يلي:
ريتيل، ويورين سيباستيان، وأندرياس بوهلمان، وجوش تشيو، وأندرياس ستيفن، ودجورك أرني كليفرت. "أداة تحميل بيانات سريعة للتعلم الآلي للمسارات اللاجينية من ملفات BigWig." المعلوماتية الحيوية 40، لا. 1 (1 يناير 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}