توفر هذه الحزمة نماذج U-net مدربة لتجزئة الرئة. في الوقت الحالي، تتوفر أربعة نماذج:
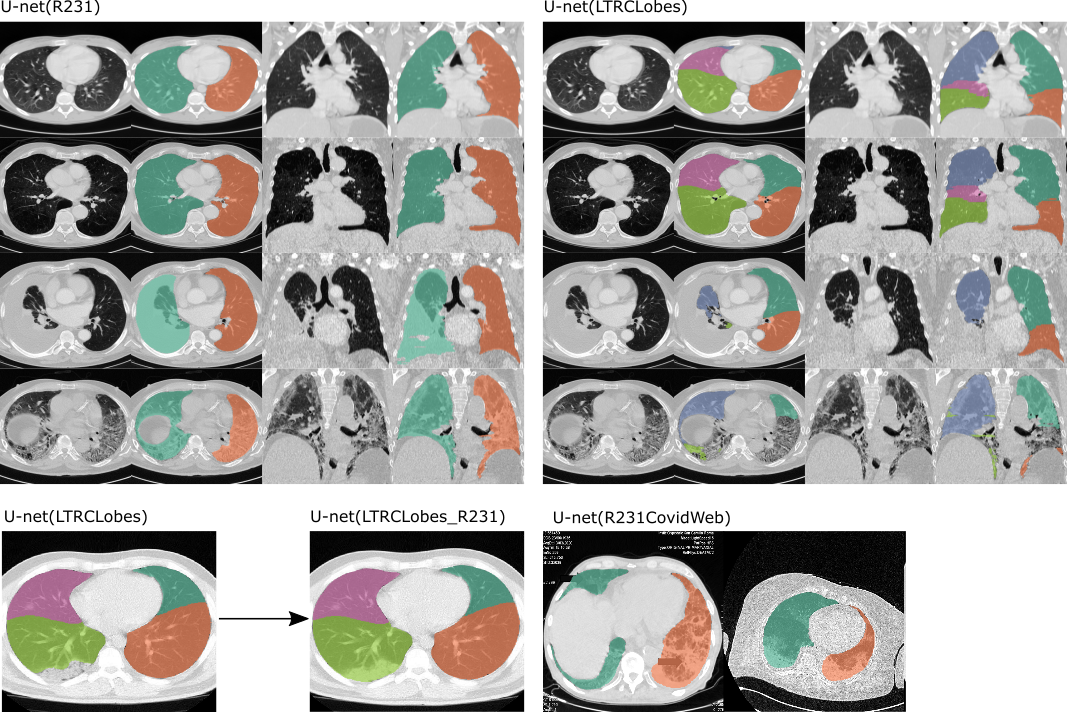
U-net(R231): تم تدريب هذا النموذج على مجموعة بيانات كبيرة ومتنوعة تغطي نطاقًا واسعًا من المتغيرات البصرية. يقوم النموذج بالتجزئة على شرائح فردية، ويستخرج الرئة اليمنى واليسرى بشكل منفصل ويتضمن الجيوب الهوائية والأورام والانصبابات. لن يتم تضمين القصبة الهوائية في تجزئة الرئة. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRClobes): تم تدريب هذا النموذج على مجموعة فرعية من مجموعة بيانات LTRC. يقوم النموذج بتجزئة فصوص الرئة الفردية ولكنه ينتج أداءً محدودًا عند وجود أمراض كثيفة أو عندما لا تكون الشقوق مرئية في كل شريحة.
U-net(LTRCLobes_R231): سيؤدي هذا إلى تشغيل نموذج R231 وLTRCLobes ودمج النتائج. سيتم ملء السلبيات الكاذبة من LTRCLobes بواسطة تنبؤات R231 وتعيينها إلى تسمية مجاورة. ستتم إزالة النتائج الإيجابية الكاذبة من LTRCLobes. عملية الدمج مكثفة من الناحية الحسابية ويمكن أن تستغرق ما يصل إلى عدة دقائق لكل وحدة تخزين، اعتمادًا على البيانات والنتائج.
يو نت (R231CovidWeb)
أمثلة على النموذجين المطبقين . اليسار: U-net (R231)، سيميز بين الرئة اليسرى واليمنى ويشمل مناطق شديدة الكثافة مثل الانصبابات (الصف الثالث)، الورم أو التليف الشديد (الصف الرابع). على اليمين: U-net (LTRLobes)، سوف يميز بين فصوص الرئة ولكنه لن يشمل المناطق شديدة الكثافة. سوف يقوم LTRCLobes_R231 بدمج نتائج LTRCLobes وR231. تم تدريب R231CovidWeb باستخدام بيانات كوفيد-19 الإضافية.
دلالات الإخراج :
نموذجان للتسمية (يسار-يمين):
1 = الرئة اليمنى
2 = الرئة اليسرى
خمسة نماذج تسمية (فصوص الرئة):
1 = الفص العلوي الأيسر
2 = الفص السفلي الأيسر
3 = الفص العلوي الأيمن
4 = الفص الأوسط الأيمن
5 = الفص السفلي الأيمن
لمزيد من الأبحاث المثيرة حول بيانات التصوير المقطعي المحوسب للرئة، قم بزيارة الموقع الإلكتروني لمجموعتنا البحثية: https://www.cir.meduniwien.ac.at/research/lung/
إذا كنت تستخدم هذا الكود أو أحد النماذج المدربة في عملك يرجى الرجوع إلى:
هوفماننجر، جيه، براير، إف، بان، جيه وآخرون. يعد تجزئة الرئة التلقائية في التصوير الروتيني في المقام الأول مشكلة تنوع البيانات، وليست مشكلة منهجية. يورو راديول إكسب 4، 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
تحتوي هذه الورقة على وصف تفصيلي لمجموعة البيانات المستخدمة، وتقييم شامل لنموذج U-net(R231)، ومقارنة بالطرق المرجعية.
pip install lungmask
أو
pip install git+https://github.com/JoHof/lungmask
على نظام التشغيل Windows، اعتمادًا على الإعداد الخاص بك، قد يكون من الضروري تثبيت torch مسبقًا: https://pytorch.org
يختلف وقت التشغيل بين الاستدلال المدعوم من وحدة المعالجة المركزية (CPU) ووحدة معالجة الرسومات (GPU) بشكل كبير. باستخدام وحدة معالجة الرسومات، تستغرق معالجة وحدة التخزين عدة ثوانٍ فقط، بينما يستغرق استخدام وحدة المعالجة المركزية فقط عدة دقائق. للاستفادة من وحدة معالجة الرسومات، تأكد من أن تثبيت الشعلة لديك يدعم CUDA. في حالة نفاد ذاكرة cuda، قم بتقليل حجم الدُفعة إلى 1 باستخدام الوسيطة الاختيارية --batchsize 1
lungmask INPUT OUTPUT
إذا أشار INPUT إلى ملف، فستتم معالجة الملف. إذا أشار INPUT إلى دليل، فسيتم البحث في الدليل عن سلسلة DICOM. سيتم استخدام أكبر حجم تم العثور عليه (من حيث عدد الأكسيل) لحساب قناع الرئة. OUTPUT هو اسم ملف الإخراج. جميع صيغ ITK مدعومة.
اختر نموذجًا:
سيتم استخدام U-net(R231) كإعداد افتراضي. ومع ذلك، يمكنك تحديد نموذج بديل مثل LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
للحصول على خيارات إضافية اكتب:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
يجب أن يكون input_image كائنًا SimpleITK.
قم بتحميل نموذج بديل مثل ذلك:
inferer = LMInferer(modelname="R231CovidWeb")
لاستخدام قدرة دمج النموذج لاستخدام (على سبيل المثال LTRCLobes_R231):
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
اعتبارًا من الإصدار 0.2.9، أصبح المصفوفات numpy مدعومة كوحدات تخزين مدخلات. يفترض هذا الوضع أن مصفوفة الإدخال numpy لها التنسيق التالي لكل محور:
النموذج يعمل على شرائح كاملة فقط. يجب أن تظهر الشريحة المراد معالجتها الرئة الكاملة ويجب أن تكون الرئة محاطة بالأنسجة حتى يتم تقسيمها. ومع ذلك، فإن النموذج مستقر تمامًا بالنسبة للحالات ذات مجال الرؤية المقصوص طالما أن الرئة محاطة بالأنسجة.
lungmask INPUT OUTPUT --modelname R231CovidWeb
يعمل نموذج U-net(R231) العادي بشكل جيد للغاية مع عمليات التصوير المقطعي المحوسب لفيروس كوفيد-19. ومع ذلك، غالبًا ما يتم اقتصاص مجموعات الشرائح وتقارير الحالة من الويب أو شرحها أو تشفيرها بتنسيقات صور عادية بحيث لا يمكن تقدير قيم وحدة هونسفيلد الأصلية (HU) إلا. تمت تعزيز بيانات التدريب الخاصة بنموذج U-net(R231CovidWeb) بشرائح كوفيد-19 التي تم تعيينها مرة أخرى من تنسيقات التصوير العادية إلى HU. تم جمع البيانات وإعدادها بواسطة MedSeg (http://medicalsegmentation.com/covid19/). في حين أظهر U-net (R231) العادي نتائج جيدة جدًا لهذه الصور، فقد تكون هناك حالات سينتج عنها هذا النموذج تحسينات طفيفة في التجزئة. لاحظ أنه يتعين عليك إعادة تعيين الصور إلى HU عند استخدام الصور من الويب. يصف منشور المدونة هذا كيف يمكنك القيام بذلك. وبدلاً من ذلك، يمكنك تعيين علامة --noHU . 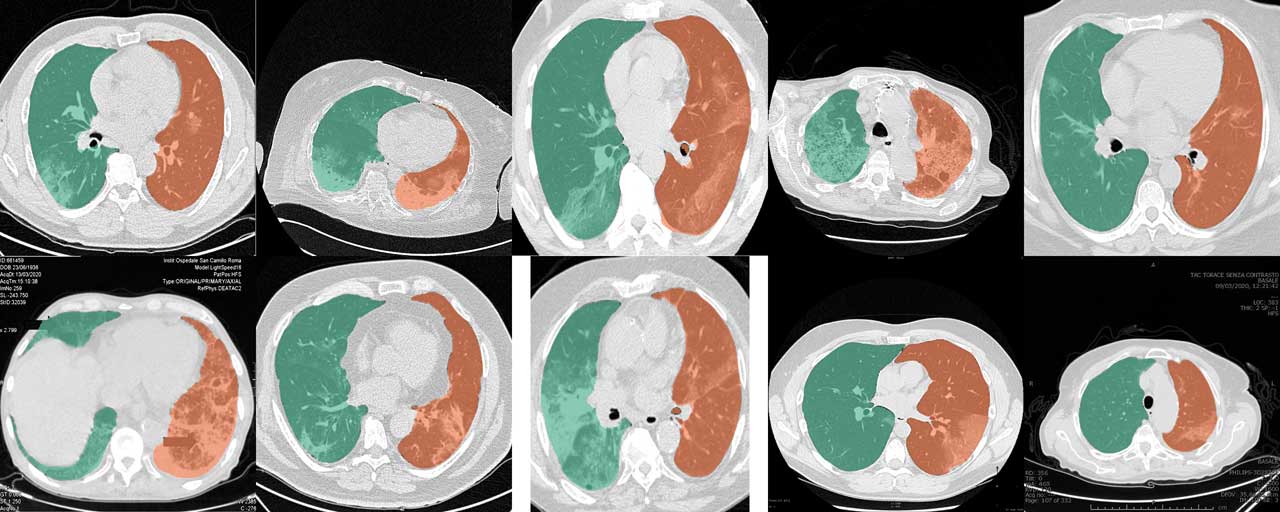
تتوفر هذه الميزة فقط في الإصدارات بين 0.2.5 و0.2.14. اعتبارًا من الإصدار 0.2.5، يتم دعم هذه الصور. استخدم علامة --noHU إذا كنت تقوم بمعالجة الصور غير المشفرة في HU. ضع في اعتبارك أنه تم تدريب النماذج على الأشعة المقطعية المناسبة المشفرة في HU. قد لا تكون النتائج على الصور المقتطعة والمشروحة والمرتفعة جدًا والمنخفضة جدًا موثوقة للغاية. عند استخدام علامة --noHU ، يمكن معالجة شرائح مفردة فقط.