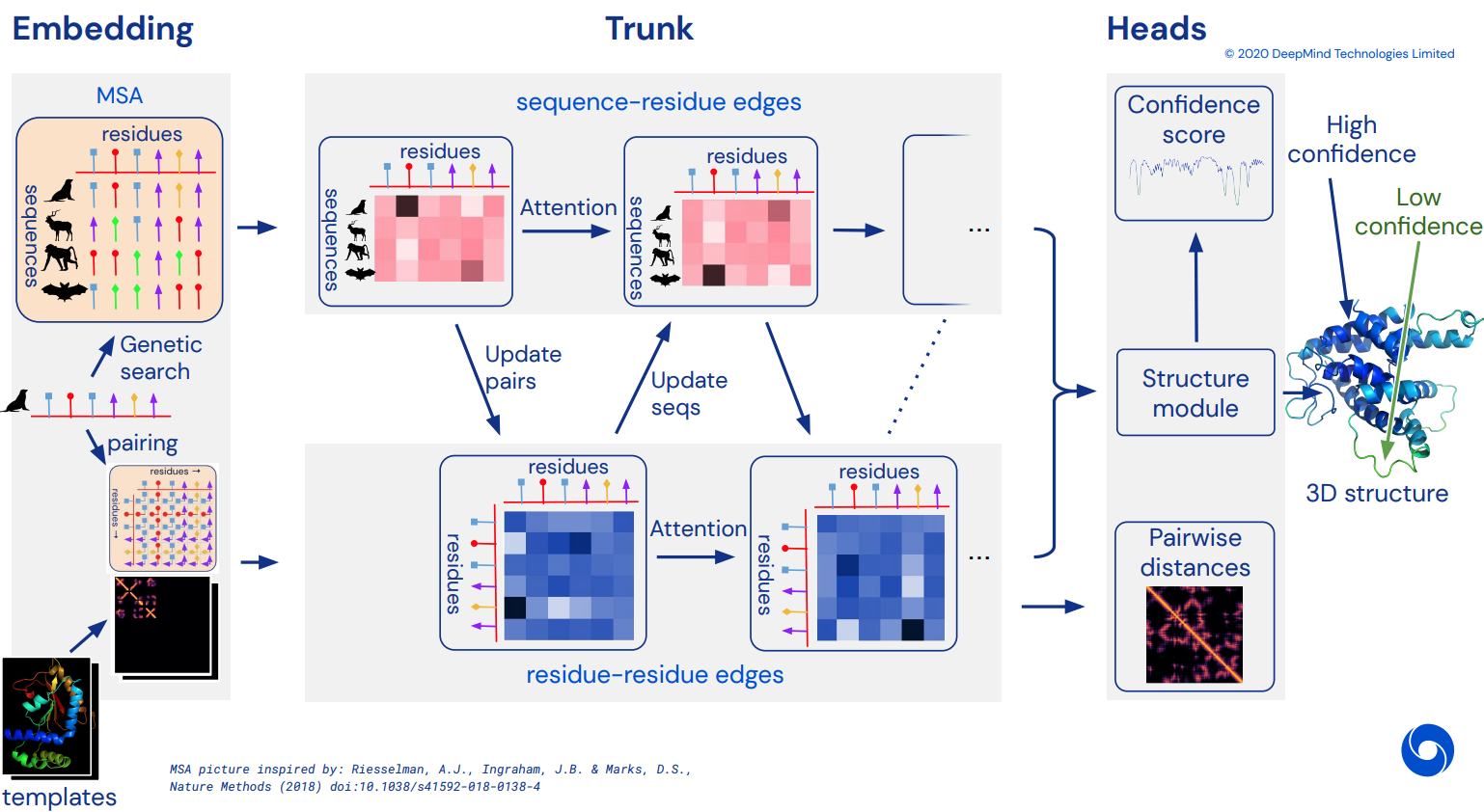
لتصبح في النهاية تطبيق Pytorch غير رسمي لـ Alphafold2، شبكة الاهتمام المذهلة التي حلت CASP14. سيتم تنفيذه تدريجيًا مع إصدار المزيد من التفاصيل حول البنية.
بمجرد تكرار هذا، أنوي طي جميع تسلسلات الأحماض الأمينية المتاحة هناك في السيليكو وإصدارها كسيل أكاديمي لمزيد من العلوم. إذا كنت مهتمًا بجهود التكرار، فيرجى زيارة #alphafold على قناة Discord هذه
تحديث: قامت شركة Deepmind بفتح الكود الرسمي في Jax، بالإضافة إلى الأوزان! سيتم الآن توجيه هذا المستودع نحو ترجمة pytorch مباشرة مع بعض التحسينات على التشفير الموضعي
فيديو أركسيف إنسايتس
$ pip install alphafold2-pytorchأبلغت lhatsk عن تدريب صندوق معدّل لهذا المستودع، باستخدام نفس الإعداد مثل trRosetta، مع نتائج تنافسية
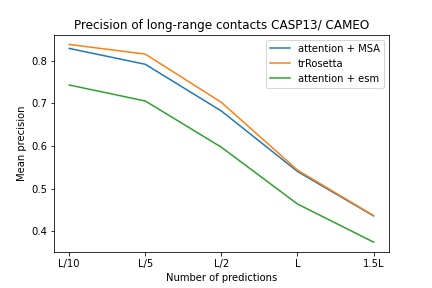
blue used the the trRosetta input (MSA -> potts -> axial attention), green used the ESM embedding (only sequence) -> tiling -> axial attention - lhatsk
توقع مخطط التوزيع، مثل Alphafold-1، ولكن مع الاهتمام
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
reversible = False # set this to True for fully reversible self / cross attention for the trunk
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda () # AA length of 128
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda () # MSA doesn't have to be the same length as primary sequence
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) يمكنك أيضًا تشغيل التنبؤ للزوايا، عن طريق تمرير predict_angles = True على init. المثال أدناه سيكون مكافئًا لـ trRosetta ولكن مع الانتباه الذاتي/التقاطعي.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_angles = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram , theta , phi , omega = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
)
# distogram - (1, 128, 128, 37),
# theta - (1, 128, 128, 25),
# phi - (1, 128, 128, 13),
# omega - (1, 128, 128, 25) تقترح ورقة فابيان الأخيرة أن تغذية الإحداثيات بشكل متكرر مرة أخرى إلى محول SE3، مع تقاسم الوزن، قد ينجح. لقد قررت التنفيذ بناءً على هذه الفكرة، على الرغم من أن كيفية عملها لا تزال غير واضحة.
يمكنك أيضًا استخدام E(n)-Transformer أو EGNN للتحسين الهيكلي.
تحديث: أظهر مختبر بيكر أن البنية الشاملة بدءًا من التسلسل وتضمينات MSA إلى محولات SE3 يمكن أن تكون أفضل من trRosetta وتسد الفجوة مع Alphafold2. سنستخدم محول الرسم البياني، الذي يعمل على تضمينات الجذع، لإنشاء المجموعة الأولية من الإحداثيات التي سيتم إرسالها إلى الشبكة المتساوية. (تم تأكيد ذلك أيضًا من قبل كوستا وآخرين في عملهم على استخراج الإحداثيات ثلاثية الأبعاد من تضمينات محولات MSA في ورقة بحثية سبقت مختبر بيكر)
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
structure_module_type = 'se3' , # use SE3 Transformer - if set to False, will use E(n)-Transformer, Victor and Max Welling's new paper
structure_module_dim = 4 , # se3 transformer dimension
structure_module_depth = 1 , # depth
structure_module_heads = 1 , # heads
structure_module_dim_head = 16 , # dimension of heads
structure_module_refinement_iters = 2 , # number of equivariant coordinate refinement iterations
structure_num_global_nodes = 1 # number of global nodes for the structure module, only works with SE3 transformer
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue الافتراض الأساسي هو أن الجذع يعمل على مستوى البقايا، ثم يشكل المستوى الذري لوحدة البنية، سواء كانت محولات SE3 أو E(n)-Transformer أو EGNN التي تقوم بالتحسين. الإعدادات الافتراضية لهذه المكتبة هي الذرات الثلاث الأساسية (C، Ca، N)، ولكن يمكنك تكوينها لتشمل أي ذرة أخرى تريدها، بما في ذلك Cb والسلاسل الجانبية.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
atoms = 'backbone-with-cbeta'
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 4, 3) <-- 4 atoms per residue (C, Ca, N, Cb) تشمل الاختيارات الصالحة atoms ما يلي:
backbone - 3 ذرات العمود الفقري (C، Ca، N) [افتراضي]backbone-with-cbeta - 3 ذرات العمود الفقري وC بيتاbackbone-with-oxygen - 3 ذرات العمود الفقري والأكسجين من الكربوكسيلbackbone-with-cbeta-and-oxygen - 3 ذرات العمود الفقري مع C بيتا والأكسجينall - العمود الفقري وجميع الذرات الأخرى من السلسلة الجانبيةيمكنك أيضًا تمرير موتر الشكل (14) لتحديد الذرات التي ترغب في تضمينها
السابق.
atoms = torch . tensor ([ 1 , 1 , 1 , 1 , 1 , 1 , 0 , 1 , 0 , 0 , 0 , 0 , 0 , 1 ])يوفر لك هذا المستودع ملحقًا سهلاً للشبكة من خلال عمليات التضمين المدربة مسبقًا من Facebook AI. يحتوي على أغلفة لمحولات ESM أو MSA أو محول البروتين المدربة مسبقًا.
هناك بعض المتطلبات الأساسية. ستحتاج إلى التأكد من تثبيت مكتبة Nvidia apex، حيث تستفيد المحولات المدربة مسبقًا من بعض العمليات المدمجة.
أو يمكنك محاولة تشغيل البرنامج النصي أدناه
git clone https://github.com/NVIDIA/apex
cd apex
pip install -v --disable-pip-version-check --no-cache-dir --global-option= " --cpp_ext " --global-option= " --cuda_ext " ./ بعد ذلك، سيتعين عليك ببساطة استيراد مثيل Alphafold2 الخاص بك وتغليفه باستخدام ESMEmbedWrapper أو MSAEmbedWrapper أو ProtTranEmbedWrapper وسيتولى تضمين كل من التسلسل ومحاذاة التسلسلات المتعددة لك (وعرضها على الأبعاد كما هو محدد في نموذج). لا شيء يحتاج إلى تغيير باستثناء إضافة المجمع.
import torch
from alphafold2_pytorch import Alphafold2
from alphafold2_pytorch . embeds import MSAEmbedWrapper
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64
)
model = MSAEmbedWrapper (
alphafold2 = alphafold2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) افتراضيًا، حتى لو قام المجمّع بتزويد صندوق السيارة بالتسلسل وتضمينات MSA، فسيتم جمعها بتضمينات الرمز المميز المعتادة. إذا كنت تريد تدريب Alphafold2 بدون تضمينات الرمز المميز (تعتمد فقط على عمليات التضمين المدربة مسبقًا)، فستحتاج إلى تعيين disable_token_embed على True في Alphafold2 init.
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
disable_token_embed = True
) تشير ورقة بحثية كتبها جينبو شو إلى أن المرء لا يحتاج إلى تجميع المسافات، ويمكنه بدلاً من ذلك التنبؤ بالمتوسط والانحراف المعياري مباشرة. يمكنك استخدام هذا عن طريق تشغيل علامة predict_real_value_distances ، وفي هذه الحالة، سيكون للتنبؤ بالمسافة الذي تم إرجاعه بُعد 2 للمتوسط والانحراف المعياري على التوالي.
إذا تم أيضًا تشغيل predict_coords ، فسيقبل MDS تنبؤات الانحراف المتوسط والمعياري مباشرة دون الحاجة إلى حساب ذلك من صناديق الرسم التخطيطي.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
predict_real_value_distances = True , # set this to True
structure_module_type = 'se3' ,
structure_module_dim = 4 ,
structure_module_depth = 1 ,
structure_module_heads = 1 ,
structure_module_dim_head = 16 ,
structure_module_refinement_iters = 2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue يمكنك إضافة كتل تلافيفية لكل من التسلسل الأساسي بالإضافة إلى MSA، وذلك ببساطة عن طريق تعيين وسيطة كلمة رئيسية إضافية use_conv = True
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)تتبع الحبات التلافيفية خطى هذه الورقة، حيث تجمع بين حبات 1d و2d في كتلة واحدة تشبه شبكة إعادة الشبكة. يمكنك تخصيص النوى بشكل كامل على هذا النحو.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
conv_seq_kernels = (( 9 , 1 ), ( 1 , 9 ), ( 3 , 3 )), # kernels for N x N primary sequence
conv_msa_kernels = (( 1 , 9 ), ( 3 , 3 )), # kernels for {num MSAs} x N MSAs
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) يمكنك أيضًا إجراء توسيع الدورة باستخدام وسيطة كلمة رئيسية إضافية. التمدد الافتراضي هو 1 لجميع الطبقات.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
dilations = ( 1 , 3 , 5 ) # cycle between dilations of 1, 3, 5
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) أخيرًا، بدلاً من اتباع نمط التلافيف، والانتباه الذاتي، والانتباه المتبادل لكل عمق متكرر، يمكنك تخصيص أي ترتيب تريده باستخدام الكلمة الأساسية custom_block_types
السابق. شبكة حيث تقوم في الغالب بالتلافيف أولاً، متبوعة بالانتباه الذاتي + كتل الانتباه المتبادل
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
heads = 8 ,
dim_head = 64 ,
custom_block_types = (
* (( 'conv' ,) * 6 ),
* (( 'self' , 'cross' ) * 6 )
)
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) يمكنك التدرب باستخدام برنامج Microsoft Deepspeed Sparse Attention، ولكن سيتعين عليك تحمل عملية التثبيت. إنها خطوتين.
أولاً، تحتاج إلى تثبيت Deepspeed مع Sparse Attention
$ sh install_deepspeed.sh بعد ذلك، تحتاج إلى تثبيت حزمة النقاط triton
$ pip install tritonإذا نجح كلا الأمرين أعلاه، فيمكنك الآن التدرب مع الانتباه المتناثر!
للأسف، يتم دعم الاهتمام المتناثر فقط من أجل الاهتمام بالذات، وليس الانتباه المتقاطع. سأقدم حلاً مختلفًا لجعل الانتباه متقاطعًا.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
max_seq_len = 2048 , # the maximum sequence length, this is required for sparse attention. the input cannot exceed what is set here
sparse_self_attn = ( True , False ) * 6 # interleave sparse and full attention for all 12 layers
). cuda ()لقد أضفت أيضًا أحد أفضل متغيرات الانتباه الخطي، على أمل تقليل عبء الحضور المتقاطع. أنا شخصياً لم أجد أن Performanceer يعمل بشكل جيد، ولكن بما أنهم أبلغوا في الورقة عن بعض الأرقام الجيدة لمعايير البروتين، اعتقدت أنني سأدرجها وأسمح للآخرين بالتجربة.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = True # simply set this to True to use Performer for all cross attention
). cuda ()يمكنك أيضًا تحديد الطبقات المحددة التي ترغب في استخدام الاهتمام الخطي بها عن طريق تمرير صف من نفس طول العمق
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = ( True , False ) * 3 # interleave linear and full attention
). cuda ()تقترح هذه الورقة أنه إذا كان لديك استعلامات أو سياقات تحتوي على محاور محددة (مثل صورة)، فيمكنك تقليل مقدار الاهتمام المطلوب عن طريق حساب المتوسط عبر تلك المحاور (الارتفاع والعرض) وتسلسل المحاور المتوسطة في تسلسل واحد. يمكنك تشغيل هذا كأسلوب لحفظ الذاكرة للانتباه المتقاطع، خاصة للتسلسل الأساسي.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_kron_primary = True # make sure primary sequence undergoes the kronecker operator during cross attention
). cuda () يمكنك أيضًا تطبيق نفس العامل على MSAs أثناء تقاطع الانتباه باستخدام علامة cross_attn_kron_msa ، إذا كانت MSAs الخاصة بك محاذية وبنفس العرض.
ما يجب القيام به
لتوفير الذاكرة لجذب الانتباه، يمكنك تعيين نسبة ضغط للمفتاح/القيم، باتباع المخطط الموضح في هذه الورقة. عادة ما تكون نسبة الضغط 2-4 مقبولة.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_compress_ratio = 3
). cuda ()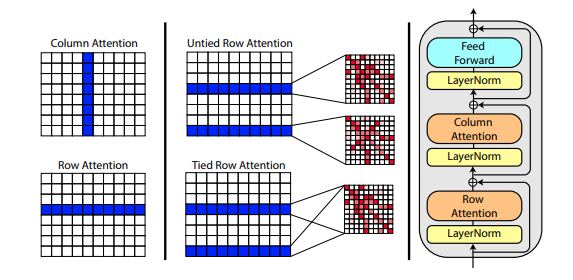
تقترح ورقة بحثية جديدة كتبها روشان راو استخدام الانتباه المحوري للتدريب المسبق على MSA. نظرًا للنتائج القوية، سيستخدم هذا المستودع نفس المخطط في الجذع، خصيصًا للانتباه الذاتي لـ MSA.
يمكنك أيضًا ربط انتباه صف MSA باستخدام الإعداد msa_tie_row_attn = True عند تهيئة Alphafold2 . ومع ذلك، لاستخدام هذا، يجب عليك التأكد من أنه إذا كان لديك عدد غير متساوٍ من MSA لكل تسلسل أساسي، فسيتم تعيين قناع MSA بشكل صحيح على False للصفوف غير المستخدمة.
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
msa_tie_row_attn = True # just set this to true
)تتم أيضًا معالجة القالب إلى حد كبير من خلال الاهتمام المحوري، مع الاهتمام المتبادل على طول عدد أبعاد القوالب. يتبع هذا إلى حد كبير نفس المخطط كما هو الحال في نهج الاهتمام الشامل الأخير لتصنيف الفيديو كما هو موضح هنا.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randint ( 0 , 37 , ( 1 , 2 , 16 , 3 )). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_coors = templates_coors ,
templates_mask = templates_mask
)إذا كانت معلومات السلسلة الجانبية موجودة أيضًا، في شكل متجه الوحدة بين إحداثيات C وC-alpha لكل وحدة بقايا، فيمكنك أيضًا تمريرها على النحو التالي.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randn ( 1 , 2 , 16 , 3 ). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
templates_sidechains = torch . randn ( 1 , 2 , 16 , 3 ). cuda () # unit vectors of difference of C and C-alpha coordinates
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_mask = templates_mask ,
templates_coors = templates_coors ,
templates_sidechains = templates_sidechains
)لقد قمت بإعداد إعادة تنفيذ SE3 Transformer، كما أوضح فابيان فوكس في إحدى المدونات التخمينية.
بالإضافة إلى ذلك، تستخدم ورقة بحثية جديدة من Victor and Welling ميزات ثابتة لتكافؤ E(n)، حيث تصل إلى SOTA وتتفوق على SE3 Transformer في عدد من المعايير، بينما تكون أسرع بكثير. لقد أخذت الأفكار الرئيسية من هذه الورقة وقمت بتعديلها لتصبح محولة (إضافة اهتمام لكل من الميزات وتحديثات التنسيق).
تم دمج جميع الشبكات الثلاث المتساوية المذكورة أعلاه وهي متاحة للاستخدام في المستودع لتحسين الإحداثيات الذرية بمجرد تعيين معلمة تشعبية واحدة structure_module_type .
محول se3 SE3
egnn إجن
en E(n)-محول
ومما يثير اهتمام القراء، أنه تم أيضًا التحقق من صحة كل إطار من الأطر الثلاثة من قبل الباحثين بشأن المشكلات ذات الصلة.
$ python setup.py test ستستخدم هذه المكتبة العمل الرائع الذي قام به جوناثان كينج في هذا المستودع. شكرا جوناثان!
لدينا أيضًا بيانات MSA، تبلغ قيمتها الإجمالية 3.5 تيرابايت تقريبًا، وقد تم تنزيلها واستضافتها بواسطة Archivist، الذي يملك مشروع The-Eye. (يستضيفون أيضًا البيانات والنماذج الخاصة بـ Eleuther AI) يرجى النظر في التبرع إذا وجدتها مفيدة.
$ curl -s https://the-eye.eu/eleuther_staging/globus_stuffs/tree.txthttps://xukui.cn/alphafold2.html
https://moalquraishi.wordpress.com/2020/12/08/alphafold2-casp14-it-feels-like-ones-child-has-left-home/

https://www.biorxiv.org/content/10.1101/2020.12.10.419994v1.full.pdf
https://pubmed.ncbi.nlm.nih.gov/33637700/
عرض tFold، من مختبرات Tencent AI
cd downloads_folder > pip install pyrosetta_wheel_filename.whlOpenMM العنبر
@misc { unpublished2021alphafold2 ,
title = { Alphafold2 } ,
author = { John Jumper } ,
year = { 2020 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @article { Rao2021.02.12.430858 ,
author = { Rao, Roshan and Liu, Jason and Verkuil, Robert and Meier, Joshua and Canny, John F. and Abbeel, Pieter and Sercu, Tom and Rives, Alexander } ,
title = { MSA Transformer } ,
year = { 2021 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/02/13/2021.02.12.430858 } ,
journal = { bioRxiv }
} @article { Rives622803 ,
author = { Rives, Alexander and Goyal, Siddharth and Meier, Joshua and Guo, Demi and Ott, Myle and Zitnick, C. Lawrence and Ma, Jerry and Fergus, Rob } ,
title = { Biological Structure and Function Emerge from Scaling Unsupervised Learning to 250 Million Protein Sequences } ,
year = { 2019 } ,
doi = { 10.1101/622803 } ,
publisher = { Cold Spring Harbor Laboratory } ,
journal = { bioRxiv }
} @article { Elnaggar2020.07.12.199554 ,
author = { Elnaggar, Ahmed and Heinzinger, Michael and Dallago, Christian and Rehawi, Ghalia and Wang, Yu and Jones, Llion and Gibbs, Tom and Feher, Tamas and Angerer, Christoph and Steinegger, Martin and BHOWMIK, DEBSINDHU and Rost, Burkhard } ,
title = { ProtTrans: Towards Cracking the Language of Life{textquoteright}s Code Through Self-Supervised Deep Learning and High Performance Computing } ,
elocation-id = { 2020.07.12.199554 } ,
year = { 2021 } ,
doi = { 10.1101/2020.07.12.199554 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554.full.pdf } ,
journal = { bioRxiv }
} @misc { king2020sidechainnet ,
title = { SidechainNet: An All-Atom Protein Structure Dataset for Machine Learning } ,
author = { Jonathan E. King and David Ryan Koes } ,
year = { 2020 } ,
eprint = { 2010.08162 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { alquraishi2019proteinnet ,
title = { ProteinNet: a standardized data set for machine learning of protein structure } ,
author = { Mohammed AlQuraishi } ,
year = { 2019 } ,
eprint = { 1902.00249 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { gomez2017reversible ,
title = { The Reversible Residual Network: Backpropagation Without Storing Activations } ,
author = { Aidan N. Gomez and Mengye Ren and Raquel Urtasun and Roger B. Grosse } ,
year = { 2017 } ,
eprint = { 1707.04585 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CV }
} @misc { fuchs2021iterative ,
title = { Iterative SE(3)-Transformers } ,
author = { Fabian B. Fuchs and Edward Wagstaff and Justas Dauparas and Ingmar Posner } ,
year = { 2021 } ,
eprint = { 2102.13419 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { satorras2021en ,
title = { E(n) Equivariant Graph Neural Networks } ,
author = { Victor Garcia Satorras and Emiel Hoogeboom and Max Welling } ,
year = { 2021 } ,
eprint = { 2102.09844 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { su2021roformer ,
title = { RoFormer: Enhanced Transformer with Rotary Position Embedding } ,
author = { Jianlin Su and Yu Lu and Shengfeng Pan and Bo Wen and Yunfeng Liu } ,
year = { 2021 } ,
eprint = { 2104.09864 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CL }
} @article { Gao_2020 ,
title = { Kronecker Attention Networks } ,
ISBN = { 9781450379984 } ,
url = { http://dx.doi.org/10.1145/3394486.3403065 } ,
DOI = { 10.1145/3394486.3403065 } ,
journal = { Proceedings of the 26th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining } ,
publisher = { ACM } ,
author = { Gao, Hongyang and Wang, Zhengyang and Ji, Shuiwang } ,
year = { 2020 } ,
month = { Jul }
} @article { Si2021.05.10.443415 ,
author = { Si, Yunda and Yan, Chengfei } ,
title = { Improved protein contact prediction using dimensional hybrid residual networks and singularity enhanced loss function } ,
elocation-id = { 2021.05.10.443415 } ,
year = { 2021 } ,
doi = { 10.1101/2021.05.10.443415 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415.full.pdf } ,
journal = { bioRxiv }
} @article { Costa2021.06.02.446809 ,
author = { Costa, Allan and Ponnapati, Manvitha and Jacobson, Joseph M. and Chatterjee, Pranam } ,
title = { Distillation of MSA Embeddings to Folded Protein Structures with Graph Transformers } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.02.446809 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809.full.pdf } ,
journal = { bioRxiv }
} @article { Baek2021.06.14.448402 ,
author = { Baek, Minkyung and DiMaio, Frank and Anishchenko, Ivan and Dauparas, Justas and Ovchinnikov, Sergey and Lee, Gyu Rie and Wang, Jue and Cong, Qian and Kinch, Lisa N. and Schaeffer, R. Dustin and Mill{'a}n, Claudia and Park, Hahnbeom and Adams, Carson and Glassman, Caleb R. and DeGiovanni, Andy and Pereira, Jose H. and Rodrigues, Andria V. and van Dijk, Alberdina A. and Ebrecht, Ana C. and Opperman, Diederik J. and Sagmeister, Theo and Buhlheller, Christoph and Pavkov-Keller, Tea and Rathinaswamy, Manoj K and Dalwadi, Udit and Yip, Calvin K and Burke, John E and Garcia, K. Christopher and Grishin, Nick V. and Adams, Paul D. and Read, Randy J. and Baker, David } ,
title = { Accurate prediction of protein structures and interactions using a 3-track network } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.14.448402 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402.full.pdf } ,
journal = { bioRxiv }
}