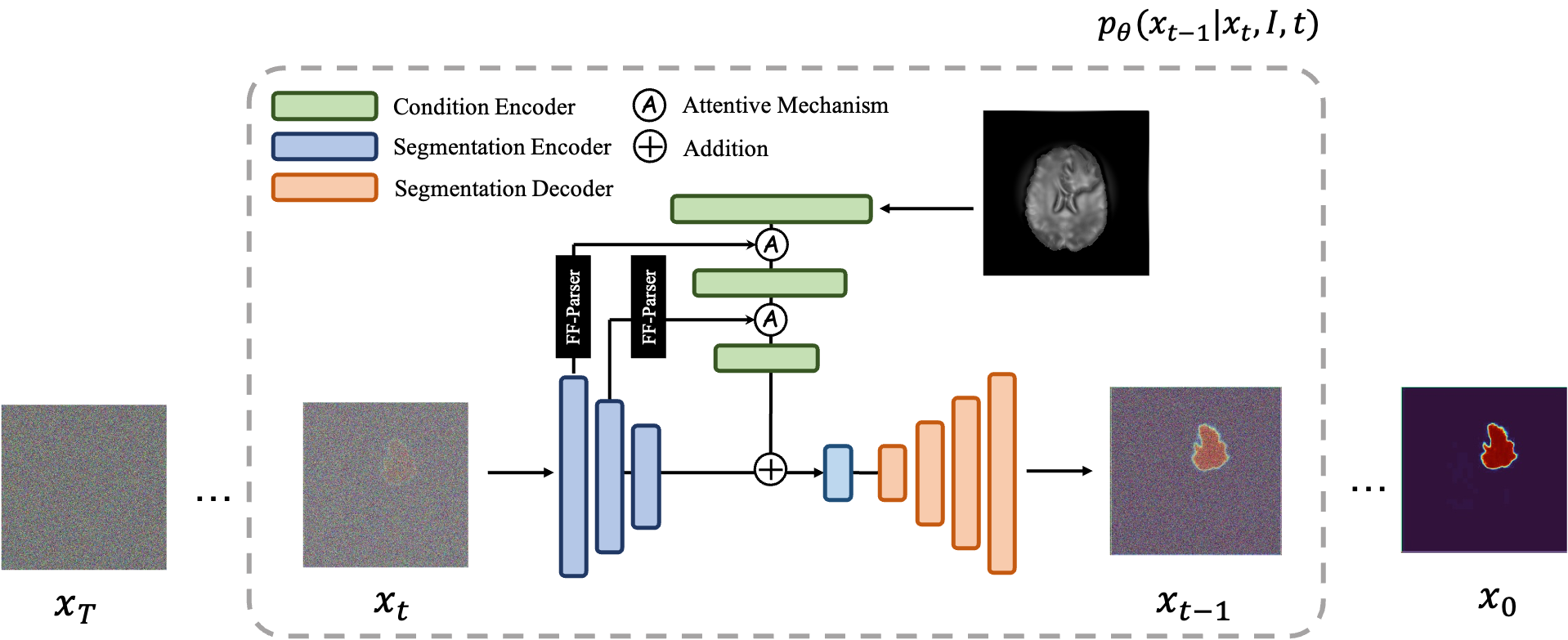
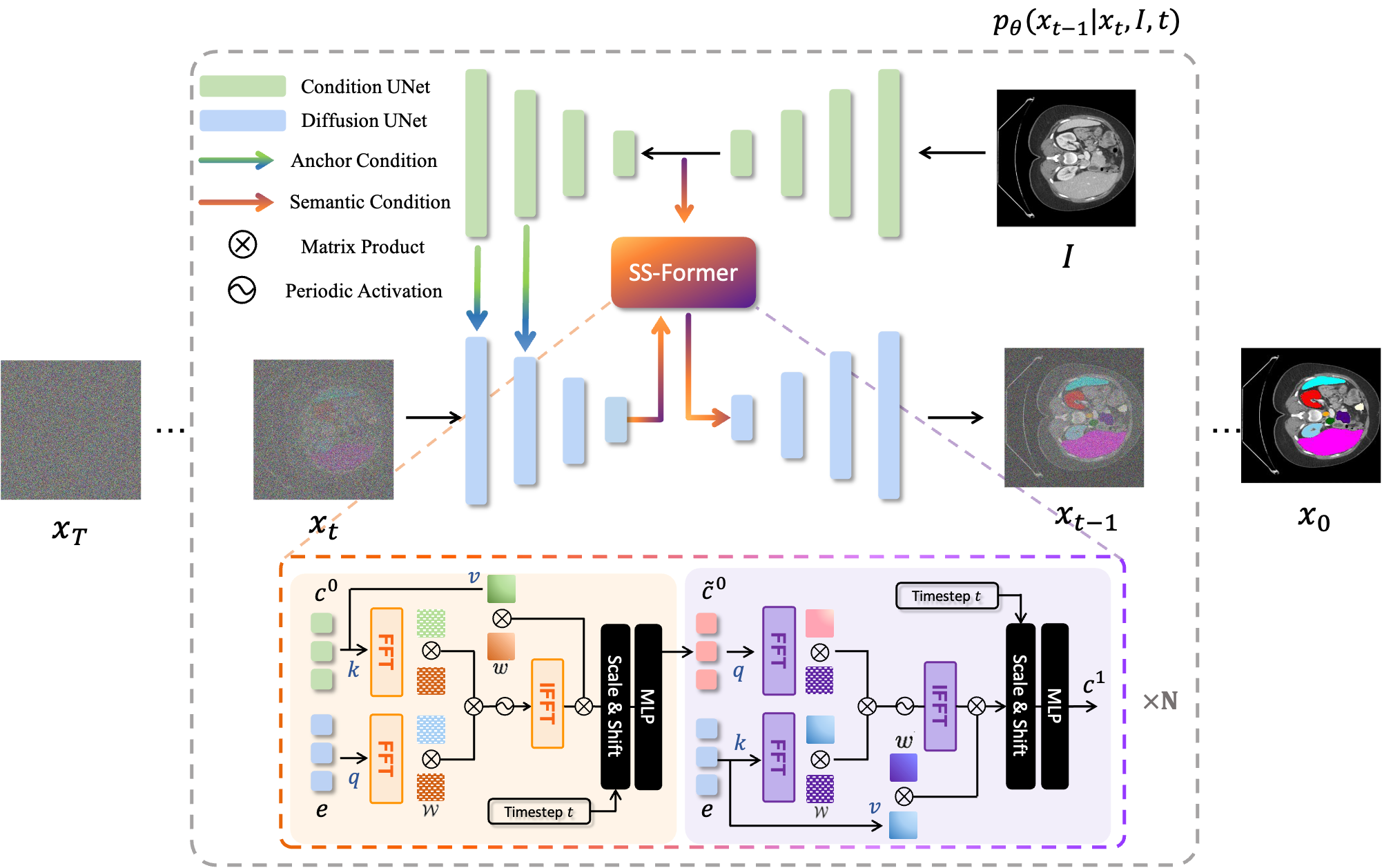
MedSegDiff هو إطار عمل قائم على نموذج الانتشار الاحتمالي (DPM) لتجزئة الصور الطبية. تم تفصيل الخوارزمية في ورقتنا MedSegDiff: تجزئة الصور الطبية باستخدام نموذج الانتشار الاحتمالي وMedSegDiff-V2: تجزئة الصور الطبية القائمة على الانتشار باستخدام المحول.
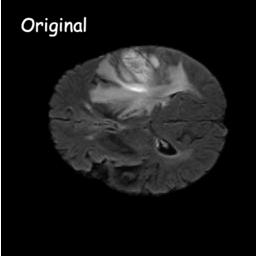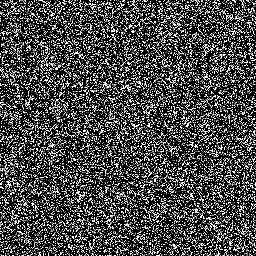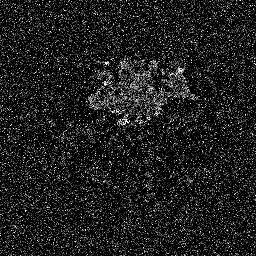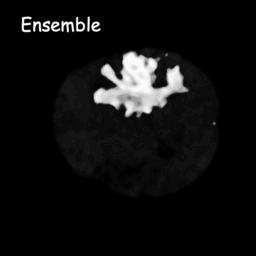 تعمل نماذج الانتشار عن طريق تدمير بيانات التدريب من خلال الإضافة المتعاقبة للضوضاء الغوسية، ثم تعلم كيفية استعادة البيانات عن طريق عكس عملية الضوضاء هذه. بعد التدريب، يمكننا استخدام نموذج الانتشار لتوليد البيانات ببساطة عن طريق تمرير عينات عشوائية من الضوضاء من خلال عملية تقليل الضوضاء المستفادة. في هذا المشروع، قمنا بتوسيع هذه الفكرة لتشمل تجزئة الصور الطبية. نحن نستخدم الصورة الأصلية كشرط وننشئ خرائط تجزئة متعددة من الضوضاء العشوائية، ثم نقوم بتجميعها للحصول على النتيجة النهائية. يلتقط هذا النهج عدم اليقين في الصور الطبية ويتفوق على الأساليب السابقة في العديد من المعايير.
تعمل نماذج الانتشار عن طريق تدمير بيانات التدريب من خلال الإضافة المتعاقبة للضوضاء الغوسية، ثم تعلم كيفية استعادة البيانات عن طريق عكس عملية الضوضاء هذه. بعد التدريب، يمكننا استخدام نموذج الانتشار لتوليد البيانات ببساطة عن طريق تمرير عينات عشوائية من الضوضاء من خلال عملية تقليل الضوضاء المستفادة. في هذا المشروع، قمنا بتوسيع هذه الفكرة لتشمل تجزئة الصور الطبية. نحن نستخدم الصورة الأصلية كشرط وننشئ خرائط تجزئة متعددة من الضوضاء العشوائية، ثم نقوم بتجميعها للحصول على النتيجة النهائية. يلتقط هذا النهج عدم اليقين في الصور الطبية ويتفوق على الأساليب السابقة في العديد من المعايير.
 |  |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
للتدريب، قم بتشغيل: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
لأخذ العينات، قم بتشغيل: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
للتقييم، قم بتشغيل python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
افتراضيًا، سيتم حفظ العينات في ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
للتدريب، قم بتشغيل: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
لأخذ العينات، قم بتشغيل: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
من السهل تشغيل MedSegDiff على مجموعات البيانات الأخرى. ما عليك سوى كتابة ملف محمل بيانات آخر يتبع ./guided_diffusion/isicloader.py أو ./guided_diffusion/bratsloader.py . مرحبا بكم في القضايا المفتوحة إذا واجهت أي مشكلة. سيكون موضع تقدير إذا كان بإمكانك المساهمة بملحقات مجموعة البيانات الخاصة بك. على عكس الصور الطبيعية، تختلف الصور الطبية كثيرًا باختلاف المهام. إن التوسع في تعميم أسلوب ما يتطلب جهود الجميع.
لتدريب نموذج جيد، أي MedSegDiff-B في الورقة، قم بتعيين المعلمات الفائقة للنموذج على النحو التالي:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
معلمات الانتشار المفرطة على النحو التالي:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
لتسريع أخذ العينات:
--diffusion_steps 50 --dpm_solver True
تعمل على وحدات معالجة الرسومات متعددة:
--multi-gpu 0,1,2 (for example)
تدريب المعلمات الفائقة على النحو التالي:
--lr 5e-5 --batch_size 8
وقم بتعيين --num_ensemble 5 في أخذ العينات.
سيتم تقارب تشغيل حوالي 100000 خطوة في التدريب على معظم مجموعات البيانات. لاحظ أنه على الرغم من أن الخسارة لن تنخفض في معظم الخطوات اللاحقة، إلا أن جودة النتائج لا تزال تتحسن. تتم ملاحظة مثل هذه العملية أيضًا في تطبيقات DPM الأخرى، مثل إنشاء الصور. آمل أن يخبرني شخص ذكي لماذا؟.
سأقوم قريبًا بنشر أدائه ضمن حجم دفعة أصغر (مناسب للتشغيل على وحدة معالجة الرسومات بسعة 24 جيجابايت) لحاجة المقارنة؟.
الإعداد لإطلاق العنان لكل إمكاناته هو (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
ثم قم بتدريبها باستخدام حجم الدفعة --batch_size 64 وأخذ عينة منها برقم المجموعة --num_ensemble 25 .
مرحبا بكم في المساهمة في MedSegDiff. أي تقنية يمكنها تحسين الأداء أو تسريع الخوارزمية موضع تقدير. أنا أكتب MedSegDiff V2، بهدف النشر في مجلات Nature/CVPR. يسعدني أن أدرج المساهمين كمؤلفين مشاركين لي؟.
تم نسخ الكود كثيرًا من openai/improved-diffusion، WuJunde/ MrPrism، WuJunde/ DiagnosisFirst، LuChengTHU/dpm-solver، JuliaWolleb/Diffusion-based-Segmentation، hojonathanho/diffusion، Guided-diffusion، bigmb/Unet-Segmentation-Pytorch-Nest -من الوحدات، nnUnet، lucidrains/vit-pytorch
يرجى الاستشهاد
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu