كتب فاروس في الأصل ويلي بروهن كأطراف البكالوريوس التي يشرف عليها ماريو ستانك. هذا المستودع عبارة عن نسخة من https://github.com/willybruhn/varus التي تم إجراؤها في نوفمبر 2018 ويحتوي على العديد من أسماك الأخطاء ، وميزة قاعدة بيانات intron الإضافية وتمديد لاستخدام برنامج المحاذاة البديل Hisat al.
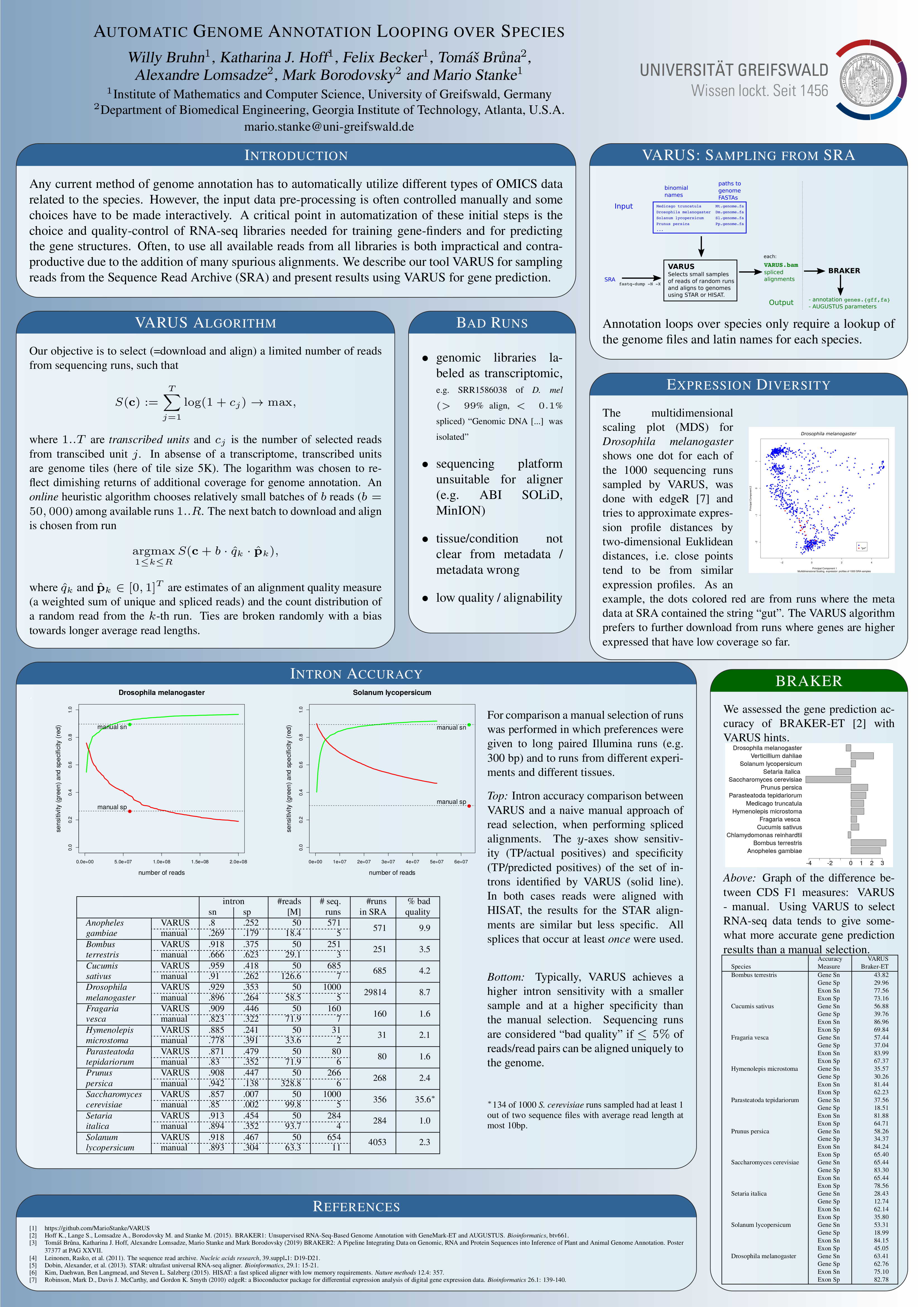
يقوم Varus بأتمتة اختيار وتنزيل عدد محدود من القراءات RNA-Seq من AT AT AT Sequence Archive Archive (SRA) التي تستهدف تغطية عالية بما فيه الكفاية للعديد من الجينات لغرض تدريب الجينات وشرح الجينوم. كل تكرار للخوارزمية عبر الإنترنت
استدعاء الأمر التالي من سطر الأوامر من أجل استنساخ المستودع:
git clone https://github.com/MarioStanke/VARUS.gitالفاروس يعتمد على
sudo apt-get install bamtools libbamtools-devتجميع الفاروس يدويًا مع
cd Implementation
make
افتراضيًا ، يقوم NCBI Tool fastq-dump بإنشاء ملفات مؤقتة ضمن ~/ncbi بنفس حجم ملف التشغيل الذي يتم تنزيل البيانات منه ، حتى لو تم تنزيل جزء صغير منه فقط. تعطيل سلوك التخزين المؤقت هذا الذي يتطلب مساحة كبيرة جدًا على محرك الأقراص الثابتة لمعظم المستخدمين
mkdir -p ~/.ncbi
echo '/repository/user/cache-disabled = "true"' >> ~/.ncbi/user-settings.mkfg
قم بتغيير example الدليل واتبع التعليمات في المثال/ReadMe.
انسخ ملف VARUSparameters.txt من المجلد إلى دليل العمل الخاص بك وضبطه إذا لزم الأمر:
أهم المعلمات:
-يحدد BatchSize عدد القراءات التي يجب تنزيلها في كل تكرار (على سبيل المثال 50000 أو 200000)
-يحدد MaxBatches عدد الدُفعات التي يجب تنزيلها على الأكثر
الإخراج النهائي هو ملف محاذاة مرسوم مرتبة (جميع الدُفعات معًا) يسمى varus.bam .
يرجى الاستشهاد: varus: أخذ عينات من الحمض النووي الريبي التكميلي من أرشيف قراءة التسلسل. 2019 ؛ BMC Bioinformatics ، 20: 558
ابحث عن أطروحة البكالوريوس لـ Willy Bruhn المقابلة لـ Varus في /docs /assis.