Die Implementierung von DiffAbXL wurde im Artikel „Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs“ einem Benchmarking unterzogen.
| Rang | Modelle | Absci HER2 | Natur | AZ Target-2 | Ave. ? | ||
|---|---|---|---|---|---|---|---|
| Null Schuss | SPR-Steuerung | HEL | HER2 | ||||
| 1 | DiffAbXL-A-DN | 0,43 | 0,22 | 0,62 | 0,37 | 0,41 | 0,41 |
| 2 | DiffAbXL-A-SG | 0,46 | 0,22 | 0,64 | -0,38 | 0,43 | 0,274 |
| 3 | DiffAbXL-H3-DN | 0,49 | 0 | 0,52 | -0,08 | 0,37 | 0,26 |
| 4 | IgBlend (nur Struktur) | 0,40 | 0,21 | 0,54 | -0,30 | 0,31 | 0,232 |
| 5 | Antifold | 0,43 | 0,22 | 0,4 | -0,47 | 0,38 | 0,192 |
| 6 | DiffAbXL-H3-SG | 0,48 | 0 | 0,4 | -0,41 | 0,29 | 0,152 |
| 7 | ESM | 0,29 | 0 | 0 | 0,18 | 0,27 | 0,148 |
| 8 | DiffAb | 0,34 | 0,21 | 0 | -0,14 | 0,22 | 0,126 |
| 9 | AbLang2 | 0,3 | 0 | 0 | -0,07 | 0,36 | 0,118 |
| 10 | IgBlend (nur seq.) | 0,27 | 0 | 0 | -0,1 | 0,36 | 0,106 |
| 11 | AbLang | 0,3 | 0 | 0 | -0,13 | 0,35 | 0,104 |
| 12 | dyMEAN | 0,37 | 0,15 | 0 | 0 | 0 | 0,104 |
| 13 | AbX | 0,28 | 0,19 | 0 | 0 | 0 | 0,094 |
| 14 | AntiBERTy | 0,26 | 0 | 0 | -0,17 | 0,35 | 0,088 |
| 15 | BEDEUTEN | 0,36 | 0 | 0 | 0,02 | 0 | 0,076 |
| 16 | ESM-IF | 0 | -0,27 | 0 | -0,53 | 0,42 | -0,076 |
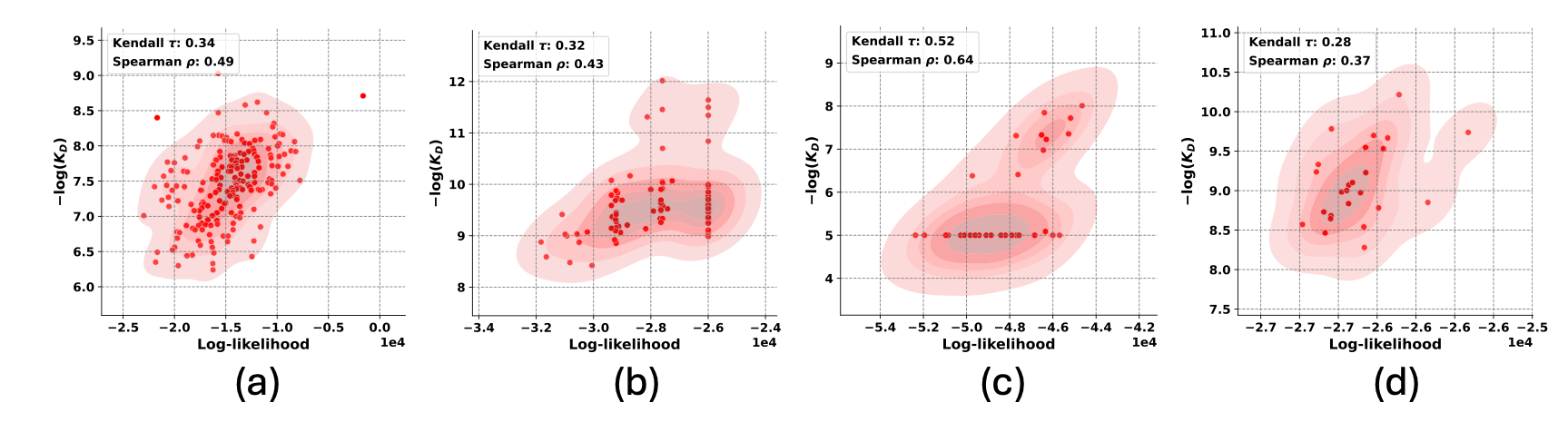
Abbildung 1: Ergebnisse für DiffAbXL: a) DiffAbXL-H3-DN für Absci Zero-Shot HER2-Daten b) DiffAbXL-A-SG für AZ Target-2, c) DiffAbXL-A-SG für Nature HEL, d) DiffAbXL- A-DN für Nature HER2.
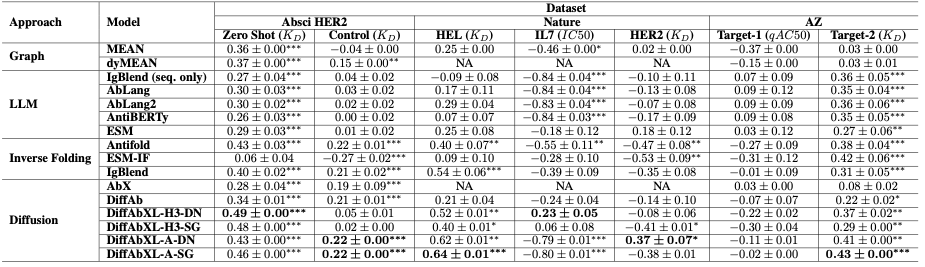
Tabelle 1: Zusammenfassung der Ergebnisse der Spearman-Korrelation. Abkürzungen: DN: De-Novo-Modus, SG: Strukturführungsmodus, NA: Epitop oder komplexe Struktur erforderlich, aber nicht verfügbar. *, **, *** geben p-Werte unter 0,05, 0,01 bzw. 1e-4 an.
Um uns das Benchmarking Ihres Modells zu erleichtern, empfehlen wir Ihnen, eine Schnittstelle als Python-Methode in einer Klasse zu implementieren, die wir problemlos in unsere Evaluierungspipeline integrieren können. Die Methode sollte die folgenden Eingaben akzeptieren:
Die Methode sollte ein Wörterbuch zurückgeben, das Folgendes enthält:
Hier ist eine grundlegende Vorlage in Python zum Implementieren dieser Schnittstelle:
def benchmark ( self , sequences , structure = None , mask = None , ** kwargs ):
"""
Benchmark the model on provided antibody sequences and structures.
Parameters:
sequences (list of str): List of antibody sequences.
structure (optional): Path to a PDB file. Currently, only one PDB file is provided per target dataset.
The PDB file may contain either just the antibody or an antibody-antigen complex,
depending on the dataset.
mask (optional): Binary list or array indicating the regions of interest in the sequences for metric calculations.
kwargs (optional): Additional parameters required by the model.
Returns:
dict: A dictionary containing log-likelihood scores and other relevant metrics.
"""
passBitte stellen Sie sicher, dass Ihr Modell die Log-Likelihood-Scores in einem Format ausgibt, das wir direkt zum Benchmarking von Antikörpersequenzdesigns verwenden können. Dadurch können wir die Leistung Ihres Modells in unseren Datensätzen effizient vergleichen.
Es gibt eine Konfigurationsdatei: sabdab.yaml, mit der beliebige Parameter geändert werden können. Sie können das Modell trainieren, indem Sie Folgendes verwenden:
python train.py # For training.
- train.py
- src
|-model.py
- Konfig
|-sabdab.yaml
- Dienstprogramme
|-load_data.py
|-arguments.py
|-model_utils.py
|-loss_functions.py
...
- Daten
|-her2
...
Gewichtung und Verzerrungen können zum Verfolgen von Experimenten verwendet werden. Es ist standardmäßig deaktiviert, kann aber durch Ändern der Option in der Konfigurationsdatei in ./config/sabdab.yaml aktiviert werden
@article {Ucar2024.10.07.617023,
author = {Ucar, Talip and Malherbe, Cedric and Gonzalez Hernandez, Ferran},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
elocation-id = {2024.10.07.617023},
year = {2024},
doi = {10.1101/2024.10.07.617023},
publisher = {Cold Spring Harbor Laboratory},
URL = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023},
eprint = {https://www.biorxiv.org/content/early/2024/10/24/2024.10.07.617023.full.pdf},
journal = {bioRxiv}
}
Wenn Sie DiffAbXL in Ihren eigenen Studien und Ihrer Arbeit verwenden, zitieren Sie es bitte wie folgt:
@Misc{talip_ucar_2024_DiffAbXL,
author = {Talip Ucar},
title = {Exploring Log-Likelihood Scores for Ranking Antibody Sequence Designs},
URL = {https://github.com/AstraZeneca/DiffAbXL},
month = {October},
year = {since 2024}
}