Schnelles Batch-Datenladen von BigWig-Dateien mit epigentischen Trackdaten und entsprechenden Sequenzen, unterstützt durch GPU für Deep-Learning-Anwendungen.
Bigwig-Loader hängt hauptsächlich von der RapidSai-Kvikio-Bibliothek und Cupy ab, die beide am besten mit Conda/Mamba installiert werden. Bigwig-Loader kann jetzt auch mit Conda/Mamba installiert werden. So erstellen Sie eine neue Umgebung mit installiertem Bigwig-Loader:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderOder fügen Sie dies Ihrer Datei „environment.yml“ hinzu:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderund aktualisieren:
mamba env update -f environment.ymlBigwig-Loader kann auch mit Pip in einer Umgebung installiert werden, in der die Rapidsai-Kvikio-Bibliothek und Cupy bereits installiert sind:
pip install bigwig-loaderWir haben das BigWigDataset in einen iterierbaren PyTorch-Datensatz verpackt, den Sie direkt verwenden können:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Ein Framework-unabhängiges Dataset-Objekt kann aus bigwig_loader.dataset importiert werden. Dieses Datensatzobjekt gibt Cupy-Tensoren zurück. Cupy-Tensoren folgen der Cuda-Array-Schnittstelle und können ohne Kopie in JAX- oder Tensorflow-Tensoren umgewandelt werden.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Weitere Beispiele finden Sie im Beispielverzeichnis.
Diese Bibliothek ist zum Laden von Datenstapeln mit derselben Dimensionalität gedacht, was einige Annahmen zulässt, die den Ladevorgang beschleunigen können. Wie aus der folgenden Darstellung hervorgeht, ist pyBigWig beim Laden einer kleinen Datenmenge sehr schnell, nutzt jedoch nicht den Batch-Charakter des Datenladens für maschinelles Lernen.
Im folgenden Benchmark haben wir auch PyTorch-Datenlader (mit set_start_method('spawn')) mit pyBigWig erstellt, um sie mit dem realistischen Szenario zu vergleichen, in dem mehrere CPUs pro GPU verwendet würden. Wir sehen, dass der Durchsatz des CPU-Datenladers nicht linear mit der Anzahl der CPUs steigt und es daher schwierig wird, den erforderlichen Durchsatz zu erreichen, um die GPU, die das neuronale Netzwerk trainiert, während der Lernschritte gesättigt zu halten.
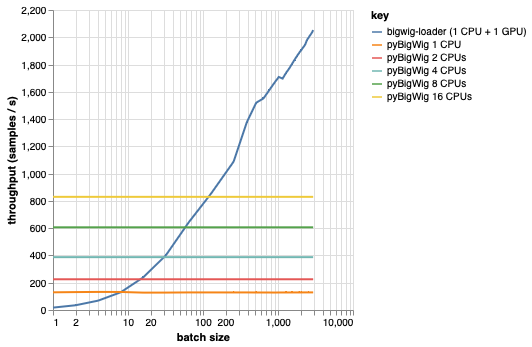
Dies ist das Problem, das Bigwig-Loader löst. Dies ist ein Beispiel für die Verwendung von bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml In dieser Umgebung sollten Sie in der Lage sein, pytest -v auszuführen und zu sehen, dass die Tests erfolgreich sind. HINWEIS: Sie benötigen eine GPU, um Bigwig-Loader verwenden zu können!
Dieser Abschnitt führt Sie durch die Schritte, die zum Hinzufügen neuer Funktionen erforderlich sind. Wenn etwas unklar ist, öffnen Sie bitte ein Problem.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install aus, um die pre-commit-Hooks zu installierenTests befinden sich im Tests-Verzeichnis. Einer der wichtigsten Tests ist test_against_pybigwig, der sicherstellt, dass ein Fehler in pyBigWIg auch im Bigwig-Loader auftritt.
pytest -vv .Wenn Github-Runner mit GPUs verfügbar werden, möchten wir diese Tests auch im CI durchführen. Aber im Moment können Sie sie lokal ausführen.
Wenn Sie diese Bibliothek verwenden, sollten Sie Folgendes zitieren:
Retel, Joren Sebastian, Andreas Poehlmann, Josh Chiou, Andreas Steffen und Djork-Arné Clevert. „Ein schneller Datenlader für maschinelles Lernen für epigenetische Spuren aus BigWig-Dateien.“ Bioinformatik 40, Nr. 1 (1. Januar 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}