
(c) 2017 Timothy Becker & Wan-Ping Lee
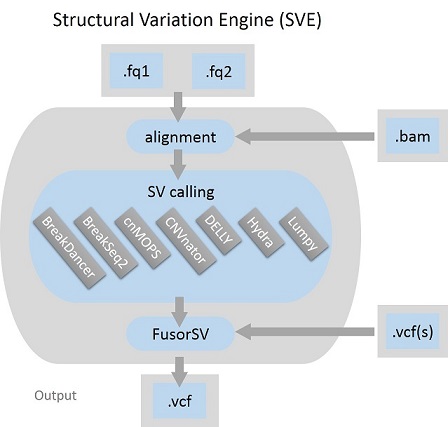
SVE ist eine auf Python-Skripten basierende Ausführungs-Engine für die Erkennung struktureller Variationen (SV). Sie kann für alle Ebenen von Dateneingaben, rohen FASTQs, ausgerichteten BAMs oder Variantenaufrufformaten (VCFs) verwendet werden und generiert als Ausgabe ein einheitliches VCF. SVE besteht standardmäßig aus Ausrichtung, Neuausrichtung und dem Ensemble modernster SV-Aufrufalgorithmen. Sie sind BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra und LUMPY. Außerdem ist FusorSV eingebettet, ein Data-Mining-Ansatz zur Bewertung der Leistung und zum Zusammenführen von Aufrufsätzen aus einem Ensemble von SV-Aufrufalgorithmen.
Bitte legen Sie die ROOT-Umgebung fest.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeBitte überprüfen Sie die Python2.7-Header-Dateien und ändern Sie „CFLAGS_FUSOR_SV“ im Makefile. Die Header-Dateien befinden sich möglicherweise auf „/usr/include/python2.7“ und verwenden stattdessen „CFLAGS_FUSOR_SV=-I /usr/include/python2.7“.
make FusorSV
Oder Sie können FusorSV über setup.py installieren
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
Alternativ werden Dockerfile und Docker-Image bereitgestellt. Bitte beachten Sie, dass je nach Computereinstellung sudo für Docker-Nutzungen erforderlich sein kann.
cd SVE
docker build .
Ziehen Sie das Docker-Image aus dem Repository.
docker pull wanpinglee/sve
SVE basiert auf /tools/SVE. Überprüfen Sie die Hilfe von
/tools/SVE/bin/sve
Kurze Lesevorgänge in FASTQ werden dem angegebenen FASTA zugeordnet und ein sortiertes BAM wird generiert.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
Wenn die Lesevorgänge im BAM-Format vorliegen, ordnet die Neuausrichtung die Lesevorgänge anhand von FASTA neu zu und generiert ein sortiertes BAM. Wir verwenden SpeedSeq, um die Neuausrichtung durchzuführen.
bin/sve realign -r <FASTA> <BAM>
Es gibt sieben SV-Aufrufalgorithmen, die für SV-Aufrufe verwendet werden können. VCF wird generiert.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
Nach dem Aufruf kann jedes Sample über mehrere VCFs verfügen, abhängig von der Anzahl der verwendeten Aufrufer. Bitte sammeln Sie VCFs einer Probe in einem Ordner.
Die VCFs sollten SVE-IDs verwenden, um die Anrufer anzuzeigen.
| SVE-ID | Anrufer |
|---|---|
| 4 | BreakDancer (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnator (v0.3.3) |
| 11 | DELLY (v2) |
| 14* | GenomeSTRiP |
| 17 | Hydra |
| 18 | KLUMPIG |
| 35 | BreakSeq (v2.2) |
| 0 | Wahrheit (optional) |
Hinweis*: Aufgrund von Lizenzproblemen ist GenomeSTRiP nicht in SVE eingebettet. Das FusorSV-Standardmodell ist jedoch in der Lage, GenomeSTRiP VCF zu verarbeiten.
Beispielhafte VCF-Eingabedateien können wie folgt organisiert werden. Bitte beachten Sie, dass vcfFiles das Argument für -i für FusorSV ist.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Laut S0.vcf wird ein neues Modell generiert und VCFs werden durch das neue Modell zusammengeführt.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Das Projekt ist unter der GPL-3.0-Lizenz lizenziert. Weitere Informationen finden Sie unter LIZENZ.