Die gesamten gut verarbeiteten und DL-fähigen Daten von 109 Probanden aus EEGMMIDB werden hochgeladen!
Dieses Tutorial enthält umsetzbare Python- und Jupyter-Notebook-Codes und Benchmark-Datensätze, um zu lernen, wie man Gehirnsignale basierend auf Deep-Learning-Modellen erkennt. Dieses Tutorial verbindet unsere Umfrage zu DL-basierten nichtinvasiven Gehirnsignalen und unser Buch über DL-basiertes BCI: Repräsentationen, Algorithmen und Anwendungen.
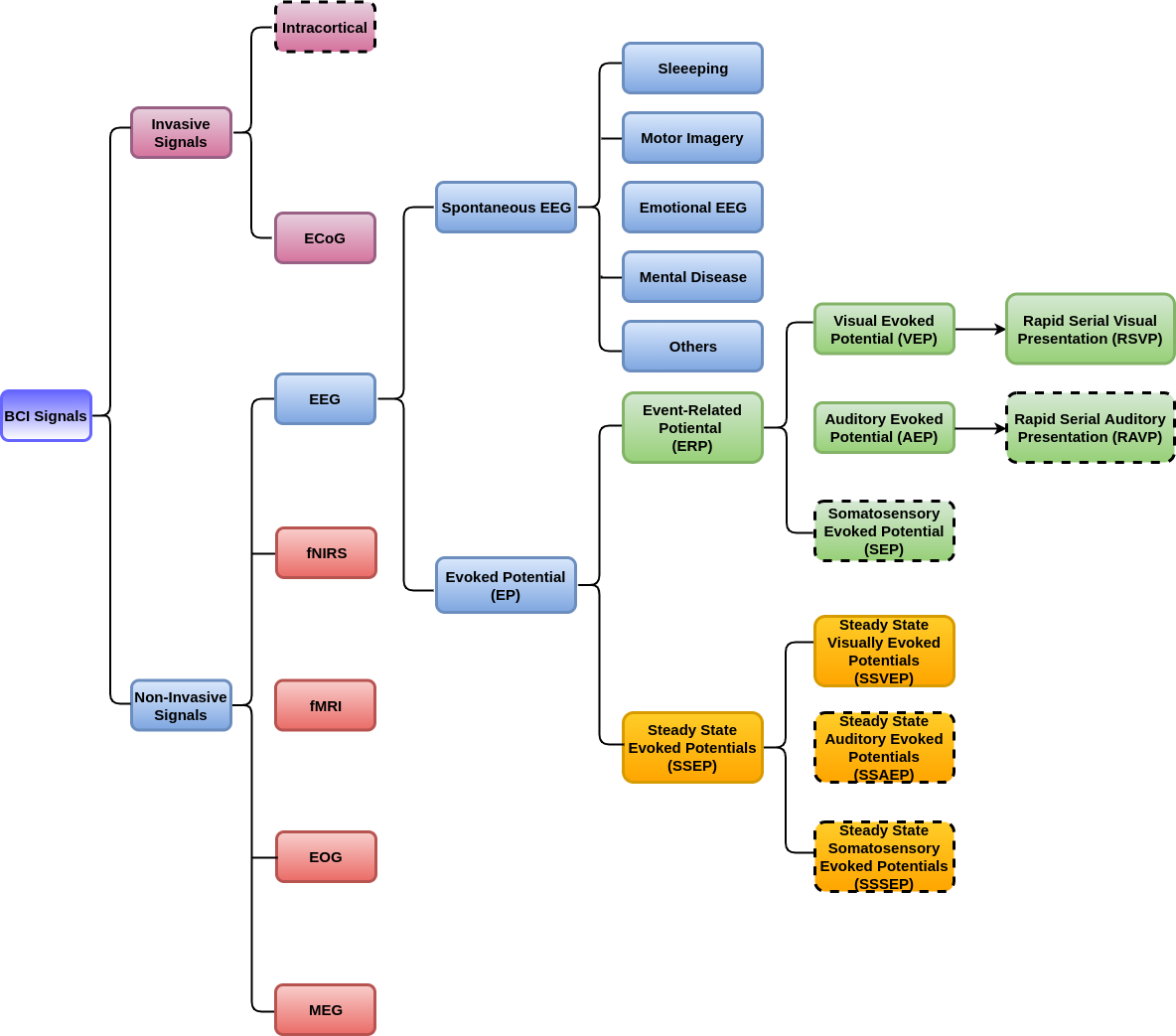
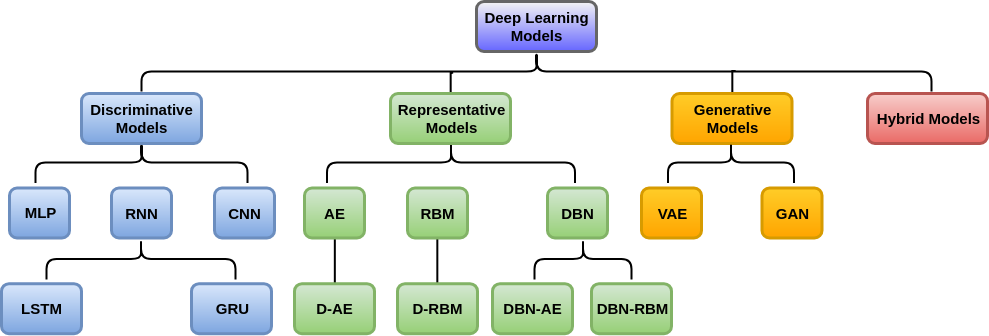
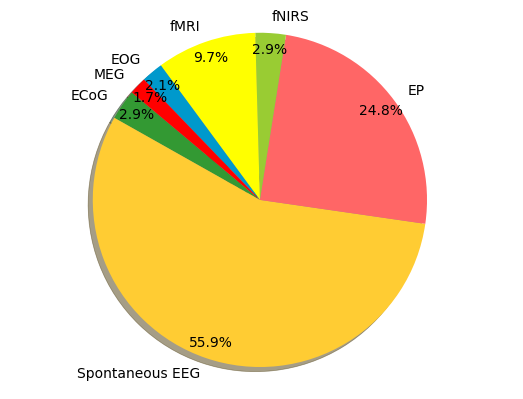
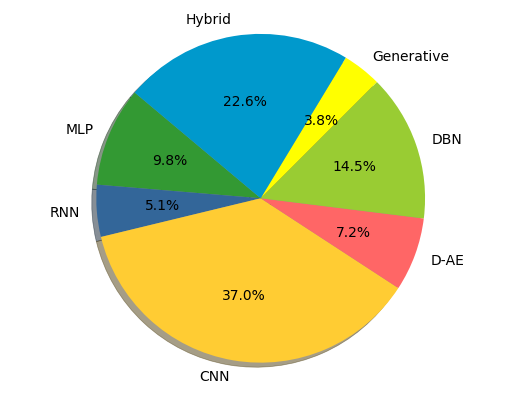
| Verteilung auf Signale | Verteilung auf DL-Modelle |
|---|---|
 |  |
Besonderes Augenmerk wurde auf die neuesten Studien zum Deep Learning für die EEG-basierte BCI-Forschung im Hinblick auf Algorithmen gelegt. Insbesondere stellen wir eine Reihe fortschrittlicher Deep-Learning-Algorithmen und -Frameworks vor, die auf mehrere wichtige Probleme bei BCI abzielen, darunter robustes Lernen der Gehirnsignaldarstellung, szenarioübergreifende Klassifizierung und halbüberwachte Klassifizierung.
Darüber hinaus werden mehrere neuartige Prototypen von Deep-Learning-basierten BCI-Systemen vorgeschlagen, die Aufschluss über reale Anwendungen wie Authentifizierung, visuelle Rekonstruktion, Sprachinterpretation und Diagnose neurologischer Störungen geben. Solche Anwendungen können sowohl gesunden Menschen als auch Menschen mit Behinderungen im wirklichen Leben enorme Vorteile bringen.
Die Erfassung von Gehirnsignalen ist sowohl finanziell als auch zeitlich kostspielig. Wir untersuchen ausführlich die Benchmark-Datensätze für die Regensignalforschung und stellen 31 öffentliche Datensätze mit Download-Links zur Verfügung, die die meisten Gehirnsignaltypen abdecken.
| Gehirnsignale | Datensatz | #-Thema | #-Klassen | Abtastrate (Hz) | #-Kanäle | Download-Link |
|---|---|---|---|---|---|---|
| FM EcoG | BCI-C IV, Datensatz IV | 3 | 5 | 1000 | 48 -- 64 | Link |
| MI EcoG | BCI-C III Datensatz I | 1 | 2 | 1000 | 64 | Link |
| Schlaf-EEG | Schlaf-EDF-Telemetrie | 22 | 6 | 100 | 2 EEG, 1 EOG, 1 EMG | Link |
| Schlaf-EEG | Schlaf-EDF: Kassette | 78 | 6 | 100, 1 | 2 EEG (100 Hz), 1 EOG (100 Hz), 1 EMG (1 Hz) | Link |
| Schlaf-EEG | MASSE-1 | 53 | 5 | 256 | 17/19 EEG, 2 EOG, 5 EMG | Link |
| Schlaf-EEG | MASSE-2 | 19 | 6 | 256 | 19 EEG, 4 EOG, 1EMG | Link |
| Schlaf-EEG | MASSE-3 | 62 | 5 | 256 | 20 EEG, 2 EOG, 3 EMG | Link |
| Schlaf-EEG | MASSE-4 | 40 | 6 | 256 | 4 EEG, 4 EOG, 1 EMG | Link |
| Schlaf-EEG | MASSE-5 | 26 | 6 | 256 | 20 EEG, 2 EOG, 3 EMG | Link |
| Schlaf-EEG | SHHS | 5804 | N / A | 125, 50 | 2 EEG (125 Hz), 1EOG (50 Hz), 1 EMG (125 Hz) | Link |
| Anfalls-EEG | CHB-MIT | 22 | 2 | 256 | 18 | Link |
| Anfalls-EEG | TUH | 315 | 2 | 200 | 19 | Link |
| MI EEG | EEGMMI | 109 | 4 | 160 | 64 | Link |
| MI EEG | BCI-C II, Datensatz III | 1 | 2 | 128 | 3 | Link |
| MI EEG | BCI-C III, Datensatz III a | 3 | 4 | 250 | 60 | Link |
| MI EEG | BCI-C III, Datensatz III b | 3 | 2 | 125 | 2 | Link |
| MI EEG | BCI-C III, Datensatz IV a | 5 | 2 | 1000 | 118 | Link |
| MI EEG | BCI-C III, Datensatz IV b | 1 | 2 | 1001 | 119 | Link |
| MI EEG | BCI-C III, Datensatz IV c | 1 | 2 | 1002 | 120 | Link |
| MI EEG | BCI-C IV, Datensatz I | 7 | 2 | 1000 | 64 | Link |
| MI EEG | BCI-C IV, Datensatz II a | 9 | 4 | 250 | 22 EEG, 3 EOG | Link |
| MI EEG | BCI-C IV, Datensatz II b | 9 | 2 | 250 | 3 EEG, 3 EOG | Link |
| Emotionales EEG | AMIGOS | 40 | 4 | 128 | 14 | Link |
| Emotionales EEG | SAMEN | 15 | 3 | 200 | 62 | Link |
| Emotionales EEG | DEAP | 32 | 4 | 512 | 32 | Link |
| Andere EEG | Öffnen Sie MIIR | 10 | 12 | 512 | 64 | Link |
| VEP | BCI-C II, Datensatz II b | 1 | 36 | 240 | 64 | Link |
| VEP | BCI-C III, Datensatz II | 2 | 26 | 240 | 64 | Link |
| fMRT | ADNI | 202 | 3 | N / A | N / A | Link |
| fMRT | Gören | 65 | 4 | N / A | N / A | Link |
| MEG | BCI-C IV, Datensatz III | 2 | 4 | 400 | 10 | Link |
Um den Lesern einen schnellen Zugriff auf den Datensatz zu ermöglichen und ihn auszuprobieren, stellen wir den gut verarbeiteten und gebrauchsfertigen Datensatz der EEG Motor Movement/Imagery Database (EEGMMIDB) zur Verfügung. Dieser Datensatz enthält 109 Probanden, während die EEG-Signale in 64 Kanälen mit einer Abtastrate von 160 Hz aufgezeichnet werden. Nach unserem Löschen und Sortieren stellt jede NPY-Datei ein Thema dar: Die Datenform jeder NPY-Datei ist [N, 65], die ersten 64 Spalten entsprechen 64 Kanalmerkmalen, die letzte Spalte gibt die Klassenbezeichnung an. Das N variiert für verschiedene Themen, aber N sollte entweder 259520 oder 255680 sein. Dies ist der inhärente Unterschied im Originaldatensatz.
In unseren Tutorial-Dateien lernen Sie die Pipeline und den Arbeitsablauf des BCI-Systems kennen, einschließlich Datenerfassung, Vorverarbeitung, Merkmalsextraktion (optional), Klassifizierung und Auswertung. Wir präsentieren notwendige Referenzen und umsetzbare Codes der typischsten Deep-Learning-Modelle (GRU, LSTM, CNN, GNN) und nutzen dabei zeitliche, räumliche und topografische Abhängigkeiten. Wir stellen auch Python-Codes zur Verfügung, die sehr praktisch sind. Um beispielsweise die EEG-Klassifizierungsleistung von CNN zu überprüfen, führen Sie den folgenden Code aus:
python 4-2_CNN.py
Für PyTorch-Anfänger empfehlen wir dringend die PyTorch-Tutorials von Morvan Zhou.
Für die im Buch vorgestellten Algorithmen und Anwendungen stellen wir die notwendigen Implementierungscodes (TensorFlow-Version) zur Verfügung:
Wenn Sie der Meinung sind, dass unsere Forschung für Ihre Forschung nützlich ist, denken Sie bitte darüber nach, unsere Umfrage oder unser Buch zu zitieren:
@article{zhang2020survey,
title={A survey on deep learning-based non-invasive brain signals: recent advances and new frontiers},
author={Zhang, Xiang and Yao, Lina and Wang, Xianzhi and Monaghan, Jessica JM and Mcalpine, David and Zhang, Yu},
journal={Journal of Neural Engineering},
year={2020},
publisher={IOP Publishing}
}
@book{zhang2021deep,
title={Deep Learning for EEG-based Brain-Computer Interface: Representations, Algorithms and Applications},
author={Zhang, Xiang and Yao, Lina},
year={2021},
publisher={World Scientific Publishing}
}
Die Tutorial-Codes wurden auf ihre Funktionsfähigkeit unter Python 3.7 getestet.
Aktuelle Versionen von Pytorch, Torch-Geometrie, Numpy und Scipy sind erforderlich. Alle erforderlichen Basispakete können mit dem folgenden Befehl installiert werden: ''' pip install -r require.txt ''' Hinweis: Für toch-geometrische und die damit verbundenen Abhängigkeiten (z. B. Cluster, Scatter, Sparse) ist möglicherweise die höhere Version erforderlich funktionieren, wurden aber noch nicht getestet.
Bitte senden Sie alle Fragen, die Sie zum Code und/oder zum Algorithmus haben, an [email protected].
Dieses Tutorial ist unter der MIT-Lizenz lizenziert.