Dieses Repository enthält Code und Daten zur Durchführung von dermoskopischen Klassifizierung von Farbvisionsmangel mit GPT-4V.
Lesen Sie das Papier
Weitere Informationen finden Sie in der detaillierten Antwort des GPT -Modellformulars Mendeley Daten
Numpy
Pil
Daltonlens
JSON
Openai
Basis64
Pandas
. ├── data # Contains subdirectories for processed data │ ├── all │ ├── all_resized │ ├── all_resized_brettel_protan_1 │ ├── all_resized_brettel_deutan_1 │ ├── all_resized_brettel_tritan_1 │ ├── bn │ ├── bn_resized │ ├── bn_resized_label │ ├── bn_resized_label_brettel_protan_1 │ ├── bn_resized_label_brettel_deutan_1 │ ├── bn_resized_label_brettel_tritan_1 │ ├── mm │ ├── mm_resized │ ├── mm_resized_label │ ├── mm_resized_label_brettel_protan_1 │ ├── mm_resized_label_brettel_deutan_1 │ ├── mm_resized_label_brettel_tritan_1 │ └── selected_images.xlsx # Image names ├── RAW # Contains raw data downloaded from ISCI Archive ├── result # Results from running API_calling will be stored here │ ├── 2_shot_brettel_protan_1 │ │ ├──rep 1 │ │ └── ... │ ├── 2_shot_brettel_deutan_1 │ └── ... ├── CVD_classification_GPT.py # Call OpenAI API for classification ├── CVD_convertor.py # Convert original image to CVD simulated image ├── data_labeling.py # Add label to the image for reference ├── data_resizing.py # Resize the data with the original aspect ratio ├── data_selection.py # Select data from RAW └── README.md
Alle Dermoskopischen Bilder werden vom ISIC -Archiv heruntergeladen.
Dieses Projekt dient nur für akademische Forschungszwecke. Der Code in diesem Repository wird unter der MIT -Lizenz veröffentlicht. Wenn Sie die bereitgestellten Daten verwenden, zitieren Sie bitte das ISIC -Archiv.
Für die CVD-Simulation haben wir das Daltonlens-Python-Paket ausgewählt. Es hat eine Vielzahl der derzeit verfügbaren Algorithmen der Farbblindheitssimulation abgedeckt. Der Blog des Autors ist auch lesenswert.
Wir haben den Brettel et al. 1997 als CVD -Simulationsmethode und setzen Sie den Schweregrad auf 1, der nach Bedarf mit dem bereitgestellten Code angepasst werden kann.
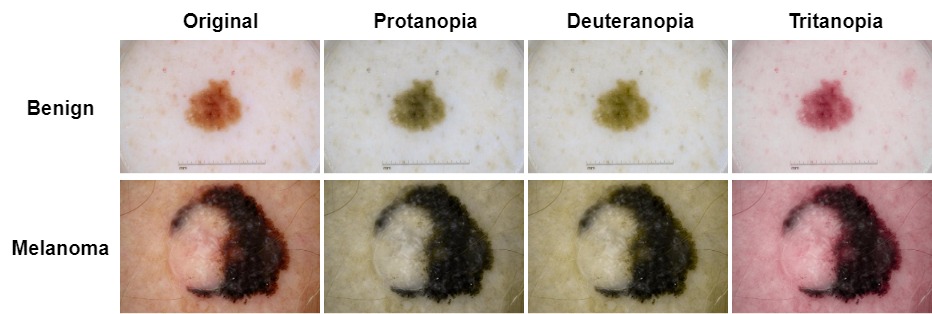
Gutartiges Beispiel: ISIC_0012656
Beispiel für Melanom: ISIC_0046725
Laden Sie die Rohdaten aus dem ISIC -Archiv herunter
Daten auswählen:
Python data_selection.py -[Optionen] ausführen
Prozessdaten:
Führen Sie Python Data_Resize.py -[Optionen] aus.
Führen Sie Python data_labeling.py -[Optionen] aus.
Daten konvertieren:
Führen Sie Python cvd_convertor.py -[Optionen] aus
API anrufen:
Führen Sie Python cvd_classification_gpt.py -[Optionen] aus.
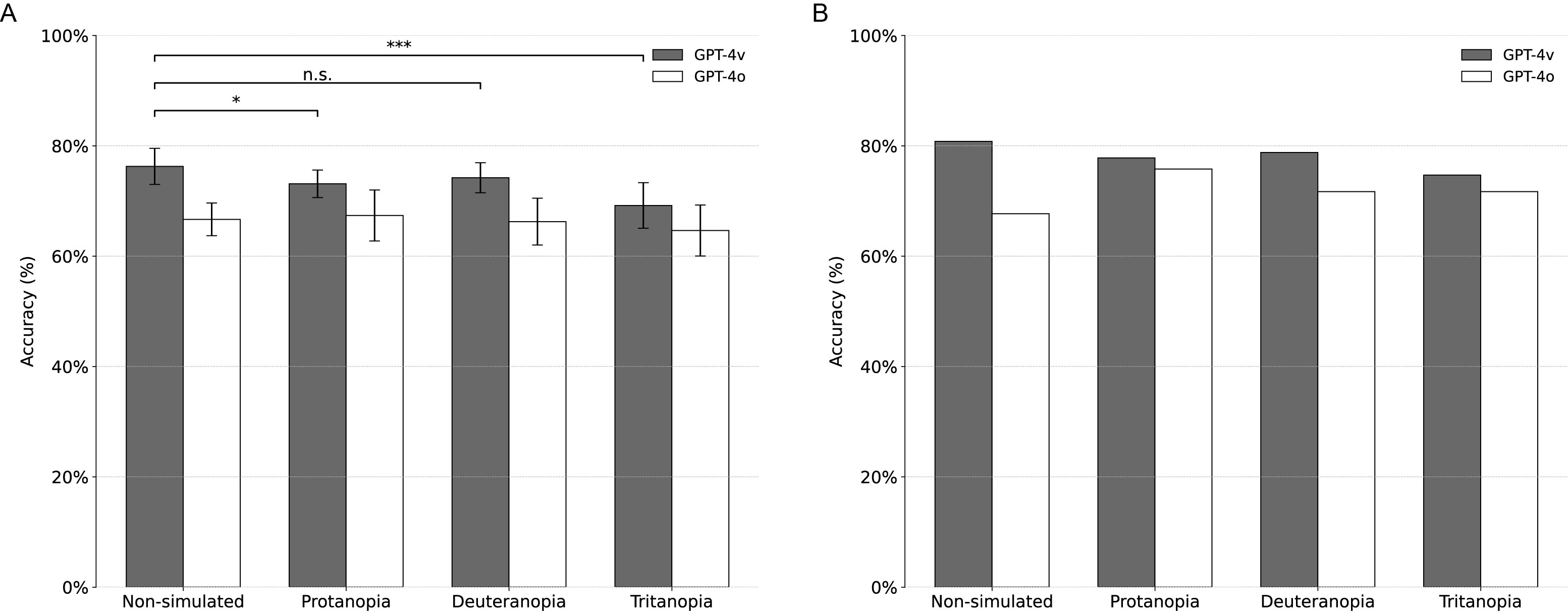
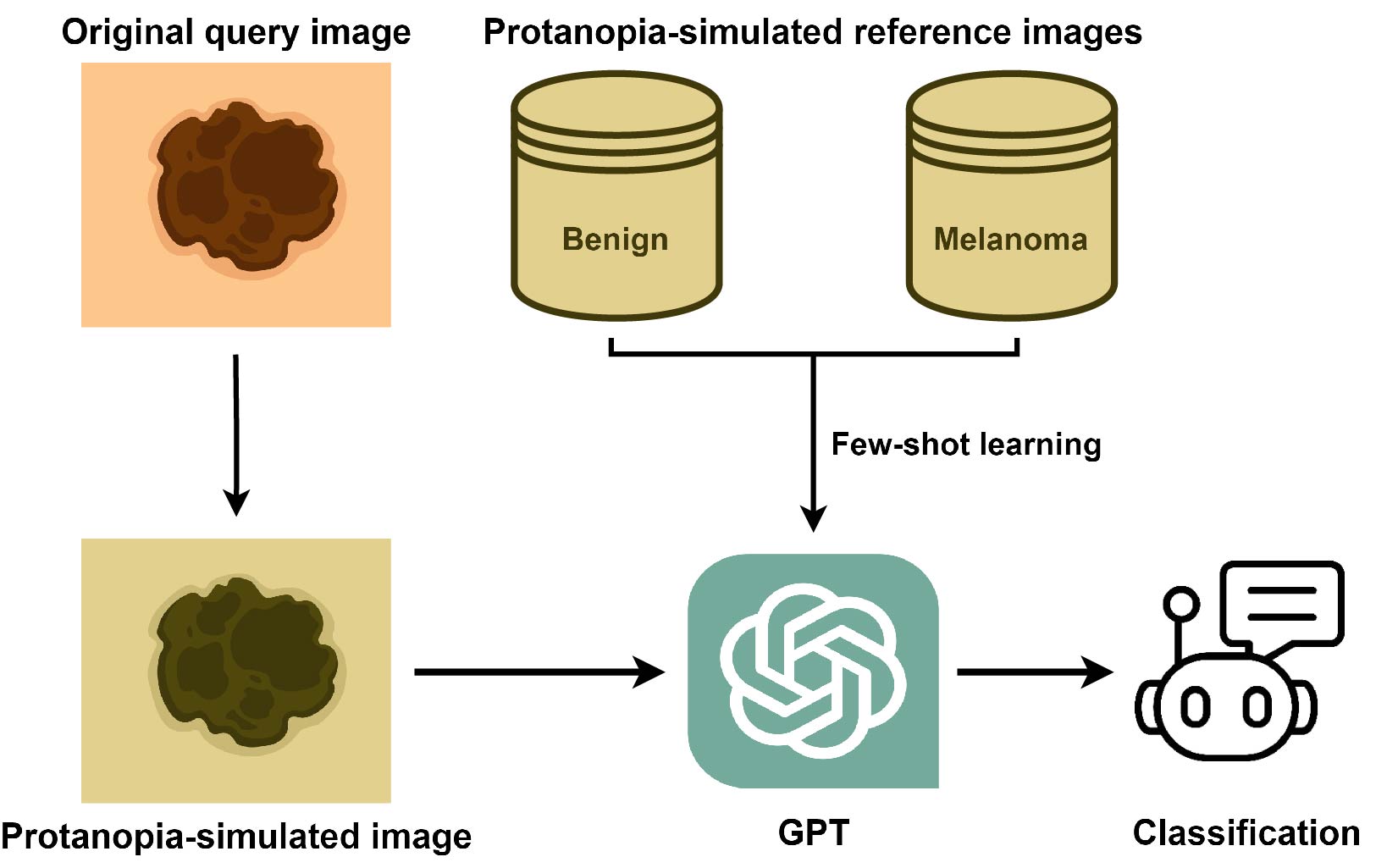
(A) Durchschnittliche Klassifizierungsgenauigkeiten von GPT-4V für nicht simulierte und cVD-simulierte Bilder (Protanopie, Deuteranopie, Tritanopie). Fehlerbalken: Standardabweichungen. *: p <0,05; **: P <0,01 (T-Test; Zwei-Schwanz). NS: Nicht signifikant. Alle Experimente waren in zehn Wiederholungen. (B) Klassifizierungsgenauigkeit von GPT-4V nach der Anwendung der Konsensstrategie über die zehn Wiederholungen für jede Bildabfrage. GPT-4O zum Vergleich enthalten.
GPT-4V passte seine Interpretation auf farbbasierte Ergebnisse an verschiedene CVD-Simulationen an. Insbesondere erwähnte es keine roten oder rosa Farben in Protanop- und Deuteranope-Simulationen, sondern diese Farben mit einer Melanom-Vorhersage in nicht simulierten Bildern. Bei Tritanopie-Simulationen-charakterisiert durch vorherrschende rosa Farbtöne-wurde GPT-4V nicht mehr als Melanom-Merkmal behandelt.
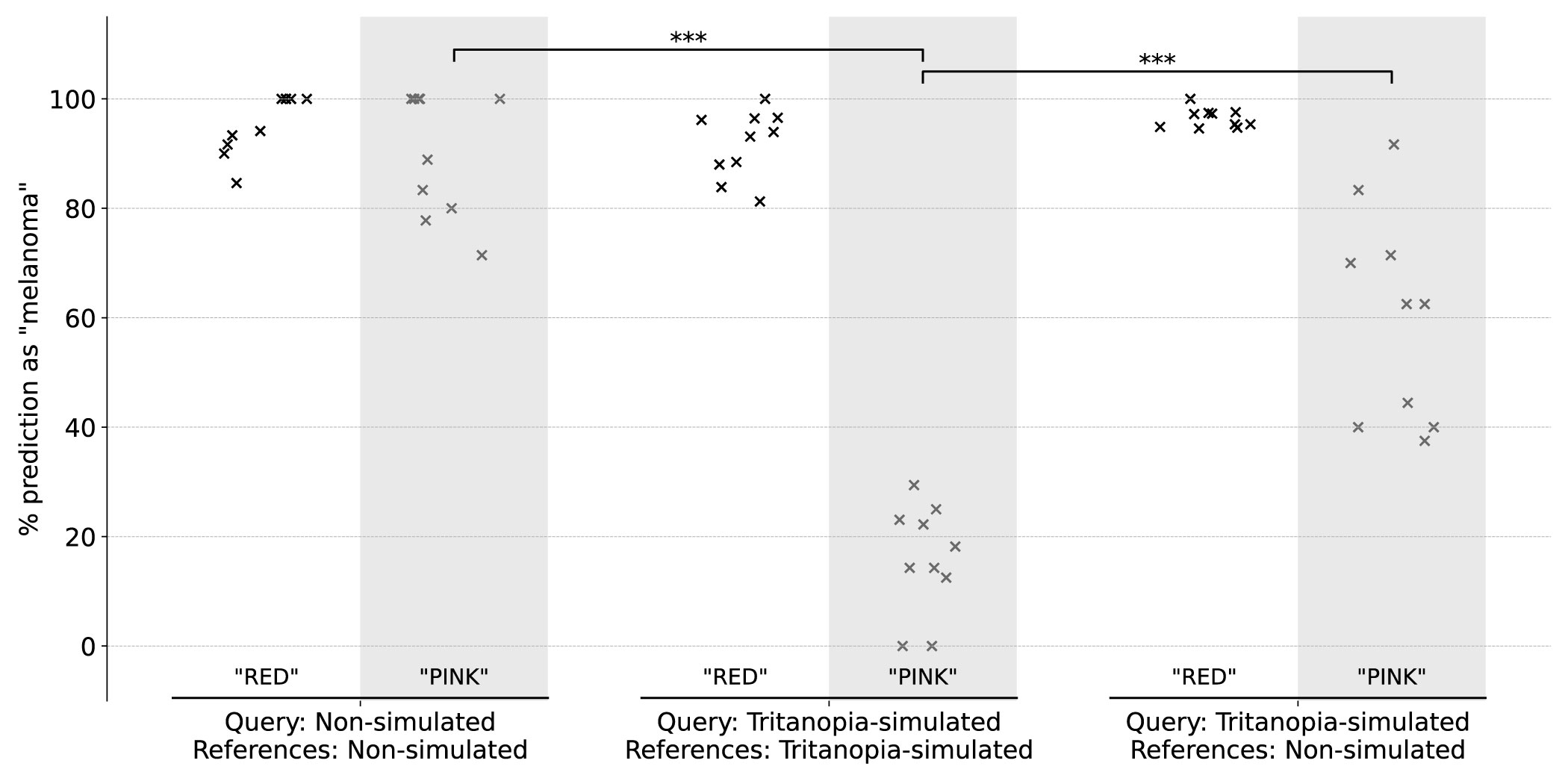
Für jede der nicht simulierten und tritanopie-simulierten Bedingungen wurden zwei Gruppen von Bilduntersuchungen anhand der Erklärungen von GPT-4V für seine Vorhersagen identifiziert. Die erste Gruppe, die als „Rot“ bezeichnet wird, enthält Beschreibungen, in denen rote Farbe erwähnt wird, um das Abfragebild zu beschreiben, unabhängig von der rosa Farbe. Die zweite Gruppe, die als „Pink“ bezeichnet wird, umfasst Beschreibungen, die ausschließlich Pink ohne Rot erwähnen. Die y-Achse zeigt den Prozentsatz der „Melanom“ -Vorvorhersagen aus jedem Replikat an. Standardmäßig wurden sowohl die Abfrage- als auch die Referenzbilder entweder nicht simuliert oder simuliert in derselben CVD-Bedingung. Für die Tritanopie-Simulation wurde ein Ablationstest (die letzten beiden Säulen) durchgeführt, indem die Referenzen aus nicht simulierten Bildern abgetastet wurden. ***: p <0,001 (T-Test; Zwei-Schwanz).