Papier: https://arxiv.org/abs/2310.00149
Autoren: Hao Liu, Jiarui Feng, Lecheng Kong, Ningyue Liang, Dacheng Tao, Yixin Chen, Muhan Zhang
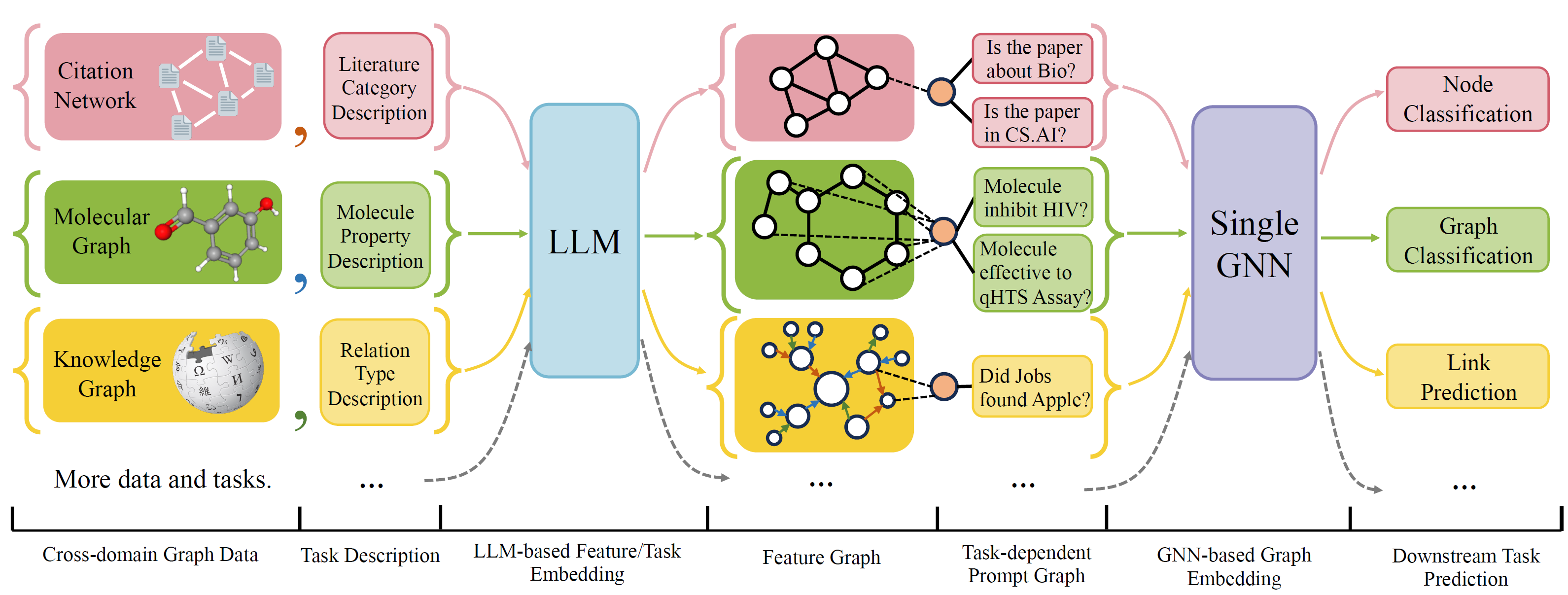
OFA ist ein allgemeines Graph -Klassifizierungs -Framework, das eine breite Palette von Graphenklassifizierungsaufgaben mit einem einzelnen Modell und einem einzelnen Satz von Parametern löst. Die Aufgaben sind Kreuzdomäne (z. B. Zitiernetzwerk, molekularer Graphen, ...) und Kreuzungen (z.
Verwenden Sie natürliche Sprachen, um alle Grafiken zu beschreiben und eine LLM zu verwenden, um alle Beschreibung in denselben Einbettungsraum einzubetten, wodurch das Cross-Domain-Training mit einem einzigen Modell ermöglicht wird.
OFA schlägt ein Auftragsparadiagm vor, dass alle Aufgabeninformationen in ein Umformpunkt konvertiert werden. Daher kann das Subsequence -Modell Informationen lesen und das Relavent -Ziel entsprechend vorhersagen, ohne Modellparameter und Architektur anzupassen. Daher kann ein einzelnes Modell eine Kreuzung sein.
Eine Liste von Diagrammdatensätzen aus verschiedenen Quellen und Domänen und beschreibt Knoten/Kanten in den Graphen mit einem systematischen Verringerungsprotokoll. Wir danken früheren Arbeiten, einschließlich OGB, Gimlet, Moleculenet, Graphlm und VillMow für die Bereitstellung wunderbarer RAW -Diagramm-/Textdaten, die unsere Arbeit ermöglichen.
OneForall wurde einer großen Überarbeitung unterzogen, bei der wir den Code aufräumten und mehrere gemeldete Fehler behoben hatten. Die Hauptaktualisierungen sind:
Wenn Sie zuvor unser Repository verwendet haben, ziehen Sie die alten generierten Funktionen/Textdateien und regenerieren Sie bitte ab. Entschuldigen Sie die Unannehmlichkeiten.
Um die Anforderungen für das Projekt mit Conda zu installieren:
conda env create -f environment.yml
Für gemeinsame End-to-End-Experimente auf allen gesammelten Datensatz
python run_cdm.py --override e2e_all_config.yaml
Alle Argumente können durch räumlich getrennte Werte wie z. B. geändert werden
python run_cdm.py --override e2e_all_config.yaml num_layers 7 batch_size 512 dropout 0.15 JK none
Benutzer können die Variable task_names in ./e2e_all_config.yaml ändern, um zu steuern, welche Datensätze während des Trainings enthalten sind. Die Länge von task_names , d_multiple und d_min_ratio sollte gleich sein. Sie können auch in Befehlszeilenargumenten durch Komma -getrennte Werte angegeben werden.
z.B
python run_cdm.py task_names cora_link,arxiv d_multiple 1,1 d_min_ratio 1,1
Ofa-ind kann durch angegeben werden durch
python run_cdm.py task_names cora_link d_multiple 1 d_min_ratio 1
Um die wenigen Experimente mit wenigen Schäden und Null-Shots durchzuführen
python run_cdm.py --override lr_all_config.yaml
Wir definieren Konfigurationen für jede Aufgabe, jede Aufgabekonfiguration enthält mehrere Datensätzekonfigurationen.
Aufgabenkonfigurationen werden in ./configs/task_config.yaml gespeichert. Eine Aufgabe besteht normalerweise aus mehreren Spaltungen von Datensätzen (nicht unbedingt die gleichen Datensätze). Beispielsweise wird in einer regelmäßigen End-to-End-Cora-Knotenklassifizierungsaufgabe die Zugspaltung des Cora-Datensatzes als Zugdatensatz, die gültige Aufteilung des Cora-Datensatzes als einen der gültigen Datensatz und ebenfalls für den Testteil aufgeteilt. Sie können auch mehr Validierung/Test durchführen, indem Sie die Zugspaltung der Cora als eine der Validierungs-/Testdatensätze angeben. Insbesondere sieht eine Aufgabenkonfiguration aus wie
arxiv :
eval_pool_mode : mean
dataset : arxiv # dataset name
eval_set_constructs :
- stage : train # a task should have one and only one train stage dataset
split_name : train
- stage : valid
split_name : valid
dataset : cora # replace the default dataset for zero-shot tasks
- stage : valid
split_name : valid
- stage : test
split_name : test
- stage : test
split_name : train # test the train split Datensatzkonfigurationen werden in ./configs/task_config.yaml gespeichert. Eine Datensatzkonfiguration definiert, wie ein Datensatz erstellt wird. Speziell,
arxiv :
task_level : e2e_node
preprocess : null # name of the preprocess function defined in task_constructor.py
construct : ConstructNodeCls # name of the dataset construction function defined in task_constructor.py
args : # additional arguments to construct function
walk_length : null
single_prompt_edge : True
eval_metric : acc # evaluation metric
eval_func : classification_func # evaluation function that process model output and batch to input to evaluator
eval_mode : max # evaluation mode (min/max)
dataset_name : arxiv # name of the OFAPygDataset
dataset_splitter : ArxivSplitter # splitting function defined in task_constructor.py
process_label_func : process_pth_label # name of process label function that transform original label to the binary labels
num_classes : 40 Wenn Sie einen Datensatz wie Cora/PubMed/Arxiv implementieren, empfehlen wir Ihnen, ein Verzeichnis Ihrer Daten hinzuzufügen $ Customized_data $ unter Daten/Single_Graph/$ Customized_data $ und implementieren Sie Gen_Data.py im Verzeichnis. PY als Beispiel.
Nachdem die Daten erstellt wurden, müssen Sie hier den Datensatznamen registrieren und einen Splitter wie hier implementieren. Wenn Sie Null-Shot/Wenig-Shot-Aufgaben ausführen, können Sie hier auch Null-Shot/Wenig-Shot-Split erhalten.
Registrieren Sie zuletzt einen Konfigurationseintrag in configs/data_config.yaml. Zum Beispiel für die End-to-End-Knotenklassifizierung
$data_name$ :
<< : *E2E-node
dataset_name : $data_name$
dataset_splitter : $splitter$
process_label_func : ... # usually processs_pth_label should work
num_classes : $number of classes$process_label_func konvertiert die Zielbezeichnung in die Binärbezeichnung und transformierte die Klasseneinbettung, wenn die Aufgabe null Schuss/wenige Shots ist, wobei die Anzahl der Klassenknoten nicht behoben ist. Eine Liste von Avalailable Process_Label_Func ist hier. Es nimmt alle Klassen ein, die einbettet und das richtige Etikett. Die Ausgabe ist ein Tupel: (Etikett, class_node_embedding, Binär-/One-Hot-Etikett).
Wenn Sie mehr Flexibilität wünschen, muss eine angepasste Unterklasse von OfapyGdataset implementiert werden. Eine Vorlage ist hier implementiert.
class CustomizedOFADataset ( OFAPygDataset ):
def gen_data ( self ):
"""
Returns a tuple of the following format
(data, text, extra)
data: a list of Pyg Data, if you only have a one large graph, you should still wrap it with the list.
text: a list of list of texts. e.g. [node_text, edge_text, label_text] this is will be converted to pooled vector representation.
extra: any extra data (e.g. split information) you want to save.
"""
def add_text_emb ( self , data_list , text_emb ):
"""
This function assigns generated embedding to member variables of the graph
data_list: data list returned in self.gen_data.
text_emb: list of torch text tensor corresponding to the returned text in self.gen_data. text_emb[0] = llm_encode(text[0])
"""
data_list [ 0 ]. node_text_feat = ... # corresponding node features
data_list [ 0 ]. edge_text_feat = ... # corresponding edge features
data_list [ 0 ]. class_node_text_feat = ... # class node features
data_list [ 0 ]. prompt_edge_text_feat = ... # edge features used in prompt node
data_list [ 0 ]. noi_node_text_feat = ... # noi node features, refer to the paper for the definition
return self . collate ( data_list )
def get_idx_split ( self ):
"""
Return the split information required to split the dataset, this optional, you can further split the dataset in task_constructor.py
"""
def get_task_map ( self ):
"""
Because a dataset can have multiple different tasks that requires different prompt/class text embedding. This function returns a task map that maps a task name to the desired text embedding. Specifically, a task map is of the following format.
prompt_text_map = {task_name1: {"noi_node_text_feat": ["noi_node_text_feat", [$Index in data[0].noi_node_text_feat$]],
"class_node_text_feat": ["class_node_text_feat",
[$Index in data[0].class_node_text_feat$]],
"prompt_edge_text_feat": ["prompt_edge_text_feat", [$Index in data[0].prompt_edge_text_feat$]]},
task_name2: similar to task_name 1}
Please refer to examples in data/ for details.
"""
return self . side_data [ - 1 ]
def get_edge_list ( self , mode = "e2e" ):
"""
Defines how to construct prompt graph
f2n: noi nodes to noi prompt node
n2f: noi prompt node to noi nodes
n2c: noi prompt node to class nodes
c2n: class nodes to noi prompt node
For different task/mode you might want to use different prompt graph construction, you can do so by returning a dictionary. For example
{"f2n":[1,0], "n2c":[2,0]} means you only want f2n and n2c edges, f2n edges have edge type 1, and its text embedding feature is data[0].prompt_edge_text_feat[0]
"""
if mode == "e2e_link" :
return { "f2n" : [ 1 , 0 ], "n2f" : [ 3 , 0 ], "n2c" : [ 2 , 0 ], "c2n" : [ 4 , 0 ]}
elif mode == "lr_link" :
return { "f2n" : [ 1 , 0 ], "n2f" : [ 3 , 0 ]}