Tenga en cuenta que este repositorio es sólo para fines de archivo. Las funciones ANCOM independientes en este repositorio ya no se mantienen. Se recomienda encarecidamente a los usuarios que utilicen las funciones ancom o ancombc en nuestro paquete ANCOMBC R. Para obtener informes de errores, vaya al repositorio de ANCOMBC.
El código actual implementa ANCOM en conjuntos de datos transversales y longitudinales al tiempo que permite el uso de covariables. Es necesario incluir las siguientes bibliotecas para que se ejecute el código R:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Adoptamos la metodología de ANCOM-II como paso de preprocesamiento para lidiar con diferentes tipos de ceros antes de realizar el análisis de abundancia diferencial.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : marco de datos o matriz que representa la tabla OTU/SV observada con taxones en filas ( rownames ) y muestras en columnas ( colnames ). Tenga en cuenta que esta es la tabla de abundancia absoluta , no la transforme en una tabla de abundancia relativa (donde los totales de las columnas son iguales a 1).meta_data : marco de datos o matriz de todas las variables y covariables de interés.sample_var : Carácter. El nombre de la columna que almacena los ID de muestra.group_var : Carácter. El nombre del indicador de grupo. group_var es necesario para detectar ceros estructurales y valores atípicos. Para conocer las definiciones de diferentes ceros (cero estructural, cero atípico y cero de muestreo), consulte ANCOM-II.out_cut Fracción numérica entre 0 y 1. Para cada taxón, las observaciones con una proporción de distribución de la mezcla menor que out_cut se detectarán como ceros atípicos; mientras que las observaciones con una proporción de distribución de la mezcla mayor que 1 - out_cut se detectarán como valores atípicos.zero_cut : Fracción numérica entre 0 y 1. Los taxones con proporción de ceros mayor que zero_cut no se incluyen en el análisis.lib_cut : numérico. Las muestras con un tamaño de biblioteca inferior a lib_cut no se incluyen en el análisis.neg_lb : Lógico. VERDADERO indica que un taxón se clasificaría como cero estructural en el grupo experimental correspondiente utilizando su límite inferior asintótico. Más específicamente, neg_lb = TRUE indica que está utilizando ambos criterios establecidos en la sección 3.2 de ANCOM-II para detectar ceros estructurales; De lo contrario, neg_lb = FALSE solo usará la ecuación 1 en la sección 3.2 de ANCOM-II para declarar ceros estructurales. feature_table : un marco de datos de una tabla OTU preprocesada.meta_data : un marco de datos de metadatos preprocesados.structure_zeros : una matriz consta de 0 y 1, donde 1 indica que el taxón está identificado como un cero estructural en el grupo correspondiente.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : marco de datos que representa la tabla OTU/SV con taxones en filas ( rownames ) y muestras en columnas ( colnames ). Puede ser el valor de salida de feature_table_pre_process . Tenga en cuenta que esta es la tabla de abundancia absoluta , no la transforme en una tabla de abundancia relativa (donde los totales de las columnas son iguales a 1).meta_data : marco de datos de variables. Puede ser el valor de salida de feature_table_pre_process .struc_zero : una matriz consta de 0 y 1, donde 1 indica que el taxón está identificado como un cero estructural en el grupo correspondiente. Puede ser el valor de salida de feature_table_pre_process .main_var : Carácter. El nombre de la principal variable de interés. ANCOM v2.1 actualmente admite main_var categórico.p_adjust_method : Carácter. Especificación del método para ajustar los valores p para comparaciones múltiples. El valor predeterminado es “BH” (procedimiento Benjamin-Hochberg).alpha : Nivel de significancia. El valor predeterminado es 0,05.adj_formula : Cadena de caracteres que representa la fórmula de ajuste (ver ejemplo).rand_formula : Cadena de caracteres que representa la fórmula para efectos aleatorios en lme . Para obtener más información, consulte ?lme .lme_control : una lista que especifica valores de control para lme fit. Para obtener más información, consulte ?lmeControl . 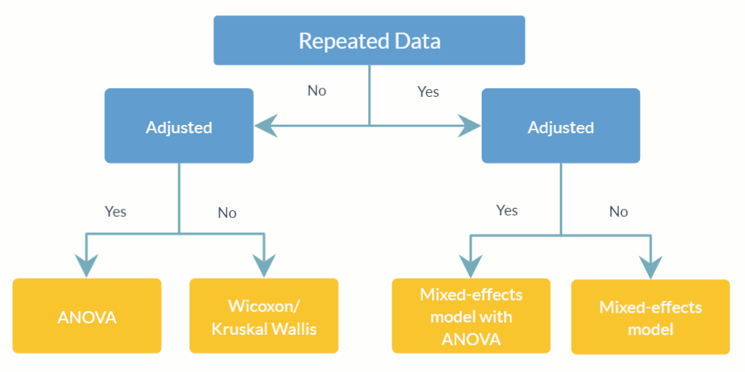
out : un marco de datos con la estadística W para cada taxón y columnas posteriores que son indicadores lógicos de si una OTU o taxón es diferencialmente abundante bajo una serie de límites (0,9, 0,8, 0,7 y 0,6). detected_0.7 se usa comúnmente. Sin embargo, puede elegir detected_0.8 o detected_0.9 si desea ser más conservador en sus resultados (FDR más pequeño), o usar detected_0.6 si desea explorar más descubrimientos (mayor potencia)
fig : Un objeto ggplot del diagrama del volcán.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Aquí nos gustaría saber si existen algunos ceros estructurales en los diferentes niveles de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Aquí nos gustaría saber si existen algunos ceros estructurales en los diferentes niveles de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Aquí nos gustaría saber si existen algunos ceros estructurales en los diferentes niveles de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )