Carga rápida de datos por lotes de archivos BigWig que contienen datos de seguimiento epigenético y secuencias correspondientes impulsadas por GPU para aplicaciones de aprendizaje profundo.
Bigwig-loader depende principalmente de la biblioteca rapidsai kvikio y cupy, las cuales se instalan mejor usando conda/mamba. Bigwig-loader ahora también se puede instalar usando conda/mamba. Para crear un nuevo entorno con bigwig-loader instalado:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderO agregue esto a su archivo Environment.yml:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loadery actualizar:
mamba env update -f environment.ymlBigwig-loader también se puede instalar usando pip en un entorno que ya tenga instalada la biblioteca rapidsai kvikio y cupy:
pip install bigwig-loaderEnvolvimos BigWigDataset en un conjunto de datos iterable de PyTorch que puedes usar directamente:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Se puede importar un objeto Dataset independiente del marco desde bigwig_loader.dataset . Este objeto de conjunto de datos devuelve tensores de cupy. Los tensores de Cupy se adhieren a la interfaz de matriz cuda y se pueden transformar con copia cero en tensores JAX o tensorflow.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Consulte el directorio de ejemplos para obtener más ejemplos.
Esta biblioteca está diseñada para cargar lotes de datos con la misma dimensionalidad, lo que permite algunas suposiciones que pueden acelerar el proceso de carga. Como se puede ver en el gráfico siguiente, cuando se carga una pequeña cantidad de datos, pyBigWig es muy rápido, pero no explota la naturaleza por lotes de la carga de datos para el aprendizaje automático.
En el punto de referencia a continuación, también creamos cargadores de datos de PyTorch (con set_start_method('spawn')) usando pyBigWig para compararlo con el escenario realista donde se usarían múltiples CPU por GPU. Vemos que el rendimiento del cargador de datos de la CPU no aumenta linealmente con la cantidad de CPU y, por lo tanto, resulta difícil obtener el rendimiento necesario para mantener la GPU, entrenando la red neuronal, saturada durante los pasos de aprendizaje.
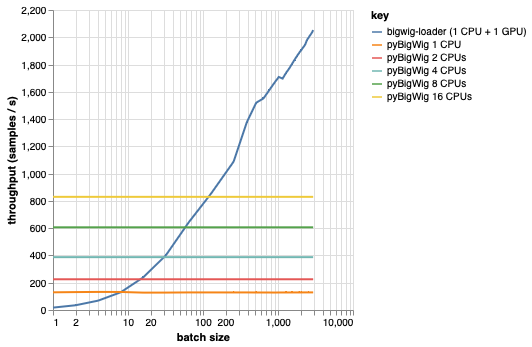
Este es el problema que resuelve bigwig-loader. Este es un ejemplo de cómo utilizar bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml En este entorno, debería poder ejecutar pytest -v y ver que las pruebas se realizan correctamente. NOTA: ¡necesita una GPU para usar bigwig-loader!
Esta sección lo guía a través de los pasos necesarios para agregar nuevas funciones. Si algo no está claro, abra un problema.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install para instalar los ganchos de confirmación previaLas pruebas están en el directorio de pruebas. Una de las pruebas más importantes es test_against_pybigwig, que garantiza que si hay un error en pyBigWIg, también lo esté en bigwig-loader.
pytest -vv .Cuando los ejecutores de GitHub con GPU estén disponibles, también nos gustaría ejecutar estas pruebas en el CI. Pero por ahora, puedes ejecutarlos localmente.
Si utiliza esta biblioteca, considere citar:
Retel, Joren Sebastian, Andreas Poehlmann, Josh Chiou, Andreas Steffen y Djork-Arné Clevert. "Un cargador de datos rápido de aprendizaje automático para pistas epigenéticas de archivos BigWig". Bioinformática 40, núm. 1 (1 de enero de 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}