
(c) 2017 Timothy Becker y Wan-Ping Lee
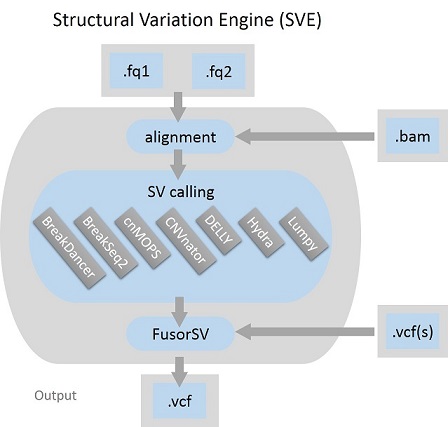
SVE es un motor de ejecución basado en script de Python para la detección de variaciones estructurales (SV) y se puede utilizar para cualquier nivel de entrada de datos, FASTQ sin procesar, BAM alineados o formato de llamada variante (VCF), y genera un VCF unificado como salida. Por diseño, SVE consta de alineación, realineación y un conjunto de algoritmos de llamada SV de última generación de forma predeterminada. Son BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra y LUMPY. FusorSV también está integrado, que es un enfoque de minería de datos para evaluar el rendimiento y fusionar conjuntos de llamadas de un conjunto de algoritmos de llamadas SV.
Configure el entorno ROOT.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeVerifique los archivos de encabezado de Python2.7 y modifique "CFLAGS_FUSOR_SV" en Makefile. Los archivos de encabezado pueden estar en "/usr/include/python2.7" y usar "CFLAGS_FUSOR_SV=-I /usr/include/python2.7" en su lugar.
make FusorSV
O puede instalar FusorSV mediante setup.py
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
Alternativamente, se proporcionan Dockerfile y la imagen de Docker. Tenga en cuenta que es posible que se requiera sudo para los usos de Docker según la configuración de su máquina.
cd SVE
docker build .
Extraiga la imagen de la ventana acoplable del repositorio.
docker pull wanpinglee/sve
SVE está construido en /tools/SVE. Consulta la ayuda por
/tools/SVE/bin/sve
Las lecturas cortas en FASTQ se asignarán al FASTA dado y se generará un BAM ordenado.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
Si las lecturas se proporcionan en formato BAM, la realineación reasignará las lecturas con FASTA y generará un BAM ordenado. Usamos SpeedSeq para lograr la realineación.
bin/sve realign -r <FASTA> <BAM>
Hay siete algoritmos de llamadas SV que se pueden utilizar para llamadas SV. Se generará VCF.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
Después de llamar, cada muestra puede tener varios VCF dependiendo de cuántas personas llamaron. Recopile los VCF de una muestra en una carpeta.
Los vcfs deben usar ID de SVE para indicar las personas que llaman.
| ID SVE | Llamador |
|---|---|
| 4 | BreakDancer (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnador (v0.3.3) |
| 11 | DELLY (v2) |
| 14* | GenomaSTRiP |
| 17 | Hidra |
| 18 | ATERRONADO |
| 35 | BreakSeq (v2.2) |
| 0 | Verdad (opcional) |
Nota*: Debido a un problema de licencia, GenomeSTRiP no está integrado en SVE. Sin embargo, el modelo predeterminado de FusorSV es capaz de manejar GenomeSTRiP VCF.
Los archivos vcf de entrada de ejemplo se pueden organizar de la siguiente manera. Tenga en cuenta que vcfFiles es el argumento para -i para FusorSV.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Según S0.vcf, se generará un nuevo modelo y el nuevo modelo fusionará los VCF.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
El proyecto está bajo la Licencia GPL-3.0. Consulte LICENCIA para obtener más detalles.