Importante
Consulte el nuevo modelo tabular de DL: TabM
arXiv? Paquete Python Otros proyectos DL tabulares
Esta es la implementación oficial del documento "Sobre incrustaciones de características numéricas en el aprendizaje profundo tabular".
En una frase: transformar las características continuas escalares originales en vectores antes de mezclarlas en la red troncal principal (por ejemplo, en MLP, Transformer, etc.) mejora el rendimiento posterior de las redes neuronales tabulares.
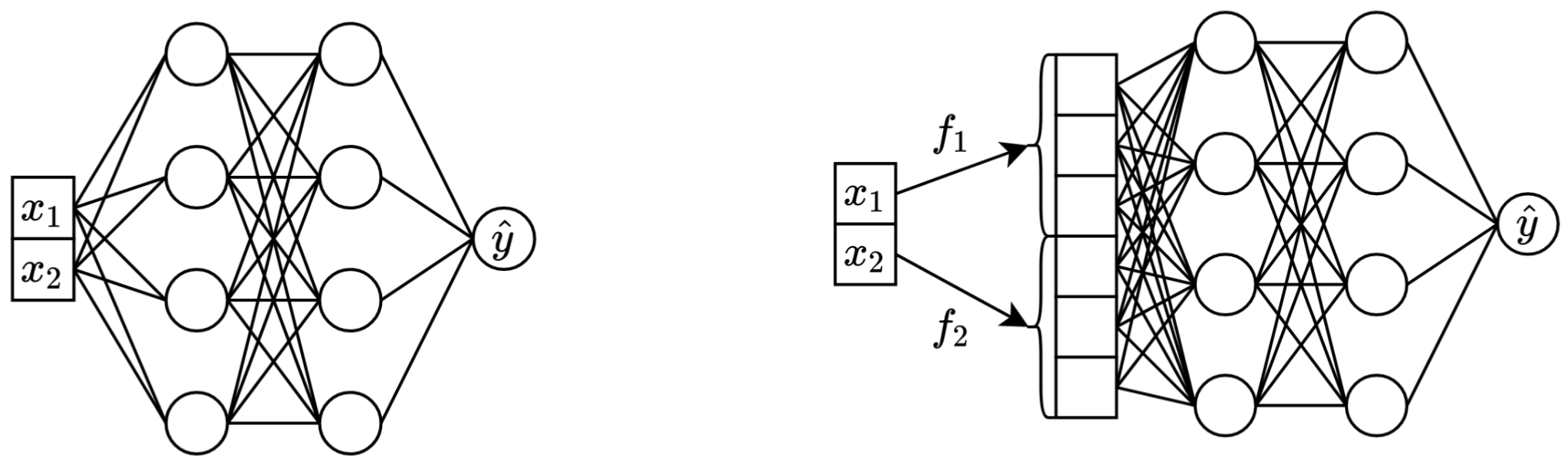
Izquierda: Vanilla MLP tomando dos características continuas como entrada.
Derecha: el mismo MLP, pero ahora con incrustaciones para funciones continuas.
Más detalladamente:
En rigor, no existe una única explicación. Evidentemente, las incorporaciones ayudan a afrontar diversos desafíos asociados con las características continuas y mejoran las propiedades generales de optimización de los modelos.
En particular, las características continuas distribuidas irregularmente (y sus distribuciones conjuntas irregulares con etiquetas) son algo habitual en los datos tabulares del mundo real y plantean un importante desafío de optimización fundamental para los modelos DL tabulares tradicionales. Una gran referencia para comprender este desafío (y un gran ejemplo de cómo abordar esos desafíos transformando el espacio de entrada) es el artículo "Las características de Fourier permiten que las redes aprendan funciones de alta frecuencia en dominios de baja dimensión".
Sin embargo, no está claro si las distribuciones irregulares son la única razón por la que las incrustaciones son útiles.
El paquete Python en el directorio package/ es la forma recomendada de utilizar el documento en la práctica y para trabajos futuros.
El resto del documento :
El directorio exp/ contiene numerosos resultados e hiperparámetros (ajustados) para varios modelos y conjuntos de datos utilizados en el artículo.
Por ejemplo, exploremos las métricas del modelo MLP. Primero, carguemos los informes (los archivos report.json ):
import json
from pathlib import Path
import pandas as pd
df = pd . json_normalize ([
json . loads ( x . read_text ())
for x in Path ( 'exp' ). glob ( 'mlp/*/0_evaluation/*/report.json' )
])Ahora, para cada conjunto de datos, calculemos el puntaje de la prueba promediado de todas las semillas aleatorias:
print ( df . groupby ( 'config.data.path' )[ 'metrics.test.score' ]. mean (). round ( 3 ))El resultado coincide exactamente con la Tabla 3 del artículo:
config.data.path
data/adult 0.854
data/california -0.495
data/churn 0.856
data/covtype 0.964
data/fb-comments -5.686
data/gesture 0.632
data/higgs-small 0.720
data/house -32039.399
data/microsoft -0.747
data/otto 0.818
data/santander 0.912
Name: metrics.test.score, dtype: float64
El enfoque anterior también se puede utilizar para explorar hiperparámetros y obtener intuición sobre los valores de hiperparámetros típicos para diferentes algoritmos. Por ejemplo, así es como se puede calcular la tasa de aprendizaje ajustada media para el modelo MLP:
Nota
Para algunos algoritmos (por ejemplo, MLP, MLP-LR, MLP-PLR), proyectos más recientes ofrecen más resultados que pueden explorarse de manera similar. Por ejemplo, consulte este artículo sobre TabR.
Advertencia
Utilice este enfoque con precaución. Al estudiar valores de hiperparámetros:
print ( df [ df [ 'config.seed' ] == 0 ][ 'config.training.lr' ]. quantile ( 0.5 ))
# Output: 0.0002716544410603358Importante
Esta sección es larga. Utilice la función "Esquema" en GitHub en su editor de texto para obtener una descripción general de esta sección.
Preliminares:
/usr/local/cuda-11.1/bin esté siempre en su variable de entorno PATH export PROJECT_DIR= < ABSOLUTE path to the repository root >
# example: export PROJECT_DIR=/home/myusername/repositories/num-embeddings
git clone https://github.com/yandex-research/tabular-dl-num-embeddings $PROJECT_DIR
cd $PROJECT_DIR
conda create -n num-embeddings python=3.9.7
conda activate num-embeddings
pip install torch==1.10.1+cu111 -f https://download.pytorch.org/whl/torch_stable.html
pip install -r requirements.txt
# if the following commands do not succeed, update conda
conda env config vars set PYTHONPATH= ${PYTHONPATH} : ${PROJECT_DIR}
conda env config vars set PROJECT_DIR= ${PROJECT_DIR}
# the following command appends ":/usr/local/cuda-11.1/lib64" to LD_LIBRARY_PATH;
# if your LD_LIBRARY_PATH already contains a path to some other CUDA, then the content
# after "=" should be "<your LD_LIBRARY_PATH without your cuda path>:/usr/local/cuda-11.1/lib64"
conda env config vars set LD_LIBRARY_PATH= ${LD_LIBRARY_PATH} :/usr/local/cuda-11.1/lib64
conda env config vars set CUDA_HOME=/usr/local/cuda-11.1
conda env config vars set CUDA_ROOT=/usr/local/cuda-11.1
# (optional) get a shortcut for toggling the dark mode with cmd+y
conda install nodejs
jupyter labextension install jupyterlab-theme-toggle
conda deactivate
conda activate num-embeddingsLICENCIA: al descargar nuestro conjunto de datos aceptas las licencias de todos sus componentes. No imponemos ninguna restricción nueva además de esas licencias. Puede encontrar la lista de fuentes en el artículo.
cd $PROJECT_DIR
wget " https://www.dropbox.com/s/r0ef3ij3wl049gl/data.tar?dl=1 " -O num_embeddings_data.tar
tar -xvf num_embeddings_data.tarEl siguiente código reproduce los resultados de MLP en el conjunto de datos de Vivienda de California. El proceso para otros algoritmos y conjuntos de datos es absolutamente el mismo.
# You must explicitly set CUDA_VISIBLE_DEVICES if you want to use GPU
export CUDA_VISIBLE_DEVICES="0"
# Create a copy of the 'official' config
cp exp/mlp/california/0_tuning.toml exp/mlp/california/1_tuning.toml
# Run tuning (on GPU, it takes ~30-60min)
python bin/tune.py exp/mlp/california/1_tuning.toml
# Evaluate single models with 15 different random seeds
python bin/evaluate.py exp/mlp/california/1_tuning 15
# Evaluate ensembles (by default, three ensembles of size five each)
python bin/ensemble.py exp/mlp/california/1_evaluation
La sección "Métricas" muestra cómo resumir los resultados obtenidos.
El código está organizado de la siguiente manera:
bintrain4.py para redes neuronales (implementa todas las incorporaciones y columnas vertebrales del artículo)xgboost_.py para XGBoostcatboost_.py para CatBoosttune.py para sintonizarevaluate.py para evaluaciónensemble.py para ensamblardatasets.py se utilizó para crear las divisiones del conjunto de datossynthetic.py para generar conjuntos de datos sintéticos compatibles con GBDT.train1_synthetic.py para los experimentos con datos sintéticoslib contiene herramientas comunes utilizadas por los programas en binexp contiene configuraciones y resultados del experimento (métricas, configuraciones ajustadas, etc.). Los nombres de las carpetas anidadas siguen los nombres del artículo (ejemplo: exp/mlp-plr corresponde al modelo MLP-PLR del artículo).package contiene el paquete Python para este documento.CUDA_VISIBLE_DEVICES al ejecutar scriptslib.dump_config y lib.load_config en lugar de bibliotecas TOML simplesEl patrón común para ejecutar scripts es:
python bin/my_script.py a/b/c.toml donde a/b/c.toml es el archivo de configuración de entrada (config). La salida estará ubicada en a/b/c . La estructura de configuración generalmente sigue la clase Config de bin/my_script.py .
También hay scripts que toman argumentos de línea de comando en lugar de configuraciones (por ejemplo, bin/{evaluate.py,ensemble.py} ).
Los necesita todos para reproducir resultados, pero solo necesita train4.py para trabajos futuros, porque:
bin/train1.py implementa un superconjunto de características de bin/train0.pybin/train3.py implementa un superconjunto de características de bin/train1.pybin/train4.py implementa un superconjunto de características de bin/train3.py Para ver cuál de los cuatro scripts se utilizó para ejecutar un experimento determinado, verifique el campo "programa" de la configuración de ajuste correspondiente. Por ejemplo, aquí está la configuración de ajuste para MLP en el conjunto de datos de Vivienda de California: exp/mlp/california/0_tuning.toml . La configuración indica que se utilizó bin/train0.py . Significa que las configuraciones en exp/mlp/california/0_evaluation son compatibles específicamente con bin/train0.py . Para verificar eso, puede copiar uno de ellos en una ubicación separada y pasarlo a bin/train0.py :
mkdir exp/tmp
cp exp/mlp/california/0_evaluation/0.toml exp/tmp/0.toml
python bin/train0.py exp/tmp/0.toml
ls exp/tmp/0
@inproceedings{gorishniy2022embeddings,
title={On Embeddings for Numerical Features in Tabular Deep Learning},
author={Yury Gorishniy and Ivan Rubachev and Artem Babenko},
booktitle={{NeurIPS}},
year={2022},
}