Una encantadora herramienta de aprendizaje automático que le permite entrenar/adaptar, probar y usar modelos sin escribir código
Nota
También estoy trabajando en una aplicación de escritorio GUI para igel basada en las solicitudes de las personas. Puede encontrarlo en Igel-UI.
Tabla de contenido
El objetivo del proyecto es proporcionar aprendizaje automático para todos , tanto usuarios técnicos como no técnicos.
A veces necesitaba una herramienta que pudiera usar para crear rápidamente un prototipo de aprendizaje automático. Ya sea para crear alguna prueba de concepto, crear un modelo borrador rápido para demostrar un punto o utilizar el aprendizaje automático automático. A menudo me encuentro estancado escribiendo código repetitivo y pensando demasiado por dónde empezar. Por eso, decidí crear esta herramienta.
igel está construido sobre otros marcos de ML. Proporciona una forma sencilla de utilizar el aprendizaje automático sin escribir una sola línea de código . Igel es altamente personalizable , pero sólo si así lo deseas. Igel no te obliga a personalizar nada. Además de los valores predeterminados, igel puede usar funciones de ml automático para encontrar un modelo que pueda funcionar muy bien con sus datos.
Todo lo que necesitas es un archivo yaml (o json ), donde debes describir lo que estás intentando hacer. ¡Eso es todo!
Igel apoya la regresión, la clasificación y la agrupación. Igel's admite funciones de ml automático como ImageClassification y TextClassification
Igel admite los tipos de conjuntos de datos más utilizados en el campo de la ciencia de datos. Por ejemplo, su conjunto de datos de entrada puede ser un archivo csv, txt, hoja de Excel, json o incluso html que desee recuperar. Si está utilizando funciones de ml automático, incluso puede enviar datos sin procesar a igel y éste descubrirá cómo manejarlos. Más sobre esto más adelante en los ejemplos.
$ pip install -U igel Modelos compatibles de Igel:
+--------------------+----------------------------+-------------------------+
| regression | classification | clustering |
+--------------------+----------------------------+-------------------------+
| LinearRegression | LogisticRegression | KMeans |
| Lasso | Ridge | AffinityPropagation |
| LassoLars | DecisionTree | Birch |
| BayesianRegression | ExtraTree | AgglomerativeClustering |
| HuberRegression | RandomForest | FeatureAgglomeration |
| Ridge | ExtraTrees | DBSCAN |
| PoissonRegression | SVM | MiniBatchKMeans |
| ARDRegression | LinearSVM | SpectralBiclustering |
| TweedieRegression | NuSVM | SpectralCoclustering |
| TheilSenRegression | NearestNeighbor | SpectralClustering |
| GammaRegression | NeuralNetwork | MeanShift |
| RANSACRegression | PassiveAgressiveClassifier | OPTICS |
| DecisionTree | Perceptron | KMedoids |
| ExtraTree | BernoulliRBM | ---- |
| RandomForest | BoltzmannMachine | ---- |
| ExtraTrees | CalibratedClassifier | ---- |
| SVM | Adaboost | ---- |
| LinearSVM | Bagging | ---- |
| NuSVM | GradientBoosting | ---- |
| NearestNeighbor | BernoulliNaiveBayes | ---- |
| NeuralNetwork | CategoricalNaiveBayes | ---- |
| ElasticNet | ComplementNaiveBayes | ---- |
| BernoulliRBM | GaussianNaiveBayes | ---- |
| BoltzmannMachine | MultinomialNaiveBayes | ---- |
| Adaboost | ---- | ---- |
| Bagging | ---- | ---- |
| GradientBoosting | ---- | ---- |
+--------------------+----------------------------+-------------------------+Para aprendizaje automático automático:
El comando de ayuda es muy útil para verificar los comandos admitidos y los argumentos/opciones correspondientes.
$ igel --helpTambién puede ejecutar ayuda sobre subcomandos, por ejemplo:
$ igel fit --helpIgel es altamente personalizable. Si sabe lo que quiere y desea configurar su modelo manualmente, consulte las siguientes secciones, que lo guiarán sobre cómo escribir un archivo de configuración yaml o json. Después de eso, sólo tienes que decirle a igel qué hacer y dónde encontrar tus datos y tu archivo de configuración. Aquí hay un ejemplo:
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml 'Sin embargo, también puedes utilizar las funciones de ml automático y dejar que igel haga todo por ti. Un gran ejemplo de esto sería la clasificación de imágenes. Imaginemos que ya tiene un conjunto de datos de imágenes sin procesar almacenadas en una carpeta llamada imágenes.
Todo lo que tienes que hacer es ejecutar:
$ igel auto-train --data_path ' path_to_your_images_folder ' --task ImageClassification¡Eso es todo! Igel leerá las imágenes del directorio, procesará el conjunto de datos (convirtiéndolo a matrices, reescalándolo, dividiéndolo, etc.) y comenzará a entrenar/optimizar un modelo que funcione bien con sus datos. Como puedes ver es bastante fácil, sólo debes proporcionar la ruta a tus datos y la tarea que deseas realizar.
Nota
Esta característica es computacionalmente costosa ya que igel probaría muchos modelos diferentes y compararía su rendimiento para encontrar el "mejor".
Puede ejecutar el comando de ayuda para obtener instrucciones. ¡También puedes ejecutar ayuda en subcomandos!
$ igel --helpEl primer paso es proporcionar un archivo yaml (también puedes usar json si quieres)
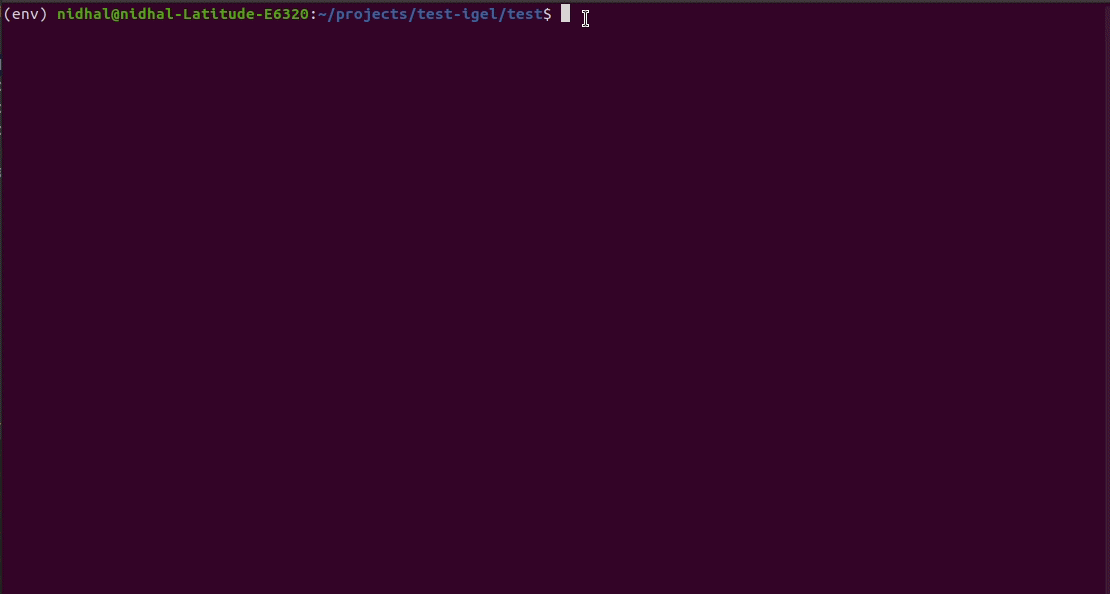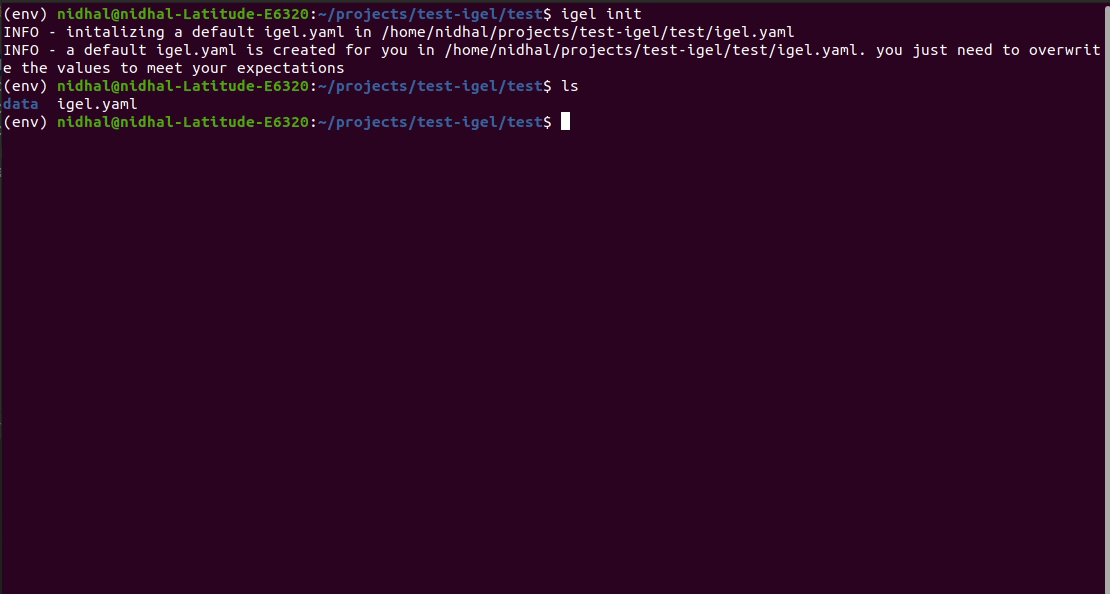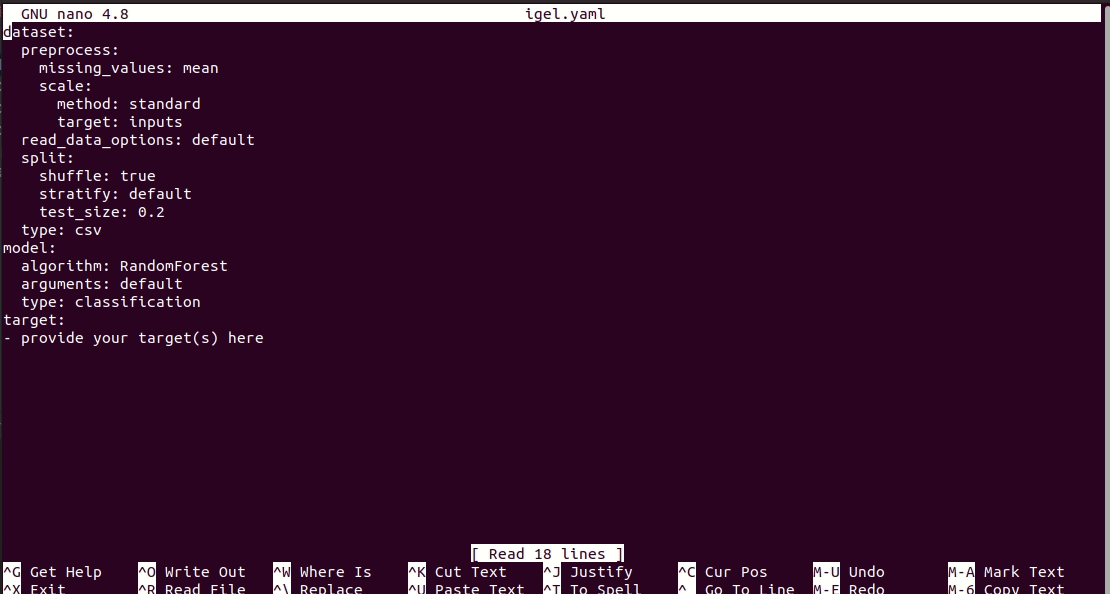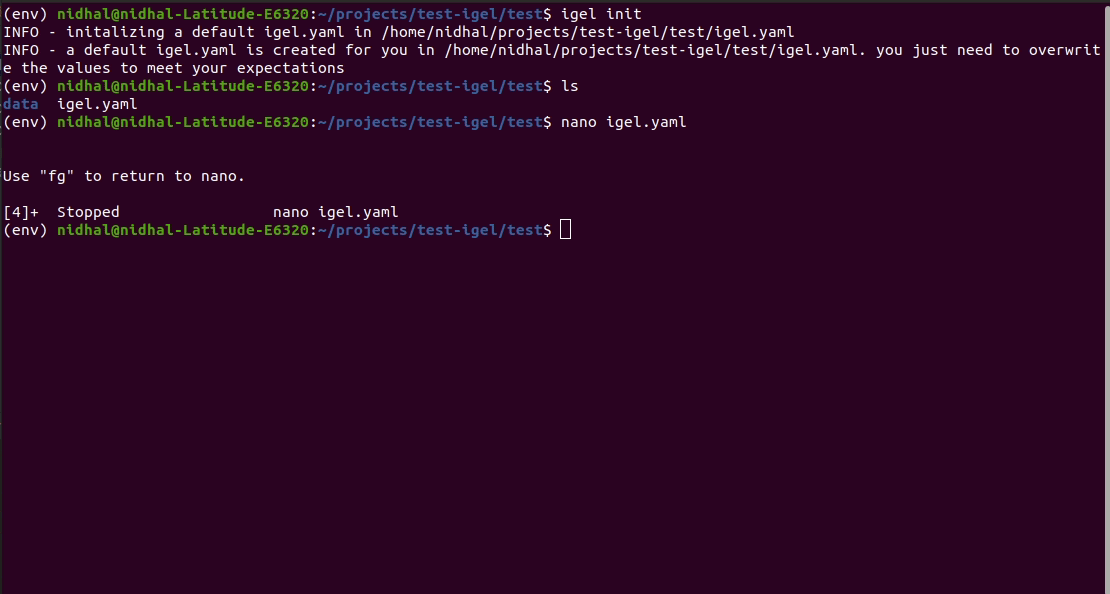
Puedes hacer esto manualmente creando un archivo .yaml (llamado igel.yaml por convención pero puedes nombrarlo si lo deseas) y editarlo tú mismo. Sin embargo, si eres vago (y probablemente lo seas, como yo :D), puedes usar el comando igel init para comenzar rápidamente, lo que creará un archivo de configuración básico sobre la marcha.
"""
igel init --help
Example:
If I want to use neural networks to classify whether someone is sick or not using the indian-diabetes dataset,
then I would use this command to initialize a yaml file n.b. you may need to rename outcome column in .csv to sick:
$ igel init -type " classification " -model " NeuralNetwork " -target " sick "
"""
$ igel initDespués de ejecutar el comando, se creará un archivo igel.yaml en el directorio de trabajo actual. Puedes consultarlo y modificarlo si lo deseas; de lo contrario, también puedes crear todo desde cero.
# model definition
model :
# in the type field, you can write the type of problem you want to solve. Whether regression, classification or clustering
# Then, provide the algorithm you want to use on the data. Here I'm using the random forest algorithm
type : classification
algorithm : RandomForest # make sure you write the name of the algorithm in pascal case
arguments :
n_estimators : 100 # here, I set the number of estimators (or trees) to 100
max_depth : 30 # set the max_depth of the tree
# target you want to predict
# Here, as an example, I'm using the famous indians-diabetes dataset, where I want to predict whether someone have diabetes or not.
# Depending on your data, you need to provide the target(s) you want to predict here
target :
- sickEn el ejemplo anterior, estoy usando un bosque aleatorio para clasificar si alguien tiene diabetes o no, dependiendo de algunas características del conjunto de datos. Usé la famosa diabetes india en este ejemplo del conjunto de datos de diabetes india).
Observe que pasé n_estimators y max_depth como argumentos adicionales para el modelo. Si no proporciona argumentos, se utilizará el valor predeterminado. No es necesario memorizar los argumentos de cada modelo. Siempre puedes ejecutar igel models en tu terminal, lo que te llevará al modo interactivo, donde se te pedirá que ingreses el modelo que deseas usar y el tipo de problema que deseas resolver. Luego, Igel le mostrará información sobre el modelo y un enlace que puede seguir para ver una lista de argumentos disponibles y cómo usarlos.
Ejecute este comando en la terminal para ajustar/entrenar un modelo, donde proporciona la ruta a su conjunto de datos y la ruta al archivo yaml.
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml '
# or shorter
$ igel fit -dp ' path_to_your_csv_dataset.csv ' -yml ' path_to_your_yaml_file.yaml '
"""
That's it. Your "trained" model can be now found in the model_results folder
(automatically created for you in your current working directory).
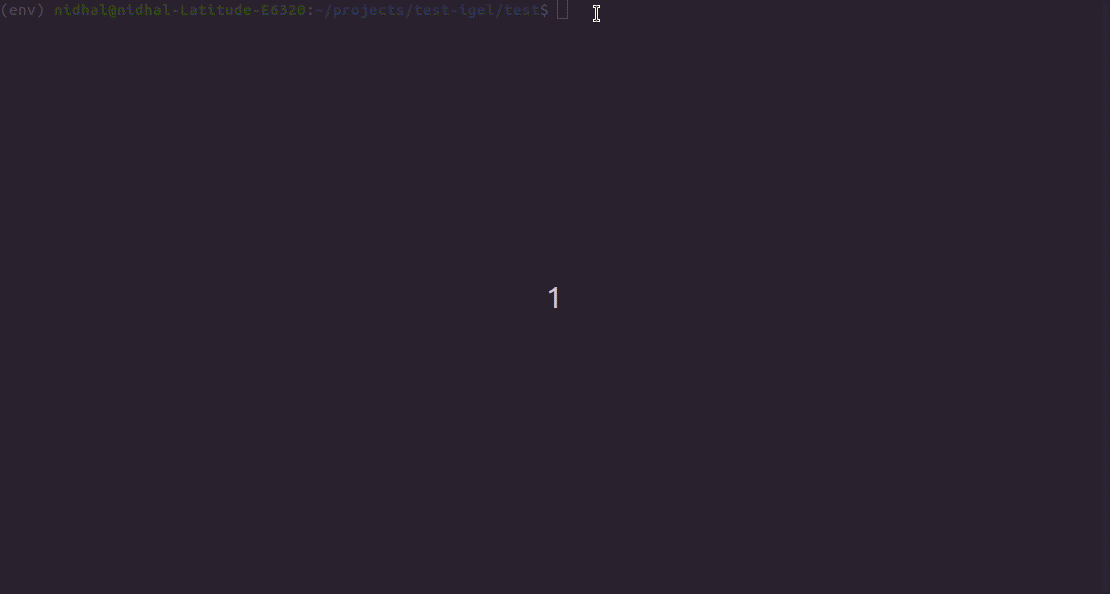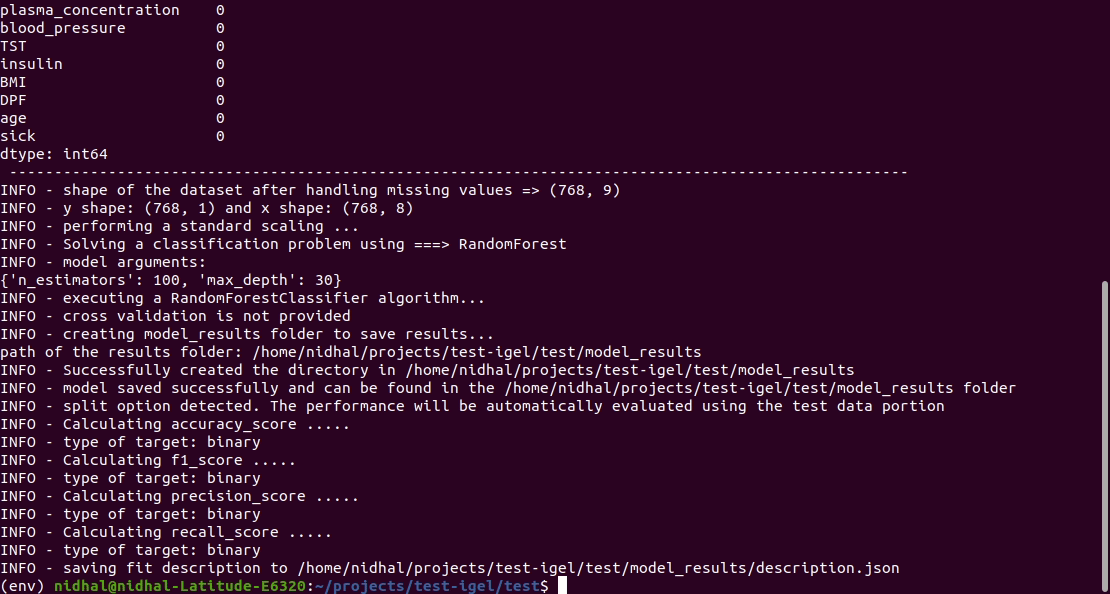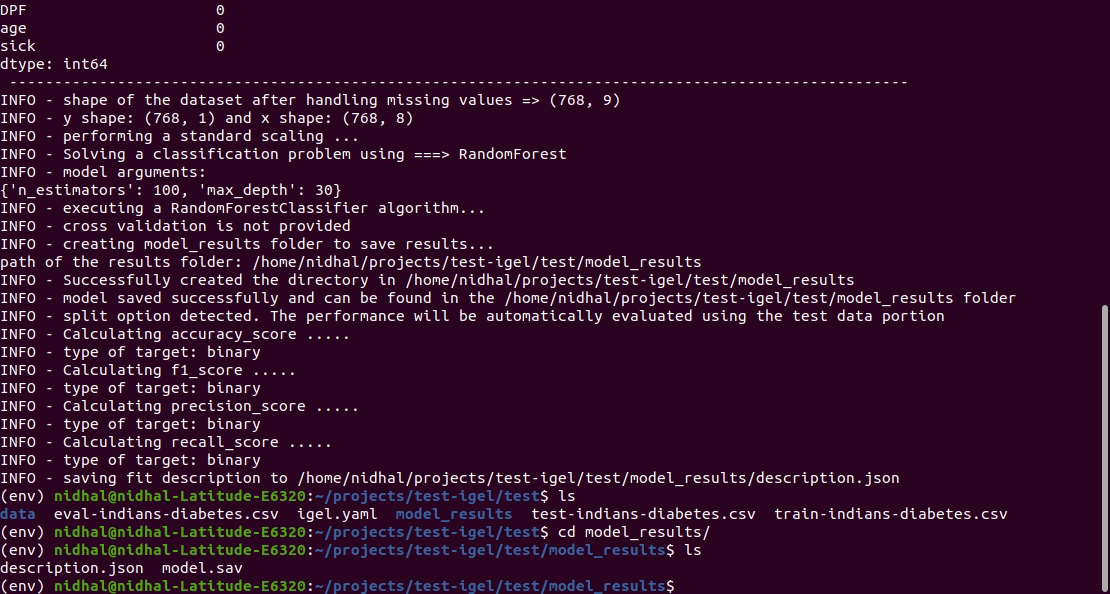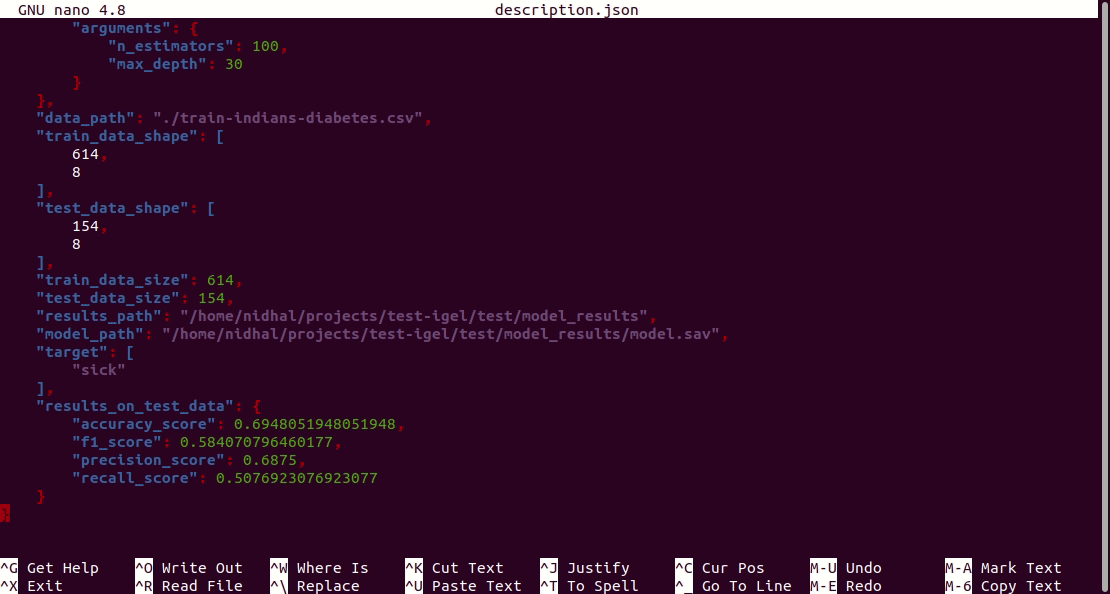
Furthermore, a description can be found in the description.json file inside the model_results folder.
"""
Luego puede evaluar el modelo entrenado/preinstalado:
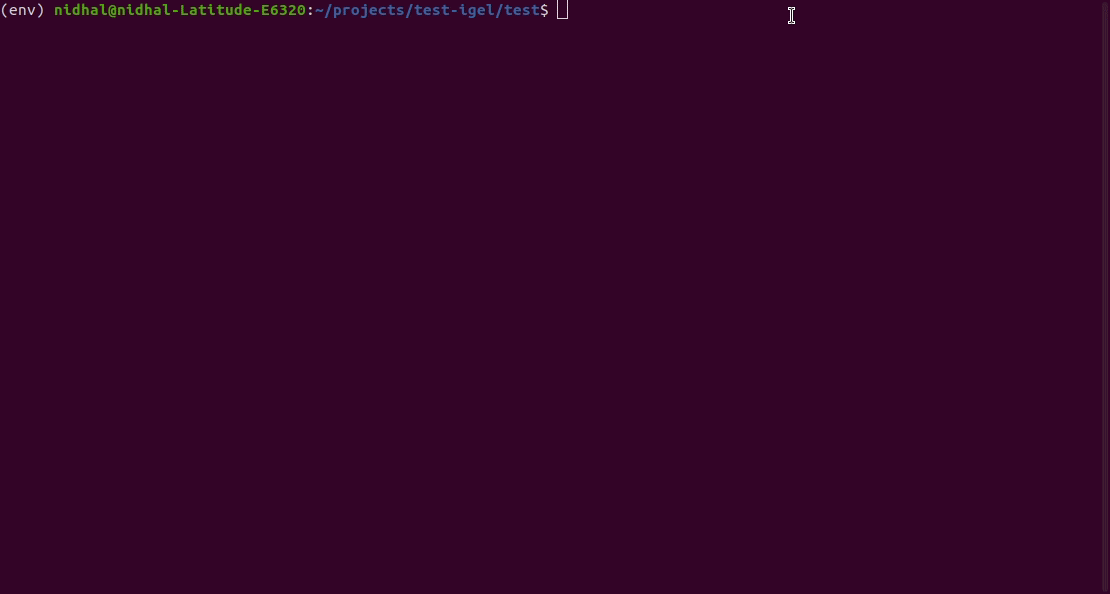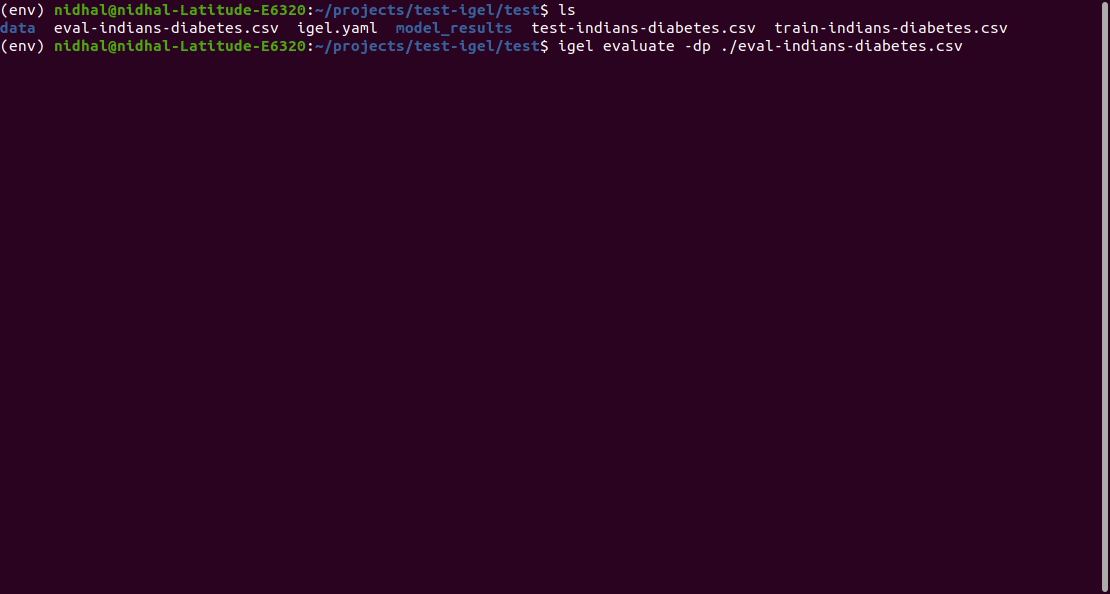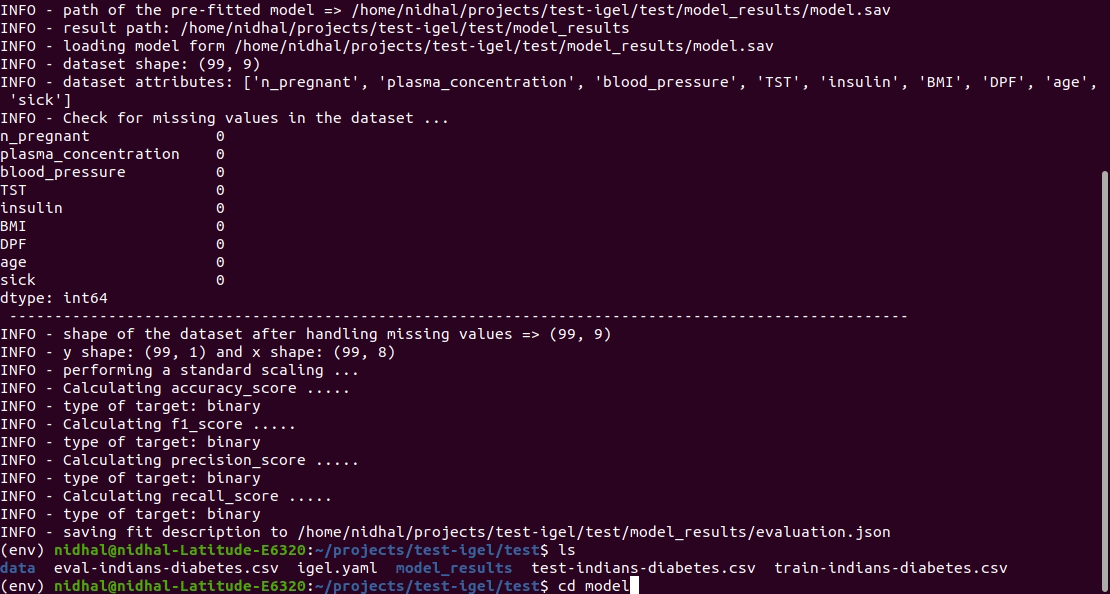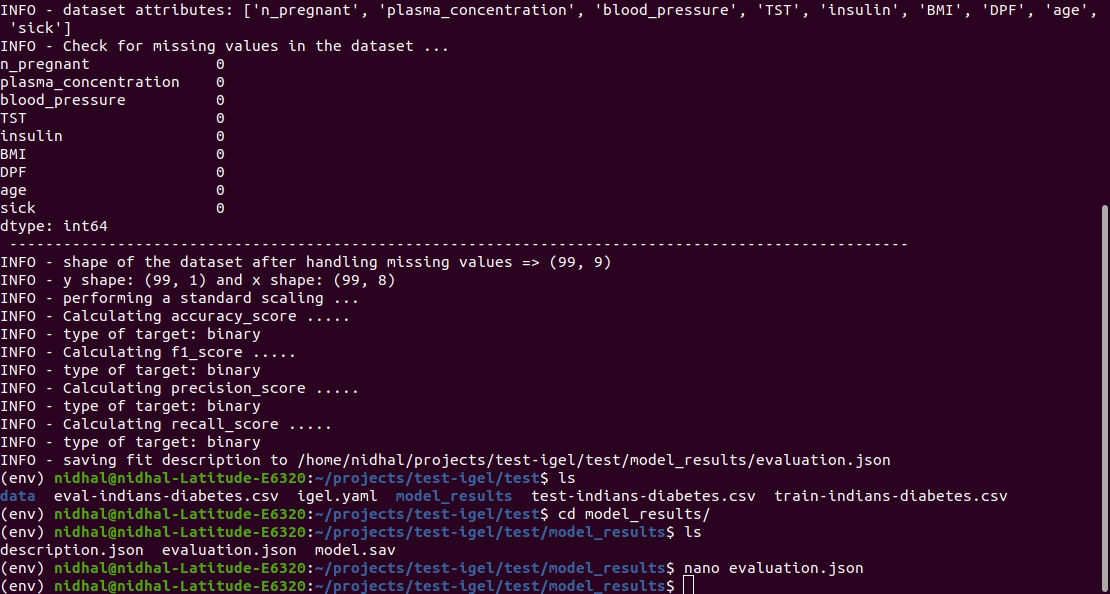
$ igel evaluate -dp ' path_to_your_evaluation_dataset.csv '
"""
This will automatically generate an evaluation.json file in the current directory, where all evaluation results are stored
"""
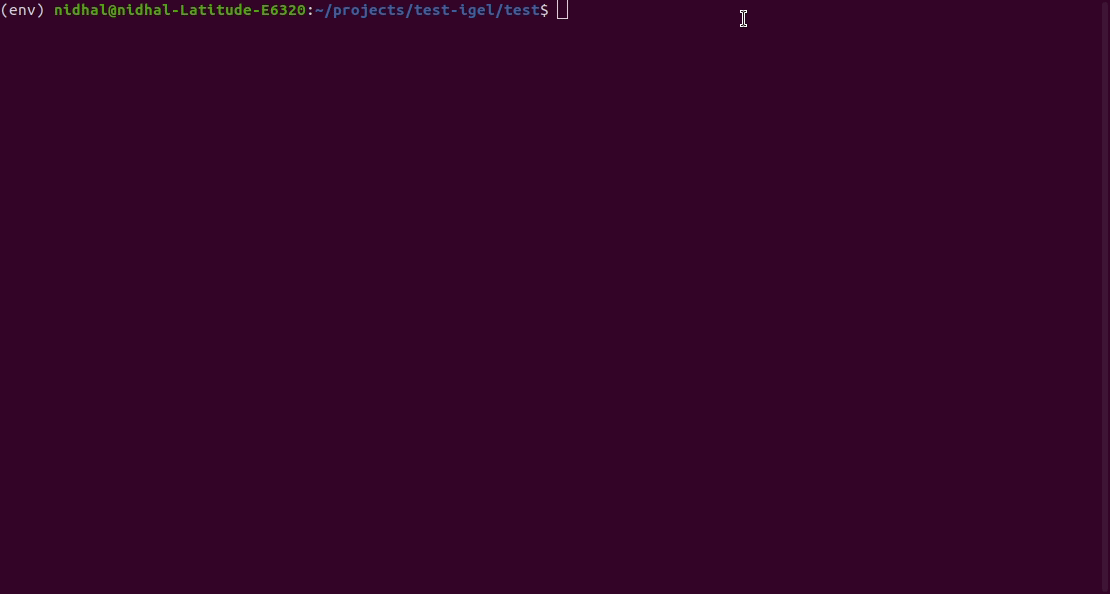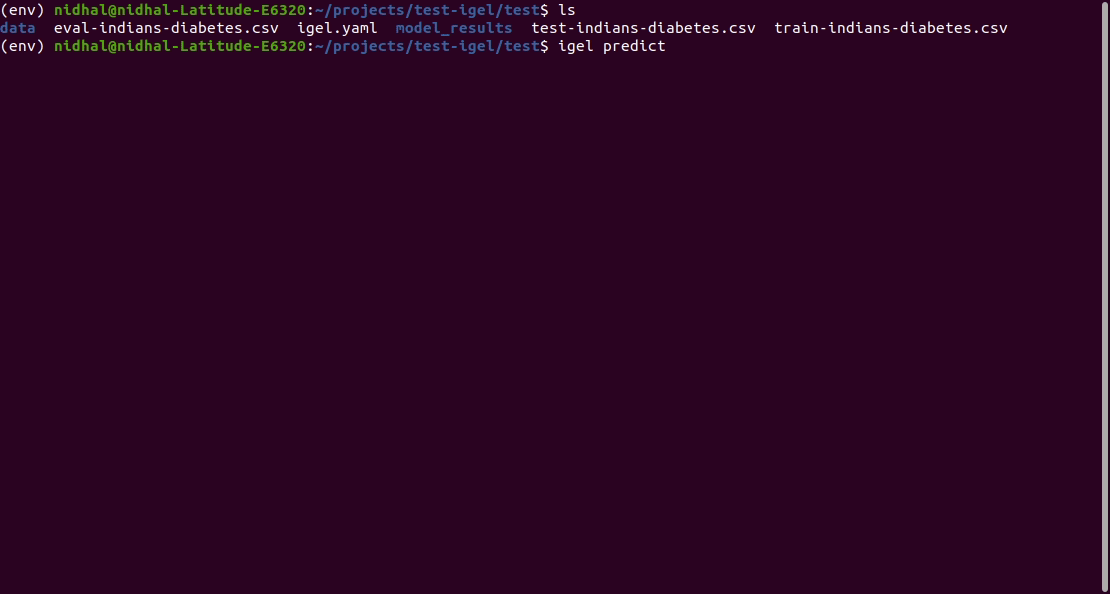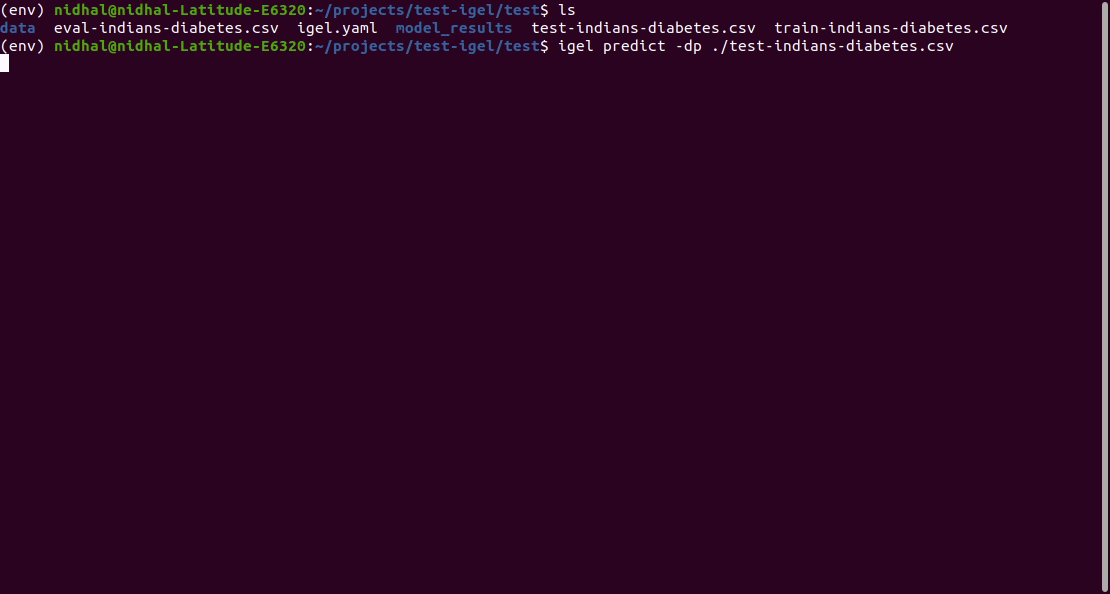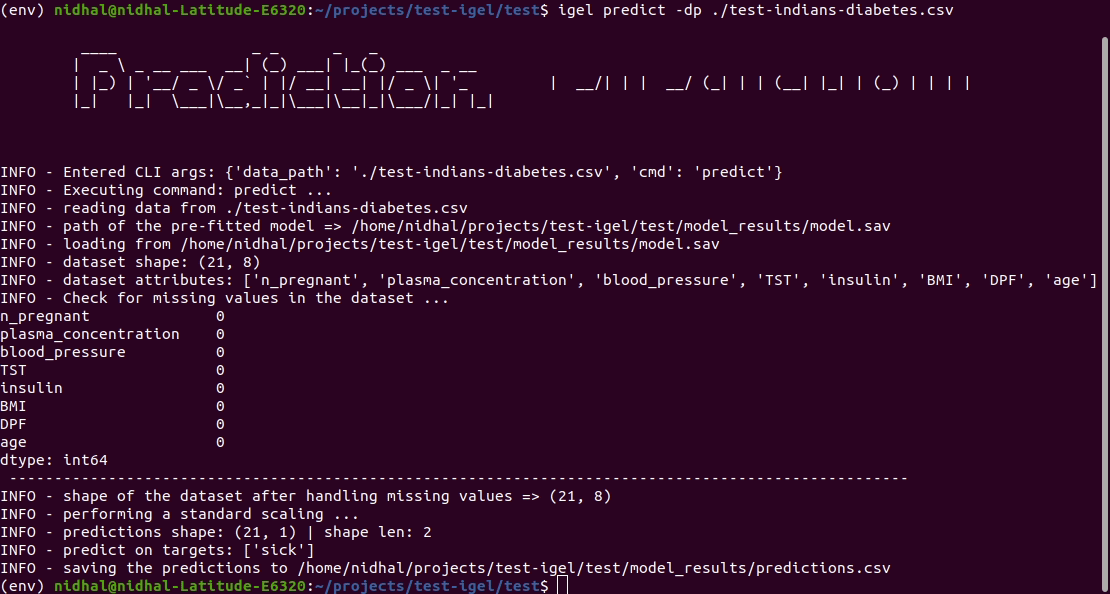
Finalmente, puede utilizar el modelo entrenado/preajustado para hacer predicciones si está satisfecho con los resultados de la evaluación:
$ igel predict -dp ' path_to_your_test_dataset.csv '
"""
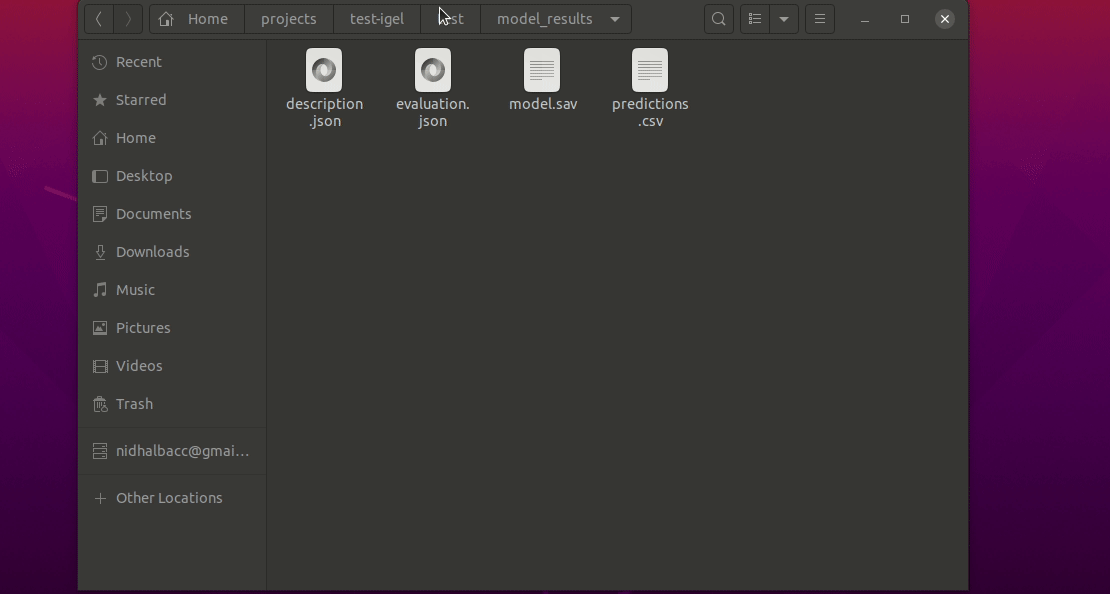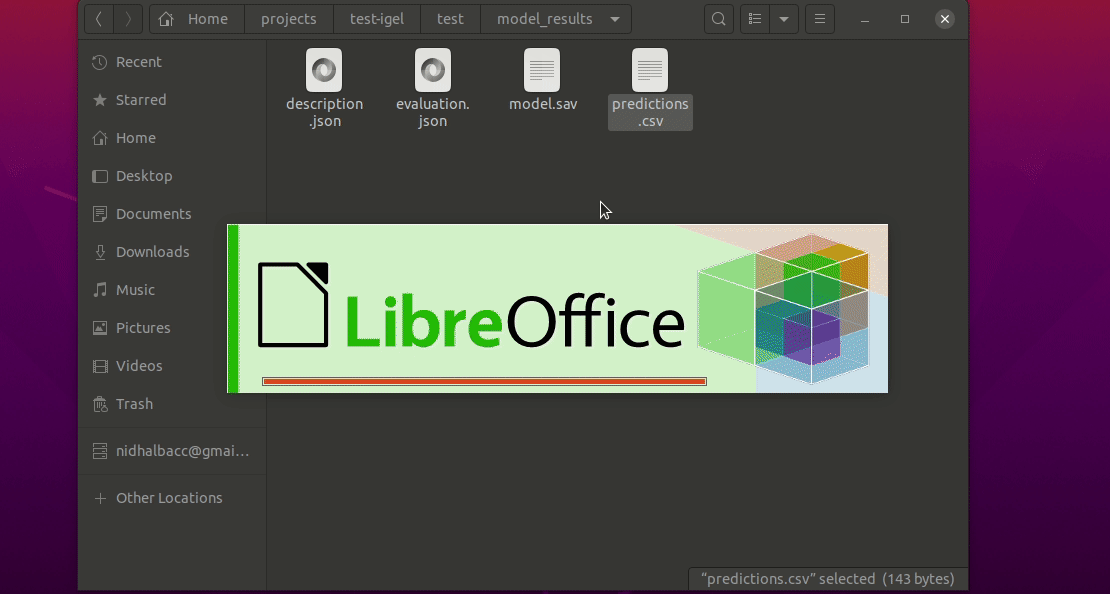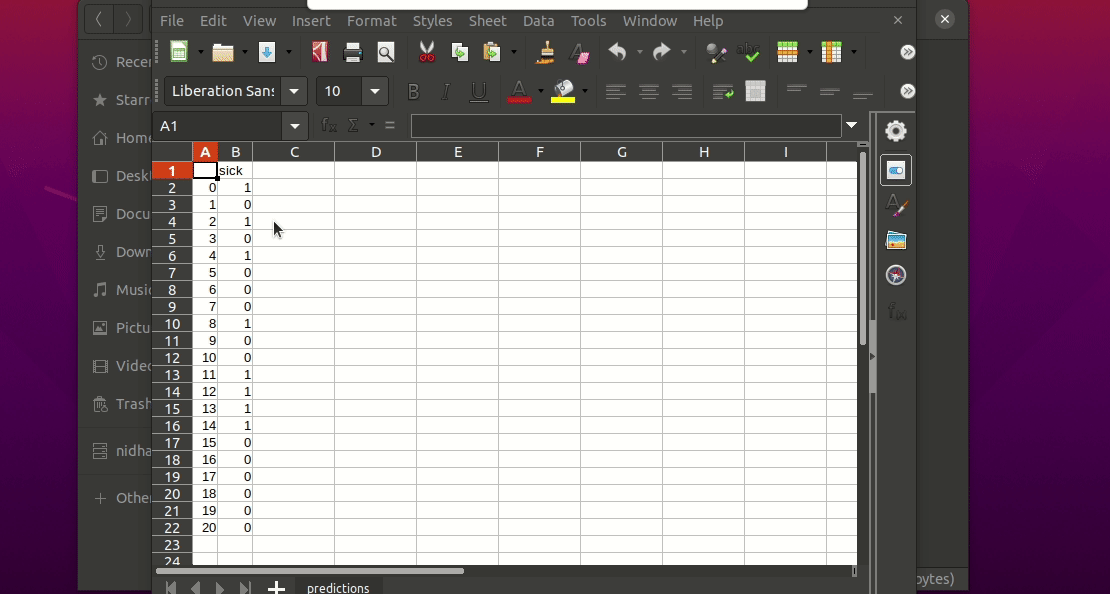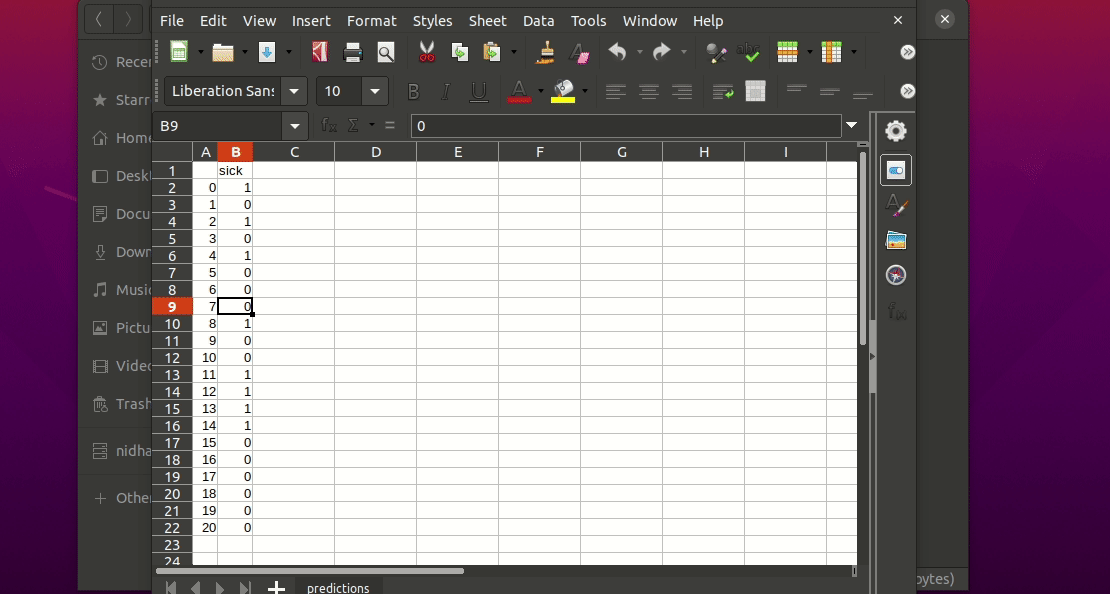
This will generate a predictions.csv file in your current directory, where all predictions are stored in a csv file
"""
Puedes combinar las fases de entrenamiento, evaluación y predicción usando un solo comando llamado experimento:
$ igel experiment -DP " path_to_train_data path_to_eval_data path_to_test_data " -yml " path_to_yaml_file "
"""
This will run fit using train_data, evaluate using eval_data and further generate predictions using the test_data
"""
Puede exportar el modelo de sklearn entrenado/preinstalado a ONNX:
$ igel export -dp " path_to_pre-fitted_sklearn_model "
"""
This will convert the sklearn model into ONNX
""" from igel import Igel
Igel ( cmd = "fit" , data_path = "path_to_your_dataset" , yaml_path = "path_to_your_yaml_file" )
"""
check the examples folder for more
"""El siguiente paso es utilizar su modelo en producción. Igel también te ayuda con esta tarea proporcionándote el comando de saque. Ejecutar el comando de servicio le indicará a igel que entregue su modelo. Precisamente, igel construirá automáticamente un servidor REST y servirá su modelo en un host y puerto específicos, que puede configurar pasándolos como opciones cli.
La forma más sencilla es ejecutar:
$ igel serve --model_results_dir " path_to_model_results_directory "Tenga en cuenta que igel necesita la opción --model_results_dir o brevemente -res_dir cli para cargar el modelo e iniciar el servidor. De forma predeterminada, igel entregará su modelo en localhost:8000 ; sin embargo, puede anular esto fácilmente proporcionando un host y un puerto cli opciones.
$ igel serve --model_results_dir " path_to_model_results_directory " --host " 127.0.0.1 " --port 8000Igel utiliza FastAPI para crear el servidor REST, que es un marco moderno de alto rendimiento y uvicorn para ejecutarlo internamente.
Este ejemplo se realizó utilizando un modelo previamente entrenado (creado ejecutando igel init --target enfermo -type Classification) y el conjunto de datos Indian Diabetes en ejemplos/datos. Los encabezados de las columnas en el CSV original son 'preg', 'plas', 'pres', 'skin', 'test', 'mass', 'pedi' y 'age'.
RIZO:
$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": 1, "plas": 180, "pres": 50, "skin": 12, "test": 1, "mass": 456, "pedi": 0.442, "age": 50} '
Outputs: {"prediction":[[0.0]]}$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": [1, 6, 10], "plas":[192, 52, 180], "pres": [40, 30, 50], "skin": [25, 35, 12], "test": [0, 1, 1], "mass": [456, 123, 155], "pedi": [0.442, 0.22, 0.19], "age": [50, 40, 29]} '
Outputs: {"prediction":[[1.0],[0.0],[0.0]]}Advertencias/Limitaciones:
Ejemplo de uso del cliente Python:
from python_client import IgelClient
# the client allows additional args with defaults:
# scheme="http", endpoint="predict", missing_values="mean"
client = IgelClient ( host = 'localhost' , port = 8080 )
# you can post other types of files compatible with what Igel data reading allows
client . post ( "my_batch_file_for_predicting.csv" )
Outputs : < Response 200 > : { "prediction" :[[ 1.0 ],[ 0.0 ],[ 0.0 ]]}El objetivo principal de igel es proporcionarle una forma de entrenar/adaptar, evaluar y utilizar modelos sin escribir código. En cambio, todo lo que necesita es proporcionar/describir lo que desea hacer en un archivo yaml simple.
Básicamente, proporciona una descripción o más bien configuraciones en el archivo yaml como pares clave-valor. Aquí hay una descripción general de todas las configuraciones admitidas (por ahora):
# dataset operations
dataset :
type : csv # [str] -> type of your dataset
read_data_options : # options you want to supply for reading your data (See the detailed overview about this in the next section)
sep : # [str] -> Delimiter to use.
delimiter : # [str] -> Alias for sep.
header : # [int, list of int] -> Row number(s) to use as the column names, and the start of the data.
names : # [list] -> List of column names to use
index_col : # [int, str, list of int, list of str, False] -> Column(s) to use as the row labels of the DataFrame,
usecols : # [list, callable] -> Return a subset of the columns
squeeze : # [bool] -> If the parsed data only contains one column then return a Series.
prefix : # [str] -> Prefix to add to column numbers when no header, e.g. ‘X’ for X0, X1, …
mangle_dupe_cols : # [bool] -> Duplicate columns will be specified as ‘X’, ‘X.1’, …’X.N’, rather than ‘X’…’X’. Passing in False will cause data to be overwritten if there are duplicate names in the columns.
dtype : # [Type name, dict maping column name to type] -> Data type for data or columns
engine : # [str] -> Parser engine to use. The C engine is faster while the python engine is currently more feature-complete.
converters : # [dict] -> Dict of functions for converting values in certain columns. Keys can either be integers or column labels.
true_values : # [list] -> Values to consider as True.
false_values : # [list] -> Values to consider as False.
skipinitialspace : # [bool] -> Skip spaces after delimiter.
skiprows : # [list-like] -> Line numbers to skip (0-indexed) or number of lines to skip (int) at the start of the file.
skipfooter : # [int] -> Number of lines at bottom of file to skip
nrows : # [int] -> Number of rows of file to read. Useful for reading pieces of large files.
na_values : # [scalar, str, list, dict] -> Additional strings to recognize as NA/NaN.
keep_default_na : # [bool] -> Whether or not to include the default NaN values when parsing the data.
na_filter : # [bool] -> Detect missing value markers (empty strings and the value of na_values). In data without any NAs, passing na_filter=False can improve the performance of reading a large file.
verbose : # [bool] -> Indicate number of NA values placed in non-numeric columns.
skip_blank_lines : # [bool] -> If True, skip over blank lines rather than interpreting as NaN values.
parse_dates : # [bool, list of int, list of str, list of lists, dict] -> try parsing the dates
infer_datetime_format : # [bool] -> If True and parse_dates is enabled, pandas will attempt to infer the format of the datetime strings in the columns, and if it can be inferred, switch to a faster method of parsing them.
keep_date_col : # [bool] -> If True and parse_dates specifies combining multiple columns then keep the original columns.
dayfirst : # [bool] -> DD/MM format dates, international and European format.
cache_dates : # [bool] -> If True, use a cache of unique, converted dates to apply the datetime conversion.
thousands : # [str] -> the thousands operator
decimal : # [str] -> Character to recognize as decimal point (e.g. use ‘,’ for European data).
lineterminator : # [str] -> Character to break file into lines.
escapechar : # [str] -> One-character string used to escape other characters.
comment : # [str] -> Indicates remainder of line should not be parsed. If found at the beginning of a line, the line will be ignored altogether. This parameter must be a single character.
encoding : # [str] -> Encoding to use for UTF when reading/writing (ex. ‘utf-8’).
dialect : # [str, csv.Dialect] -> If provided, this parameter will override values (default or not) for the following parameters: delimiter, doublequote, escapechar, skipinitialspace, quotechar, and quoting
delim_whitespace : # [bool] -> Specifies whether or not whitespace (e.g. ' ' or ' ') will be used as the sep
low_memory : # [bool] -> Internally process the file in chunks, resulting in lower memory use while parsing, but possibly mixed type inference.
memory_map : # [bool] -> If a filepath is provided for filepath_or_buffer, map the file object directly onto memory and access the data directly from there. Using this option can improve performance because there is no longer any I/O overhead.
random_numbers : # random numbers options in case you wanted to generate the same random numbers on each run
generate_reproducible : # [bool] -> set this to true to generate reproducible results
seed : # [int] -> the seed number is optional. A seed will be set up for you if you didn't provide any
split : # split options
test_size : 0.2 # [float] -> 0.2 means 20% for the test data, so 80% are automatically for training
shuffle : true # [bool] -> whether to shuffle the data before/while splitting
stratify : None # [list, None] -> If not None, data is split in a stratified fashion, using this as the class labels.
preprocess : # preprocessing options
missing_values : mean # [str] -> other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # [str] -> other possible values: [labelEncoding]
scale : # scaling options
method : standard # [str] -> standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # [str] -> scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification # [str] -> type of the problem you want to solve. | possible values: [regression, classification, clustering]
algorithm : NeuralNetwork # [str (notice the pascal case)] -> which algorithm you want to use. | type igel algorithms in the Terminal to know more
arguments : # model arguments: you can check the available arguments for each model by running igel help in your terminal
use_cv_estimator : false # [bool] -> if this is true, the CV class of the specific model will be used if it is supported
cross_validate :
cv : # [int] -> number of kfold (default 5)
n_jobs : # [signed int] -> The number of CPUs to use to do the computation (default None)
verbose : # [int] -> The verbosity level. (default 0)
hyperparameter_search :
method : grid_search # method you want to use: grid_search and random_search are supported
parameter_grid : # put your parameters grid here that you want to use, an example is provided below
param1 : [val1, val2]
param2 : [val1, val2]
arguments : # additional arguments you want to provide for the hyperparameter search
cv : 5 # number of folds
refit : true # whether to refit the model after the search
return_train_score : false # whether to return the train score
verbose : 0 # verbosity level
# target you want to predict
target : # list of strings: basically put here the column(s), you want to predict that exist in your csv dataset
- put the target you want to predict here
- you can assign many target if you are making a multioutput prediction Nota
igel usa pandas bajo el capó para leer y analizar los datos. Por lo tanto, puede encontrar estos parámetros opcionales de datos también en la documentación oficial de pandas.
A continuación se proporciona una descripción detallada de las configuraciones que puede proporcionar en el archivo yaml (o json). Tenga en cuenta que ciertamente no necesitará todos los valores de configuración para el conjunto de datos. Son opcionales. Generalmente, igel descubrirá cómo leer su conjunto de datos.
Sin embargo, puedes ayudarlo proporcionando campos adicionales usando esta sección read_data_options. Por ejemplo, en mi opinión, uno de los valores útiles es "sep", que define cómo se separan las columnas del conjunto de datos csv. Generalmente, los conjuntos de datos csv están separados por comas, que también es el valor predeterminado aquí. Sin embargo, en su caso puede estar separado por un punto y coma.
Por lo tanto, puede proporcionar esto en read_data_options. Simplemente agregue el sep: ";" en read_data_options.
| Parámetro | Tipo | Explicación |
|---|---|---|
| sep | cadena, predeterminado ',' | Delimitador a utilizar. Si sep es Ninguno, el motor C no puede detectar automáticamente el separador, pero el motor de análisis de Python sí, lo que significa que este último será utilizado y detectará automáticamente el separador mediante la herramienta de rastreo incorporada de Python, csv.Sniffer. Además, los separadores de más de 1 carácter y diferentes de 's+' se interpretarán como expresiones regulares y también forzarán el uso del motor de análisis Python. Tenga en cuenta que los delimitadores de expresiones regulares tienden a ignorar los datos citados. Ejemplo de expresión regular: 'rt'. |
| delimitador | predeterminado Ninguno | Alias de septiembre. |
| encabezamiento | int, lista de int, predeterminado 'inferir' | Número(s) de fila que se utilizarán como nombres de columna y el inicio de los datos. El comportamiento predeterminado es inferir los nombres de las columnas: si no se pasan nombres, el comportamiento es idéntico a encabezado=0 y los nombres de las columnas se infieren de la primera línea del archivo; si los nombres de las columnas se pasan explícitamente, entonces el comportamiento es idéntico a encabezado=Ninguno . Pase explícitamente header=0 para poder reemplazar los nombres existentes. El encabezado puede ser una lista de números enteros que especifican ubicaciones de filas para un índice múltiple en las columnas, por ejemplo, [0,1,3]. Se omitirán las filas intermedias que no estén especificadas (por ejemplo, se omite 2 en este ejemplo). Tenga en cuenta que este parámetro ignora las líneas comentadas y las líneas vacías si skip_blank_lines=True, por lo que header=0 denota la primera línea de datos en lugar de la primera línea del archivo. |
| nombres | tipo matriz, opcional | Lista de nombres de columnas a utilizar. Si el archivo contiene una fila de encabezado, debe pasar explícitamente header=0 para anular los nombres de las columnas. No se permiten duplicados en esta lista. |
| col_índice | int, str, secuencia de int/str, o Falso, predeterminado Ninguno | Columnas que se utilizarán como etiquetas de fila del DataFrame, ya sea como nombre de cadena o índice de columna. Si se proporciona una secuencia de int/str, se utiliza un MultiIndex. Nota: index_col=False se puede usar para forzar a pandas a no usar la primera columna como índice, por ejemplo, cuando tiene un archivo mal formado con delimitadores al final de cada línea. |
| usocols | tipo lista o invocable, opcional | Devuelve un subconjunto de las columnas. Si son tipo lista, todos los elementos deben ser posicionales (es decir, índices enteros en las columnas del documento) o cadenas que correspondan a nombres de columnas proporcionados por el usuario en los nombres o inferidos de las filas del encabezado del documento. Por ejemplo, un parámetro usecols similar a una lista válido sería [0, 1, 2] o ['foo', 'bar', 'baz']. Se ignora el orden de los elementos, por lo que usecols=[0, 1] es lo mismo que [1, 0]. Para crear una instancia de un DataFrame a partir de datos con el orden de los elementos conservado, use pd.read_csv(data, usecols=['foo', 'bar'])[['foo', 'bar']] para las columnas en ['foo', 'bar '] orden o pd.read_csv(data, usecols=['foo', 'bar'])[['bar', 'foo']] para ['bar', 'foo'] orden. Si se puede invocar, la función invocable se evaluará con respecto a los nombres de las columnas y devolverá nombres donde la función invocable se evalúe como Verdadero. Un ejemplo de un argumento invocable válido sería lambda x: x.upper() en ['AAA', 'BBB', 'DDD']. El uso de este parámetro da como resultado un tiempo de análisis mucho más rápido y un menor uso de memoria. |
| estrujar | booleano, predeterminado Falso | Si los datos analizados solo contienen una columna, devuelva una Serie. |
| prefijo | cadena, opcional | Prefijo para agregar a los números de columna cuando no hay encabezado, por ejemplo, 'X' para X0, X1,… |
| mangle_dupe_cols | booleano, valor predeterminado Verdadero | Las columnas duplicadas se especificarán como 'X', 'X.1',…'X.N', en lugar de 'X'…'X'. Pasar False hará que los datos se sobrescriban si hay nombres duplicados en las columnas. |
| tipo d | {'c', 'python'}, opcional | Motor analizador a utilizar. El motor C es más rápido, mientras que el motor Python actualmente tiene más funciones. |
| convertidores | dictado, opcional | Dictado de funciones para convertir valores en determinadas columnas. Las claves pueden ser números enteros o etiquetas de columna. |
| valores_verdaderos | lista, opcional | Valores a considerar como Verdaderos. |
| valores_falsos | lista, opcional | Valores a considerar como Falso. |
| omitir espacio inicial | booleano, predeterminado Falso | Saltar espacios después del delimitador. |
| salta | tipo lista, int o invocable, opcional | Números de línea para omitir (indexado 0) o número de líneas para omitir (int) al comienzo del archivo. Si se puede invocar, la función invocable se evaluará con respecto a los índices de fila y devolverá True si la fila debe omitirse y False en caso contrario. Un ejemplo de un argumento invocable válido sería lambda x: x en [0, 2]. |
| saltador | int, predeterminado 0 | Número de líneas en la parte inferior del archivo para omitir (no compatible con motor='c'). |
| filas | int, opcional | Número de filas de archivo a leer. Útil para leer fragmentos de archivos grandes. |
| valores_na | escalar, str, tipo lista o dict, opcional | Cadenas adicionales para reconocer como NA/NaN. Si se aprueba el dictado, valores NA específicos por columna. De forma predeterminada, los siguientes valores se interpretan como NaN: '', '#N/A', '#N/AN/A', '#NA', '-1.#IND', '-1.#QNAN', '-NaN', '-nan', '1.#IND', '1.#QNAN', '<NAN>', 'N/A', 'NA', 'NULL', 'NaN', 'n /a', 'nan', 'nulo'. |
| mantener_default_na | booleano, valor predeterminado Verdadero | Si se incluyen o no los valores NaN predeterminados al analizar los datos. Dependiendo de si se pasa na_values, el comportamiento es el siguiente: Si keep_default_na es True y se especifican na_values, na_values se agrega a los valores NaN predeterminados utilizados para el análisis. Si keep_default_na es True y no se especifican na_values, solo se utilizan los valores NaN predeterminados para el análisis. Si keep_default_na es False y se especifican na_values, solo se utilizan para el análisis los valores NaN especificados na_values. Si keep_default_na es False y no se especifican na_values, ninguna cadena se analizará como NaN. Tenga en cuenta que si na_filter se pasa como False, se ignorarán los parámetros keep_default_na y na_values. |
| na_filtro | booleano, valor predeterminado Verdadero | Detectar marcadores de valores faltantes (cadenas vacías y el valor de na_values). En datos sin NA, pasar na_filter=False puede mejorar el rendimiento de la lectura de un archivo grande. |
| verboso | booleano, predeterminado Falso | Indique el número de valores de NA colocados en columnas no numéricas. |
| saltar_líneas_en blanco | booleano, valor predeterminado Verdadero | Si es Verdadero, omita las líneas en blanco en lugar de interpretarlas como valores NaN. |
| analizar_fechas | bool o lista de int o nombres o lista de listas o dict, predeterminado Falso | El comportamiento es el siguiente: booleano. Si es Verdadero -> intente analizar el índice. lista de int o nombres. por ejemplo, si [1, 2, 3] -> intente analizar las columnas 1, 2, 3 cada una como una columna de fecha separada. lista de listas. por ejemplo, si [[1, 3]] -> combina las columnas 1 y 3 y analiza como una sola columna de fecha. dict, por ejemplo, {'foo': [1, 3]} -> analizar las columnas 1, 3 como fecha y llamar al resultado 'foo' Si una columna o índice no se puede representar como una matriz de fechas y horas, por ejemplo, debido a un valor no analizable o En una combinación de zonas horarias, la columna o el índice se devolverán sin modificaciones como un tipo de datos de objeto. |
| infer_datetime_format | booleano, predeterminado Falso | Si True y parse_dates están habilitados, pandas intentará inferir el formato de las cadenas de fecha y hora en las columnas y, si se puede inferir, cambiará a un método más rápido para analizarlas. En algunos casos, esto puede aumentar la velocidad de análisis entre 5 y 10 veces. |
| mantener_fecha_col | booleano, predeterminado Falso | Si True y parse_dates especifican la combinación de varias columnas, mantenga las columnas originales. |
| analizador_fecha | función, opcional | Función que se utilizará para convertir una secuencia de columnas de cadena en una matriz de instancias de fecha y hora. El valor predeterminado usa dateutil.parser.parser para realizar la conversión. Pandas intentará llamar a date_parser de tres maneras diferentes, avanzando a la siguiente si ocurre una excepción: 1) Pasar una o más matrices (según lo definido por parse_dates) como argumentos; 2) concatenar (por filas) los valores de cadena de las columnas definidas por parse_dates en una única matriz y pasarla; y 3) llamar a date_parser una vez para cada fila usando una o más cadenas (correspondientes a las columnas definidas por parse_dates) como argumentos. |
| primer día | booleano, predeterminado Falso | Fechas en formato DD/MM, formato internacional y europeo. |
| fechas_caché | booleano, valor predeterminado Verdadero | Si es Verdadero, utilice una caché de fechas convertidas únicas para aplicar la conversión de fecha y hora. Puede producir una aceleración significativa al analizar cadenas de fechas duplicadas, especialmente aquellas con diferencias de zona horaria. |
| miles | cadena, opcional | Separador de miles. |
| decimal | cadena, predeterminado '.' | Carácter a reconocer como punto decimal (por ejemplo, utilice ',' para datos europeos). |
| terminador de línea | str (longitud 1), opcional | Carácter para dividir el archivo en líneas. Sólo es válido con el analizador C. |
| escapechar | str (longitud 1), opcional | Cadena de un carácter utilizada para escapar de otros caracteres. |
| comentario | cadena, opcional | Indica que el resto de la línea no debe analizarse. Si se encuentra al principio de una línea, la línea se ignorará por completo. |
| codificación | cadena, opcional | Codificación que se utilizará para UTF al leer/escribir (por ejemplo, 'utf-8'). |
| dialecto | str o csv.Dialect, opcional | Si se proporciona, este parámetro anulará los valores (predeterminados o no) de los siguientes parámetros: delimitador, comillas dobles, caracteres de escape, skipinitialspace, quotechar y comillas. |
| memoria_baja | booleano, valor predeterminado Verdadero | Procese internamente el archivo en fragmentos, lo que da como resultado un menor uso de memoria durante el análisis, pero posiblemente una inferencia de tipos mixtos. Para garantizar que no haya tipos mixtos, establezca False o especifique el tipo con el parámetro dtype. Tenga en cuenta que todo el archivo se lee en un solo DataFrame independientemente, |
| mapa_memoria | booleano, predeterminado Falso | Asigne el objeto de archivo directamente a la memoria y acceda a los datos directamente desde allí. El uso de esta opción puede mejorar el rendimiento porque ya no hay sobrecarga de E/S. |
En esta sección se proporciona una solución completa de extremo a extremo para demostrar las capacidades de igel . Como se explicó anteriormente, debe crear un archivo de configuración yaml. A continuación se muestra un ejemplo completo para predecir si alguien tiene diabetes o no utilizando el algoritmo del árbol de decisiones . El conjunto de datos se puede encontrar en la carpeta de ejemplos.
model :
type : classification
algorithm : DecisionTree
target :
- sick $ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_fileEso es todo, igel ahora ajustará el modelo por usted y lo guardará en una carpeta model_results en su directorio actual.
Evaluar el modelo preinstalado. Igel cargará el modelo preinstalado desde el directorio model_results y lo evaluará por usted. Sólo necesita ejecutar el comando de evaluación y proporcionar la ruta a sus datos de evaluación.
$ igel evaluate -dp path_to_the_evaluation_dataset¡Eso es todo! Igel evaluará el modelo y almacenará estadísticas/resultados en un archivo de evaluación.json dentro de la carpeta model_results.
Utilice el modelo predefinido para predecir datos nuevos. Esto lo hace igel automáticamente, solo necesita proporcionar la ruta a sus datos en los que desea utilizar la predicción.
$ igel predict -dp path_to_the_new_dataset¡Eso es todo! Igel utilizará el modelo predefinido para hacer predicciones y guardarlo en un archivo predictions.csv dentro de la carpeta model_results.
También puede llevar a cabo algunos métodos de preprocesamiento u otras operaciones proporcionándolos en el archivo yaml. A continuación se muestra un ejemplo en el que los datos se dividen en un 80 % para entrenamiento y un 20 % para validación/prueba. Además, los datos se mezclan mientras se dividen.
Además, los datos se preprocesan reemplazando los valores faltantes con la media (también puede usar la mediana, la moda, etc.). revisa este enlace para más información
# dataset operations
dataset :
split :
test_size : 0.2
shuffle : True
stratify : default
preprocess : # preprocessing options
missing_values : mean # other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # other possible values: [labelEncoding]
scale : # scaling options
method : standard # standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification
algorithm : RandomForest
arguments :
# notice that this is the available args for the random forest model. check different available args for all supported models by running igel help
n_estimators : 100
max_depth : 20
# target you want to predict
target :
- sickLuego, puede ajustar el modelo ejecutando el comando igel como se muestra en los otros ejemplos.
$ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_filePara evaluación
$ igel evaluate -dp path_to_the_evaluation_datasetPara la producción
$ igel predict -dp path_to_the_new_dataset En la carpeta de ejemplos del repositorio, encontrará una carpeta de datos, donde se almacenan los famosos conjuntos de datos de diabetes india, iris y linnerud (de sklearn). Además, hay ejemplos completos dentro de cada carpeta, donde hay scripts y archivos yaml que lo ayudarán a comenzar.
La carpeta indian-diabetes-example contiene dos ejemplos que le ayudarán a empezar:
La carpeta iris-example contiene un ejemplo de regresión logística , donde se realiza algún preprocesamiento (una codificación en caliente) en la columna de destino para mostrarle más las capacidades de igel.
Además, el ejemplo de salida múltiple contiene un ejemplo de regresión de salida múltiple . Finalmente, el ejemplo cv contiene un ejemplo que utiliza el clasificador Ridge mediante validación cruzada.
También puede encontrar ejemplos de validación cruzada y búsqueda de hiperparámetros en la carpeta.
Te sugiero que juegues con los ejemplos y igel cli. Sin embargo, también puede ejecutar directamente fit.py, evalua.py y predict.py si lo desea.
Primero, cree o modifique un conjunto de datos de imágenes que estén categorizadas en subcarpetas según la etiqueta/clase de la imagen. Por ejemplo, si tiene imágenes de perros y gatos, necesitará 2 subcarpetas:
Suponiendo que estas dos subcarpetas están contenidas en una carpeta principal llamada imágenes, simplemente envíe datos a igel:
$ igel auto-train -dp ./images --task ImageClassificationIgel se encargará de todo, desde el preprocesamiento de los datos hasta la optimización de los hiperparámetros. Al final, el mejor modelo se almacenará en el directorio de trabajo actual.
Primero, cree o modifique un conjunto de datos de texto que esté categorizado en subcarpetas según la etiqueta/clase de texto. Por ejemplo, si tiene un conjunto de datos de texto de comentarios positivos y negativos, necesitará 2 subcarpetas:
Suponiendo que estas dos subcarpetas están contenidas en una carpeta principal llamada textos, simplemente envíe datos a igel:
$ igel auto-train -dp ./texts --task TextClassificationIgel se encargará de todo, desde el preprocesamiento de los datos hasta la optimización de los hiperparámetros. Al final, el mejor modelo se almacenará en el directorio de trabajo actual.
También puede ejecutar la interfaz de usuario de igel si no está familiarizado con el terminal. Simplemente instale igel en su máquina como se mencionó anteriormente. Luego ejecute este único comando en su terminal
$ igel guiEsto abrirá la interfaz gráfica de usuario, que es muy sencilla de usar. Consulte ejemplos de cómo se ve la interfaz gráfica de usuario y cómo usarla aquí: https://github.com/nidhaloff/igel-ui
Puedes extraer la imagen primero desde Docker Hub.
$ docker pull nidhaloff/igelEntonces úsalo:
$ docker run -it --rm -v $( pwd ) :/data nidhaloff/igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv 'Puedes ejecutar igel dentro de la ventana acoplable construyendo primero la imagen:
$ docker build -t igel .Y luego ejecútelo y adjunte su directorio actual (no es necesario que sea el directorio igel) como /data (el directorio de trabajo) dentro del contenedor:
$ docker run -it --rm -v $( pwd ) :/data igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv ' Si tiene algún problema, no dude en abrir un problema. Además, puede ponerse en contacto con el autor para obtener más información o preguntas.
¿Te gusta igel? Siempre puedes ayudar al desarrollo de este proyecto:
¿Crees que este proyecto es útil y quieres aportar nuevas ideas, nuevas funciones, correcciones de errores y ampliar los documentos?
Las contribuciones siempre son bienvenidas. Asegúrate de leer las pautas primero.
licencia MIT
Copyright (c) 2020-presente, Nidhal Baccouri