¡Se cargan todos los datos bien procesados y listos para DL de 109 sujetos de EEGMMIDB!
Este tutorial contiene códigos de cuadernos de Python y Jupyter implementables y conjuntos de datos de referencia para aprender a reconocer señales cerebrales basadas en modelos de aprendizaje profundo. Este tutorial asocia nuestra encuesta sobre señales cerebrales no invasivas basadas en DL y el libro sobre BCI basado en DL: representaciones, algoritmos y aplicaciones.
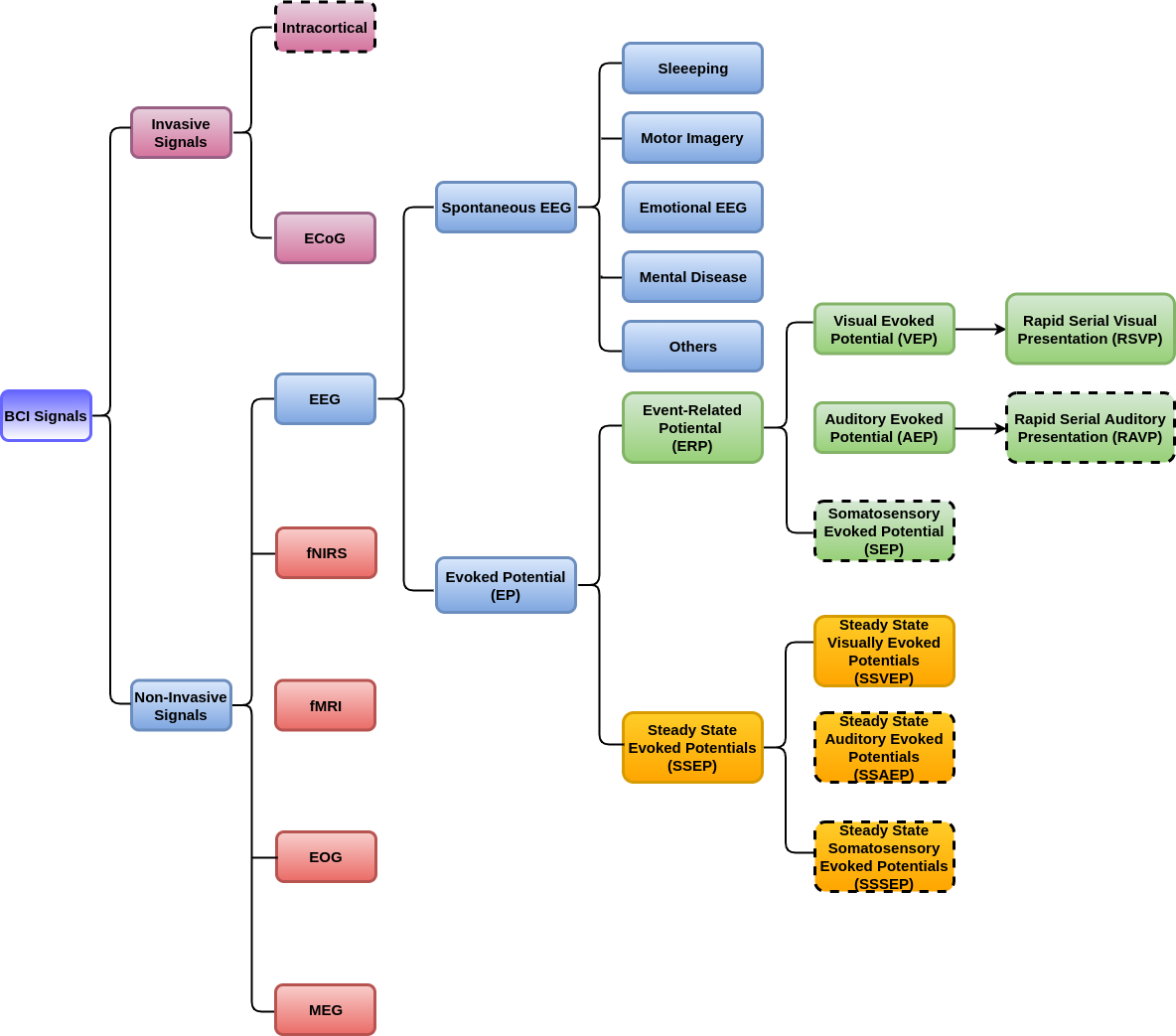
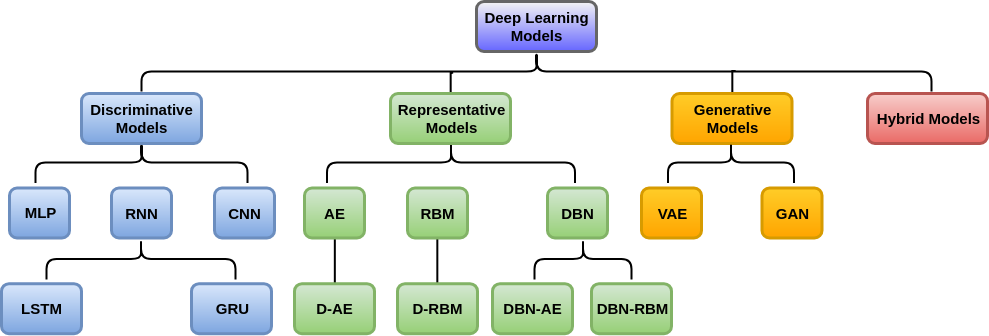
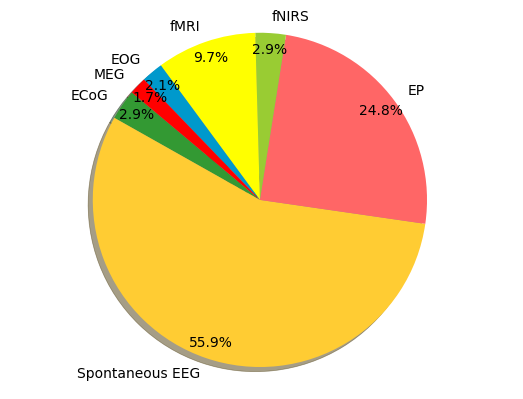
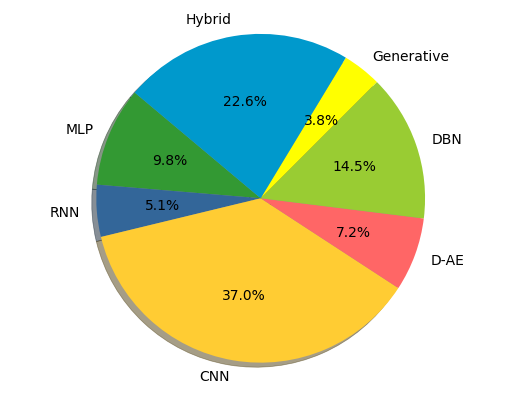
| Distribución de señales. | Distribución en modelos DL. |
|---|---|
 |  |
Se ha prestado especial atención a los estudios más recientes sobre aprendizaje profundo para la investigación BCI basada en EEG en términos de algoritmos. Específicamente, presentamos una serie de marcos y algoritmos avanzados de aprendizaje profundo destinados a varios problemas importantes en BCI, incluido el aprendizaje robusto de representación de señales cerebrales, la clasificación entre escenarios y la clasificación semisupervisada.
Además, se proponen varios prototipos novedosos de sistemas BCI basados en aprendizaje profundo que arrojan luz sobre aplicaciones del mundo real, como autenticación, reconstrucción visual, interpretación del lenguaje y diagnóstico de trastornos neurológicos. Estas aplicaciones pueden beneficiar enormemente tanto a las personas sanas como a las personas con discapacidades en la vida real.
La recopilación de señales cerebrales es costosa tanto financiera como temporalmente. Exploramos exhaustivamente los conjuntos de datos de referencia aplicables a la investigación de señales de lluvia y proporcionamos 31 conjuntos de datos públicos con enlaces de descarga que cubren la mayoría de los tipos de señales cerebrales.
| Señales cerebrales | Conjunto de datos | #-Sujeto | #-Clases | Frecuencia de muestreo (Hz) | #-Canales | Enlace de descarga |
|---|---|---|---|---|---|---|
| FM EcoG | BCI-C IV, conjunto de datos IV | 3 | 5 | 1000 | 48 -- 64 | Enlace |
| MI EcoG | BCI-C III conjunto de datos yo | 1 | 2 | 1000 | 64 | Enlace |
| EEG durmiente | Telemetría Sleep-EDF | 22 | 6 | 100 | 2 EEG, 1 EOG, 1 EMG | Enlace |
| EEG dormido | Sleep-EDF: casete | 78 | 6 | 100, 1 | 2 EEG (100 Hz), 1 EOG (100 Hz), 1 EMG (1Hz) | Enlace |
| EEG dormido | MASA-1 | 53 | 5 | 256 | 17/19 EEG, 2 EOG, 5 EMG | Enlace |
| EEG dormido | MASA-2 | 19 | 6 | 256 | 19 EEG, 4 EOG, 1EMG | Enlace |
| EEG dormido | MASA-3 | 62 | 5 | 256 | 20 EEG, 2 EOG, 3 EMG | Enlace |
| EEG dormido | MASA-4 | 40 | 6 | 256 | 4 EEG, 4 EOG, 1 EMG | Enlace |
| EEG durmiente | MASA-5 | 26 | 6 | 256 | 20 EEG, 2 EOG, 3 EMG | Enlace |
| EEG durmiente | SHHS | 5804 | N / A | 125, 50 | 2 EEG (125 Hz), 1 EOG (50 Hz), 1 EMG (125 Hz) | Enlace |
| EEG de convulsiones | CHB-MIT | 22 | 2 | 256 | 18 | Enlace |
| EEG de convulsiones | Eh | 315 | 2 | 200 | 19 | Enlace |
| EEG | EEGMMI | 109 | 4 | 160 | 64 | Enlace |
| EEG | BCI-C II, conjunto de datos III | 1 | 2 | 128 | 3 | Enlace |
| EEG | BCI-C III, conjunto de datos IIIa | 3 | 4 | 250 | 60 | Enlace |
| EEG | BCI-C III, Conjunto de datos III b | 3 | 2 | 125 | 2 | Enlace |
| MI EEG | BCI-C III, Conjunto de datos IV a | 5 | 2 | 1000 | 118 | Enlace |
| MI EEG | BCI-C III, conjunto de datos IV b | 1 | 2 | 1001 | 119 | Enlace |
| MI EEG | BCI-C III, conjunto de datos IV c | 1 | 2 | 1002 | 120 | Enlace |
| MI EEG | BCI-C IV, Conjunto de datos I | 7 | 2 | 1000 | 64 | Enlace |
| MI EEG | ICC-C IV, Conjunto de datos IIa | 9 | 4 | 250 | 22 EEG, 3 EOG | Enlace |
| EEG | BCI-C IV, Conjunto de datos II b | 9 | 2 | 250 | 3 EEG, 3 EOG | Enlace |
| EEG emocional | AMIGOS | 40 | 4 | 128 | 14 | Enlace |
| EEG emocional | SEMILLA | 15 | 3 | 200 | 62 | Enlace |
| EEG emocional | DEAP | 32 | 4 | 512 | 32 | Enlace |
| Otros EEG | Abrir MIIR | 10 | 12 | 512 | 64 | Enlace |
| PEV | BCI-C II, Conjunto de datos II b | 1 | 36 | 240 | 64 | Enlace |
| PEV | BCI-C III, Conjunto de datos II | 2 | 26 | 240 | 64 | Enlace |
| resonancia magnética funcional | ADNI | 202 | 3 | N / A | N / A | Enlace |
| resonancia magnética funcional | MOCOSOS | 65 | 4 | N / A | N / A | Enlace |
| MEG | BCI-C IV, Conjunto de datos III | 2 | 4 | 400 | 10 | Enlace |
Para permitir que los lectores tengan un acceso rápido al conjunto de datos y puedan jugar con él, proporcionamos el conjunto de datos bien procesado y listo para usar de EEG Motor Movement/Imagery Database (EEGMMIDB). Este conjunto de datos contiene 109 sujetos, mientras que las señales de EEG se registran en 64 canales con una frecuencia de muestreo de 160 Hz. Después de nuestra limpieza y clasificación, cada archivo npy representa un tema: la forma de los datos de cada archivo npy es [N, 65], las primeras 64 columnas corresponden a 64 características del canal, la última columna denota la etiqueta de clase. El N varía para diferentes sujetos, pero N debería ser 259520 o 255680. Ésta es la diferencia inherente en el conjunto de datos original.
En nuestros archivos de tutorial, aprenderá la línea y el flujo de trabajo del sistema BCI, incluida la adquisición de datos, el preprocesamiento, la extracción de características (opcional), la clasificación y la evaluación. Presentamos las referencias necesarias y los códigos procesables de los modelos de aprendizaje profundo más típicos (GRU, LSTM, CNN, GNN) aprovechando las dependencias temporales, espaciales y topográficas. También proporcionamos códigos Python que son muy útiles. Por ejemplo, para comprobar el rendimiento de la clasificación EEG de CNN, ejecute el siguiente código:
python 4-2_CNN.py
Para los principiantes de PyTorch, recomendamos encarecidamente los tutoriales de PyTorch de Morvan Zhou.
Para los algoritmos y aplicaciones presentados en el libro, proporcionamos los códigos de implementación necesarios (versión TensorFlow):
Si considera que nuestra investigación es útil para su investigación, considere citar nuestra encuesta o libro:
@article{zhang2020survey,
title={A survey on deep learning-based non-invasive brain signals: recent advances and new frontiers},
author={Zhang, Xiang and Yao, Lina and Wang, Xianzhi and Monaghan, Jessica JM and Mcalpine, David and Zhang, Yu},
journal={Journal of Neural Engineering},
year={2020},
publisher={IOP Publishing}
}
@book{zhang2021deep,
title={Deep Learning for EEG-based Brain-Computer Interface: Representations, Algorithms and Applications},
author={Zhang, Xiang and Yao, Lina},
year={2021},
publisher={World Scientific Publishing}
}
Los códigos del tutorial están probados para funcionar en Python 3.7.
Se requieren versiones recientes de Pytorch, torch-geometric, numpy y scipy. Todos los paquetes básicos requeridos se pueden instalar usando el siguiente comando: ''' pip install -r requisitos.txt ''' Nota: Para toch-geometric y las dependencias relacionadas (por ejemplo, cluster, scatter, sparse), la versión superior puede Funciona pero aún no se ha probado.
Envíe cualquier pregunta que pueda tener sobre el código y/o el algoritmo a [email protected].
Este tutorial tiene la licencia MIT.