Este repositorio contiene código y datos para realizar una clasificación dermoscópica acomodada con la deficiencia de visión de color con GPT-4V.
Leer el periódico
Encuentre la respuesta detallada de los datos del formulario de modelo GPT Mendeley
numpy
Pilotear
daltonlens
json
opadai
base64
pandas
. ├── data # Contains subdirectories for processed data │ ├── all │ ├── all_resized │ ├── all_resized_brettel_protan_1 │ ├── all_resized_brettel_deutan_1 │ ├── all_resized_brettel_tritan_1 │ ├── bn │ ├── bn_resized │ ├── bn_resized_label │ ├── bn_resized_label_brettel_protan_1 │ ├── bn_resized_label_brettel_deutan_1 │ ├── bn_resized_label_brettel_tritan_1 │ ├── mm │ ├── mm_resized │ ├── mm_resized_label │ ├── mm_resized_label_brettel_protan_1 │ ├── mm_resized_label_brettel_deutan_1 │ ├── mm_resized_label_brettel_tritan_1 │ └── selected_images.xlsx # Image names ├── RAW # Contains raw data downloaded from ISCI Archive ├── result # Results from running API_calling will be stored here │ ├── 2_shot_brettel_protan_1 │ │ ├──rep 1 │ │ └── ... │ ├── 2_shot_brettel_deutan_1 │ └── ... ├── CVD_classification_GPT.py # Call OpenAI API for classification ├── CVD_convertor.py # Convert original image to CVD simulated image ├── data_labeling.py # Add label to the image for reference ├── data_resizing.py # Resize the data with the original aspect ratio ├── data_selection.py # Select data from RAW └── README.md
Todas las imágenes dermoscópicas se descargan del archivo ISIC.
Este proyecto es solo para fines de investigación académica. El código en este repositorio se publica bajo la licencia MIT. Si usa los datos proporcionados, cite el archivo ISIC.s
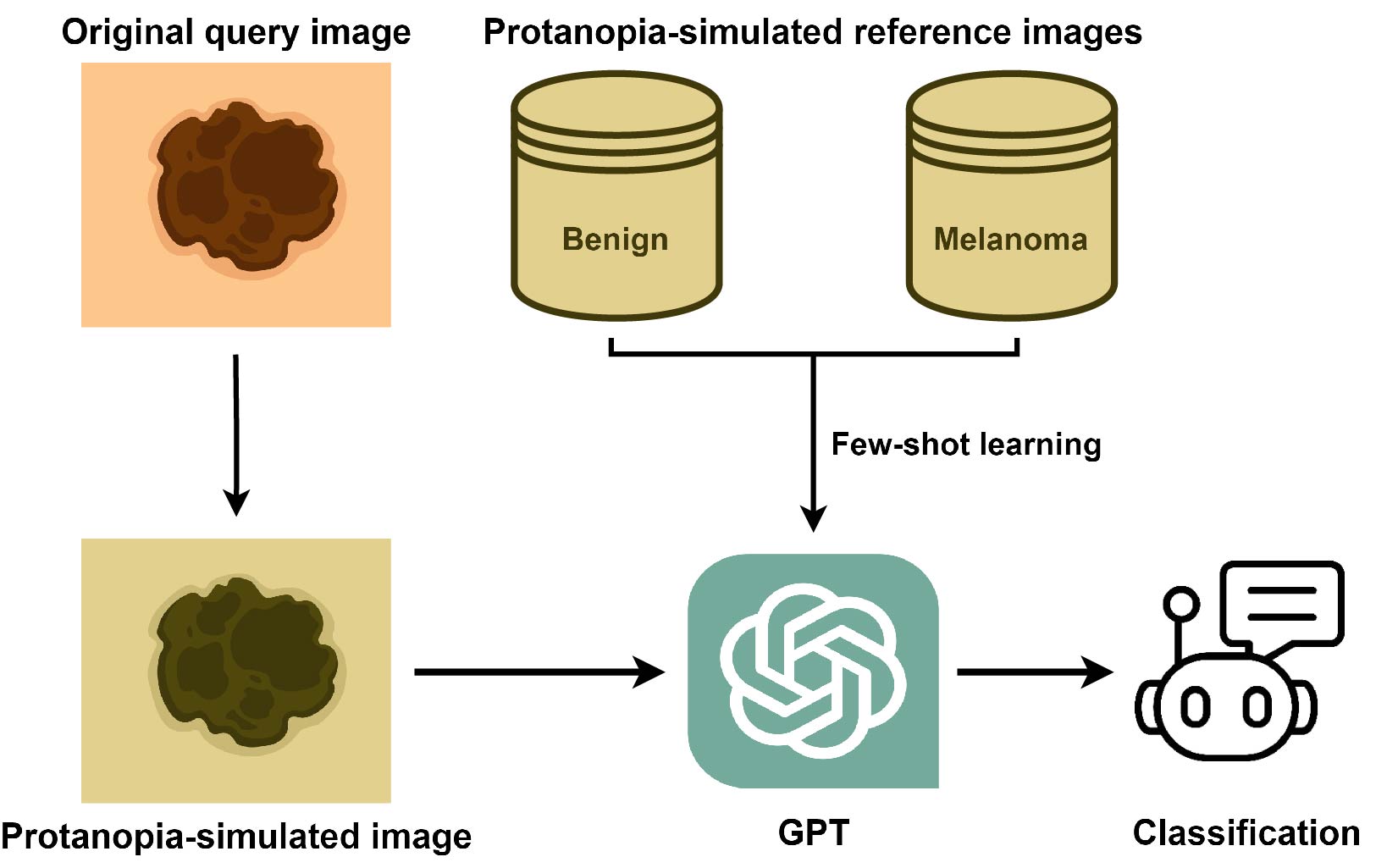
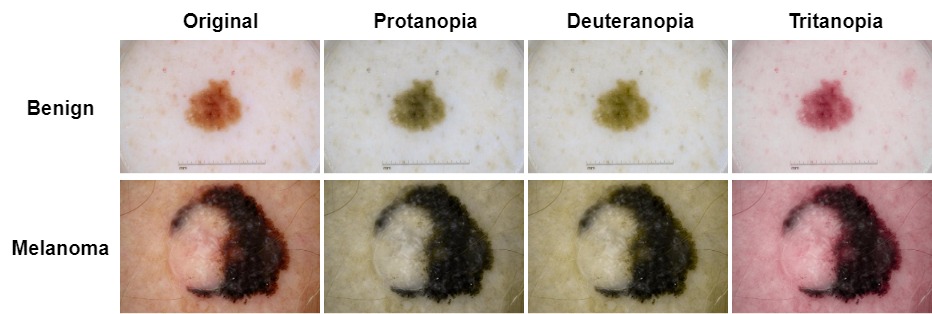
Para la simulación CVD, elegimos el paquete Daltonlens-Python. Ha cubierto una variedad de los algoritmos disponibles actualmente de simulación Colorblindness. También vale la pena leer el blog del autor.
Hemos seleccionado el Brettel et al. 1997 como el método de simulación CVD y establece la gravedad en 1, que se puede ajustar con el código proporcionado según sea necesario.
Ejemplo benigno: ISIC_0012656
Ejemplo de melanoma: ISIC_0046725
Descargue los datos sin procesar del archivo ISIC
Seleccionar datos:
Ejecute python data_selection.py -[Opciones]
Datos de proceso:
Ejecute python data_resizeing.py -[Opciones]
Ejecute python data_labeling.py -[Opciones]
Convertir datos:
Ejecute python cvd_conververor.py -[opciones]
Llame a la API:
Ejecute Python CVD_Classification_gpt.py -[Opciones]
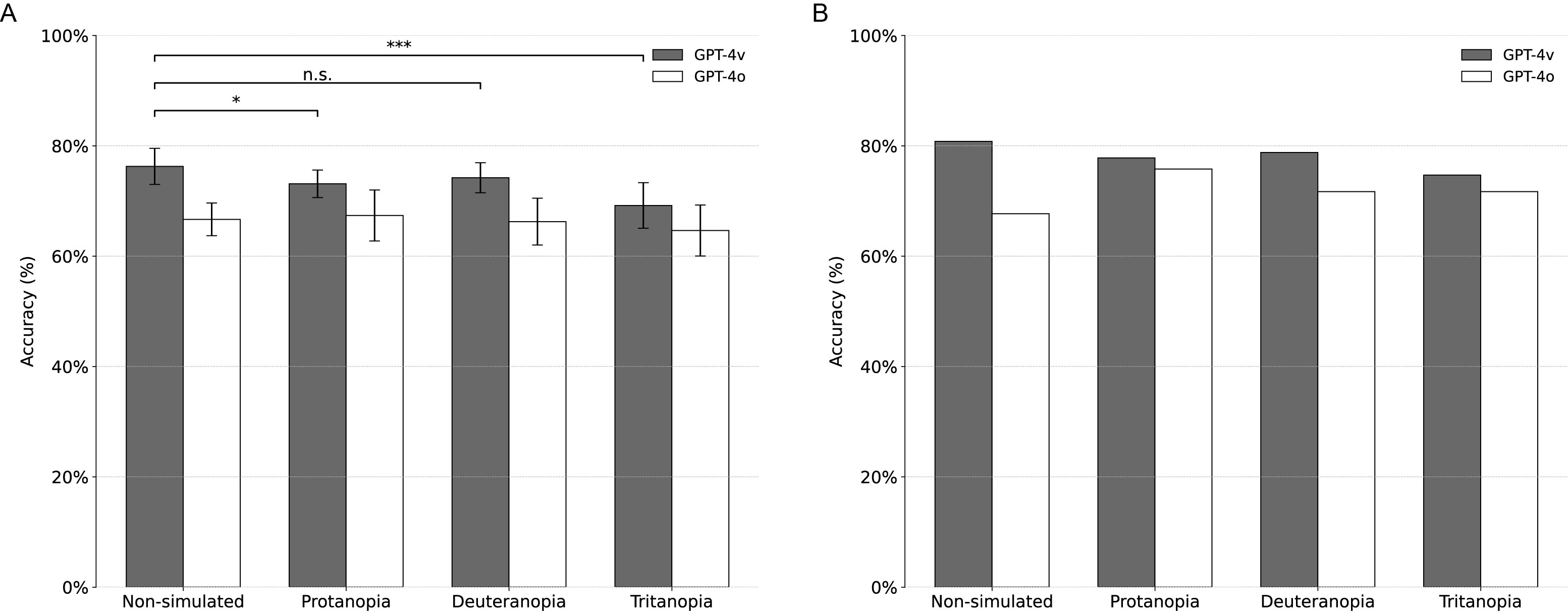
(A) Precios de clasificación promedio de GPT-4V para imágenes no simuladas y simuladas por CVD (Protanopia, Deuteranopia, Tritanopia). Barras de error: desviaciones estándar. *: p <0.05; **: P <0.01 (prueba t; dos colas). NS: No es significativo. Todos los experimentos fueron en diez repeticiones. (B) Precisión de clasificación de GPT-4V después de la aplicación de la estrategia de consenso en las diez repeticiones para cada consulta de imagen. GPT-4O incluido para comparación.
GPT-4V adaptó su interpretación de los hallazgos basados en color a diferentes simulaciones de CVD. Específicamente, no mencionó los colores rojos o rosados en las simulaciones de Protanope y Deuteranope, pero asoció estos colores con una predicción de melanoma en imágenes no simuladas. En las simulaciones tritanopías, caracterizadas por tonos rosados prevalentes, GPT-4V ya no trataron el rosa como una característica de melanoma.
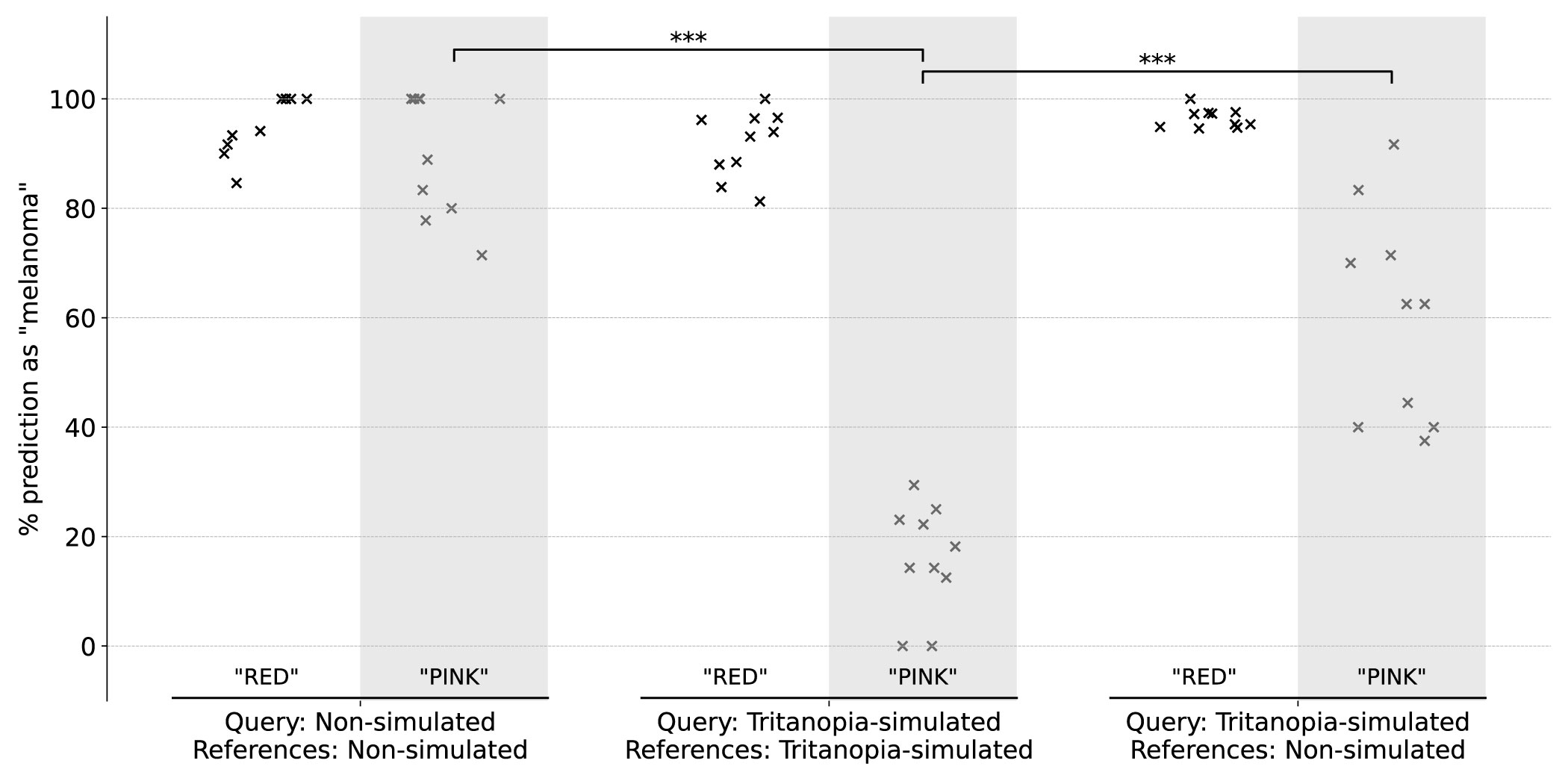
Para cada una de las condiciones no simuladas y simuladas por la tritanopia, se identificaron dos grupos de consultas de imágenes en base a las explicaciones de GPT-4V para sus predicciones. El primer grupo, etiquetado como "rojo", incluye descripciones en las que se menciona el color rojo para describir la imagen de consulta, independientemente del color rosa. El segundo grupo, etiquetado como "rosa", comprende descripciones que mencionan exclusivamente rosa sin rojo. El eje y muestra el porcentaje de predicciones de "melanoma" de cada replicada. Por defecto, tanto la consulta como las imágenes de referencia no estaban simuladas o simuladas a la misma condición CVD. Se realizó una prueba de ablación (las últimas dos columnas) para la simulación de la tritanopía muestreando las referencias de imágenes no simuladas. ***: P <0.001 (prueba t; dos colas).