Veuillez noter que ce référentiel est uniquement destiné à des fins d'archivage. Les fonctions ANCOM autonomes de ce référentiel ne sont plus maintenues. Il est fortement recommandé aux utilisateurs d'utiliser les fonctions ancom ou ancombc dans notre package ANCOMBC R. Pour les rapports de bogues, veuillez vous rendre sur le référentiel ANCOMBC.
Le code actuel implémente ANCOM dans des ensembles de données transversales et longitudinales tout en permettant l'utilisation de covariables. Les bibliothèques suivantes doivent être incluses pour que le code R s'exécute :
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Nous avons adopté la méthodologie ANCOM-II comme étape de prétraitement pour traiter différents types de zéros avant d'effectuer une analyse d'abondance différentielle.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : trame de données ou matrice représentant le tableau OTU/SV observé avec les taxons en lignes ( rownames ) et les échantillons en colonnes ( colnames ). Notez qu'il s'agit du tableau d'abondance absolue , ne le transformez pas en tableau d'abondance relative (où les totaux des colonnes sont égaux à 1).meta_data : trame de données ou matrice de toutes les variables et covariables d'intérêt.sample_var : Caractère. Nom de la colonne stockant les ID d’échantillon.group_var : Caractère. Le nom de l'indicateur de groupe. group_var est requis pour détecter les zéros structurels et les valeurs aberrantes. Pour les définitions des différents zéros (zéro structurel, zéro aberrant et zéro d'échantillonnage), veuillez vous référer à l'ANCOM-II.out_cut Fraction numérique comprise entre 0 et 1. Pour chaque taxon, les observations avec une proportion de distribution de mélange inférieure à out_cut seront détectées comme des zéros aberrants ; tandis que les observations avec une proportion de distribution de mélange supérieure à 1 - out_cut seront détectées comme des valeurs aberrantes.zero_cut : Fraction numérique comprise entre 0 et 1. Les taxons avec une proportion de zéros supérieure à zero_cut ne sont pas inclus dans l'analyse.lib_cut : Numérique. Les échantillons dont la taille de bibliothèque est inférieure à lib_cut ne sont pas inclus dans l'analyse.neg_lb : Logique. VRAI indique qu'un taxon serait classé comme zéro structurel dans le groupe expérimental correspondant en utilisant sa limite inférieure asymptotique. Plus précisément, neg_lb = TRUE indique que vous utilisez les deux critères énoncés dans la section 3.2 d'ANCOM-II pour détecter les zéros structurels ; Sinon, neg_lb = FALSE n'utilisera que l'équation 1 de la section 3.2 de l'ANCOM-II pour déclarer les zéros structurels. feature_table : Une trame de données de la table OTU prétraitée.meta_data : Une trame de données de métadonnées prétraitées.structure_zeros : Une matrice se compose de 0 et de 1, 1 indiquant que le taxon est identifié comme un zéro structurel dans le groupe correspondant.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : bloc de données représentant la table OTU/SV avec des taxons en lignes ( rownames ) et des échantillons en colonnes ( colnames ). Il peut s'agir de la valeur de sortie de feature_table_pre_process . Notez qu'il s'agit du tableau d'abondance absolue , ne le transformez pas en tableau d'abondance relative (où les totaux des colonnes sont égaux à 1).meta_data : trame de données de variables. Peut être la valeur de sortie de feature_table_pre_process .struc_zero : Une matrice se compose de 0 et de 1, 1 indiquant que le taxon est identifié comme un zéro structurel dans le groupe correspondant. Peut être la valeur de sortie de feature_table_pre_process .main_var : Caractère. Le nom de la principale variable d’intérêt. ANCOM v2.1 prend actuellement en charge la catégorie main_var .p_adjust_method : Caractère. Spécifier la méthode pour ajuster les valeurs p pour plusieurs comparaisons. La valeur par défaut est « BH » (procédure Benjamin-Hochberg).alpha : Niveau de signification. La valeur par défaut est 0,05.adj_formula : Chaîne de caractères représentant la formule de réglage (voir exemple).rand_formula : Chaîne de caractères représentant la formule des effets aléatoires dans lme . Pour plus de détails, voir ?lme .lme_control : Une liste spécifiant les valeurs de contrôle pour lme fit. Pour plus de détails, consultez ?lmeControl . 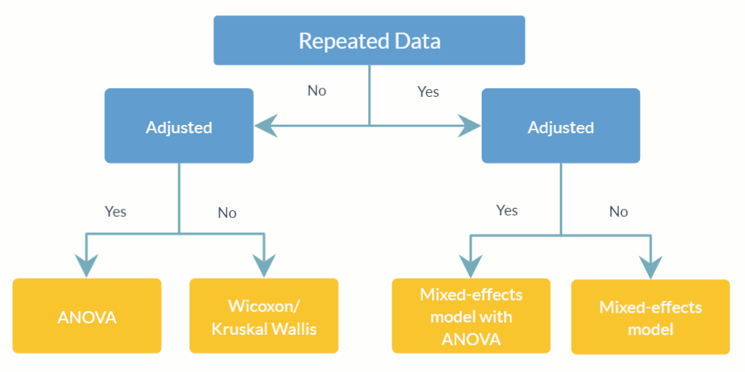
out : Une base de données avec la statistique W pour chaque taxon et les colonnes suivantes qui sont des indicateurs logiques indiquant si une OTU ou un taxon est différentiellement abondant sous une série de seuils (0,9, 0,8, 0,7 et 0,6). detected_0.7 est couramment utilisé. Cependant, vous pouvez choisir detected_0.8 ou detected_0.9 si vous souhaitez être plus conservateur dans vos résultats (FDR plus petit), ou utiliser detected_0.6 si vous souhaitez explorer plus de découvertes (puissance plus grande).
fig : Un objet ggplot de volcan plot.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Ici, nous aimerions savoir s'il existe des zéros structurels aux différents niveaux de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Ici, nous aimerions savoir s'il existe des zéros structurels aux différents niveaux de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Ici, nous aimerions savoir s'il existe des zéros structurels aux différents niveaux de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )