Chargement rapide de données par lots de fichiers BigWig contenant des données de piste épigente et des séquences correspondantes alimentées par GPU pour les applications d'apprentissage en profondeur.
Bigwig-loader dépend principalement de la bibliothèque rapidsai kvikio et de cupy, qui sont toutes deux mieux installées en utilisant conda/mamba. Bigwig-loader peut désormais également être installé en utilisant conda/mamba. Pour créer un nouvel environnement avec bigwig-loader installé :
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderOu ajoutez ceci à votre fichier environnement.yml :
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderet mise à jour :
mamba env update -f environment.ymlBigwig-loader peut également être installé à l'aide de pip dans un environnement sur lequel la bibliothèque rapidsai kvikio et cupy sont déjà installés :
pip install bigwig-loaderNous avons encapsulé le BigWigDataset dans un ensemble de données itérable PyTorch que vous pouvez directement utiliser :
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Un objet Dataset indépendant du framework peut être importé à partir de bigwig_loader.dataset . Cet objet d'ensemble de données renvoie des tenseurs Cupy. Les tenseurs Cupy adhèrent à l'interface du tableau cuda et peuvent être transformés sans copie en tenseurs JAX ou tensorflow.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Voir le répertoire d'exemples pour plus d'exemples.
Cette bibliothèque est destinée au chargement de lots de données avec la même dimensionnalité, ce qui permet certaines hypothèses pouvant accélérer le processus de chargement. Comme le montre le graphique ci-dessous, lors du chargement d'une petite quantité de données, pyBigWig est très rapide, mais n'exploite pas la nature par lots du chargement des données pour l'apprentissage automatique.
Dans le benchmark ci-dessous, nous avons également créé des chargeurs de données PyTorch (avec set_start_method('spawn')) en utilisant pyBigWig pour comparer au scénario réaliste où plusieurs processeurs seraient utilisés par GPU. Nous voyons que le débit du chargeur de données CPU n'augmente pas de manière linéaire avec le nombre de CPU, et il devient donc difficile d'obtenir le débit nécessaire pour maintenir le GPU, entraînant le réseau neuronal, saturé pendant les étapes d'apprentissage.
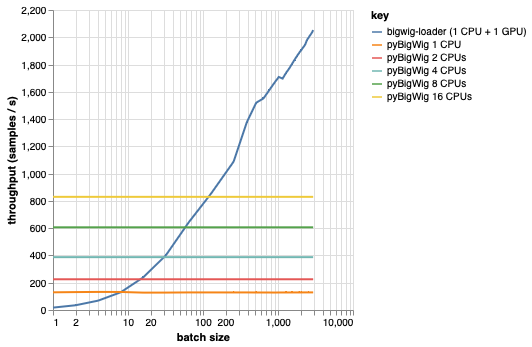
C’est le problème que bigwig-loader résout. Voici un exemple d'utilisation de bigwig-loader :
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml Dans cet environnement, vous devriez pouvoir exécuter pytest -v et voir les tests réussir. REMARQUE : vous avez besoin d'un GPU pour utiliser bigwig-loader !
Cette section vous guide à travers les étapes nécessaires pour ajouter de nouvelles fonctionnalités. Si quelque chose n'est pas clair, veuillez ouvrir un problème.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install pour installer les hooks de pré-validationLes tests sont dans le répertoire tests. L'un des tests les plus importants est test_against_pybigwig qui garantit que s'il y a une erreur dans pyBigWIg, elle l'est également dans bigwig-loader.
pytest -vv .Lorsque les exécuteurs Github avec GPU seront disponibles, nous aimerions également exécuter ces tests dans le CI. Mais pour l’instant, vous pouvez les exécuter localement.
Si vous utilisez cette bibliothèque, pensez à citer :
Retel, Joren Sebastian, Andreas Poehlmann, Josh Chiou, Andreas Steffen et Djork-Arné Clevert. «Un chargeur de données d'apprentissage automatique rapide pour les pistes épigénétiques à partir de fichiers BigWig.» Bioinformatique 40, non. 1 (1er janvier 2024) : btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}