
(c) 2017 Timothy Becker et Wan-Ping Lee
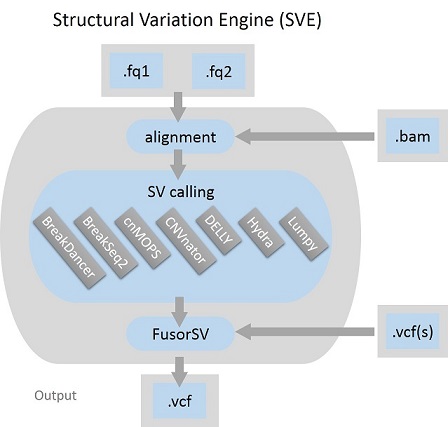
SVE est un moteur d'exécution basé sur un script Python pour la détection des variations structurelles (SV). Il peut être utilisé pour tous les niveaux d'entrées de données, de FASTQ bruts, de BAM alignés ou de format d'appel de variantes (VCF), et génère un VCF unifié comme sortie. De par sa conception, SVE comprend par défaut l'alignement, le réalignement et l'ensemble des algorithmes d'appel SV de pointe. Il s'agit de BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra et LUMPY. FusorSV est également intégré, il s'agit d'une approche d'exploration de données permettant d'évaluer les performances et de fusionner des ensembles d'appels à partir d'un ensemble d'algorithmes d'appel SV.
Veuillez définir l'environnement ROOT.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeVeuillez vérifier les fichiers d'en-tête python2.7 et modifier "CFLAGS_FUSOR_SV" dans Makefile. Les fichiers d'en-tête peuvent se trouver sur "/usr/include/python2.7" et utiliser "CFLAGS_FUSOR_SV=-I /usr/include/python2.7" à la place.
make FusorSV
Ou, vous pouvez installer FusorSV par setup.py
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
Alternativement, Dockerfile et Docker image sont fournis. Veuillez noter que sudo peut être requis pour les utilisations de Docker en fonction des paramètres de votre machine.
cd SVE
docker build .
Extrayez l'image Docker du référentiel.
docker pull wanpinglee/sve
SVE est construit sur /tools/SVE. Consultez l'aide par
/tools/SVE/bin/sve
Les lectures courtes dans FASTQ seront mappées sur le FASTA donné et un BAM trié sera généré.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
Si les lectures sont données au format BAM, le réalignage remappera les lectures par rapport à FASTA et générera un BAM trié. Nous utilisons SpeedSeq pour réaliser le réalignage.
bin/sve realign -r <FASTA> <BAM>
Il existe sept algorithmes d'appel SV qui peuvent être utilisés pour les appels SV. VCF sera généré.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
Après l'appel, chaque échantillon peut avoir plusieurs VCF en fonction du nombre d'appelants utilisés. Veuillez collecter les VCF d'un échantillon dans un dossier.
Les vcfs doivent utiliser les ID SVE pour indiquer les appelants.
| ID SVE | Demandeur |
|---|---|
| 4 | BreakDancer (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnateur (v0.3.3) |
| 11 | DELLY (v2) |
| 14* | GénomeSTRIP |
| 17 | Hydre |
| 18 | GRUMELEUX |
| 35 | BreakSeq (v2.2) |
| 0 | Vérité (facultatif) |
Remarque* : En raison d'un problème de licence, GenomeSTRiP n'est pas intégré à SVE. Cependant, le modèle par défaut de FusorSV est capable de gérer GenomeSTRiP VCF.
Des exemples de fichiers vcf d’entrée peuvent être organisés comme suit. Veuillez noter que vcfFiles est l'argument de -i pour FusorSV.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Selon S0.vcf, un nouveau modèle sera généré et les VCF seront fusionnés par le nouveau modèle.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Le projet est sous licence GPL-3.0. Veuillez consulter LICENCE pour plus de détails.