
Le langage R# est une sorte de langage apprécié par R implémenté sur l'environnement .NET pour l'analyse des données bioinformatiques.
[AVERTISSEMENT] Ce projet est un travail en cours et n'est pas recommandé pour une utilisation en production.
Le dernier runtime sciBASIC.NET Framework est également requis
Le langage R# sa syntaxe est originale dérivée du langage R , mais avec des styles de programmation plus modernisés. Le langage R# son interpréteur et son compilateur .NET sont écrits à l'origine en langage VisualBasic, avec une prise en charge native du runtime .NET.
Le langage R# n'est pas conçu à des fins générales d'analyse de données, mais il est spécialement conçu pour mes travaux dans l'entreprise, implémente le système d'analyse de données bioinformatiques basé sur la plateforme GCModeller, pour construire la pile de science des données bioinformatiques avec le langage R et VisualBasic. .
R# Le moteur d'exécution et de script principal du langage R#Library La bibliothèque de base dans le système de script R#Rscript L'hôte de script R#R-terminal Le programme shell R#Rsharp_kit La boîte à outils R-sharpnjl Le langage Julia aimait le moteur de scriptnpy Le langage Python aime le moteur de scriptRData Le lecteur de fichiers *.rda/*.rds données du langage R # declare a variable
let word as string = [ ' world ' , ' R# user ' , ' GCModeller user ' ];
# declare a function
let echo as function ( words ) {
print( `Hello ${ words }!` );
}
# or declare a lambda function
let echo.lambda = words - > print( `Hello ${ words }!` );
# and then invoke function via pipeline operator
word : > echo ;
# [3] "Hello world!" "Hello R# user!" "Hello GCModeller user!"
word : > echo.lambda ;
# [3] "Hello world!" "Hello R# user!" "Hello GCModeller user!"Utilisé dans la programmation VisualBasic.NET :
Dim R As New RInterpreter()
' Run script by invoke method
Call R.Evaluate( "
# test script
let word as string = [ 'world', 'R# user', 'GCModeller user'];
let echo as function (words) {
print( ` Hello $ { words }! ` ) ;
}
word :> echo ;
" )
' or assign variable
Call R.Add( "word" , { "world" , "R# user" , "GCModeller user" })
' then declare R function throught script
Call R.Add( "echo" ,
Function (words As String ()) As String ()
Return Internal.print(words)
End Function )
' at last, invoke R function throught Invoke method
Call R.Invoke( "echo" , R!word) 
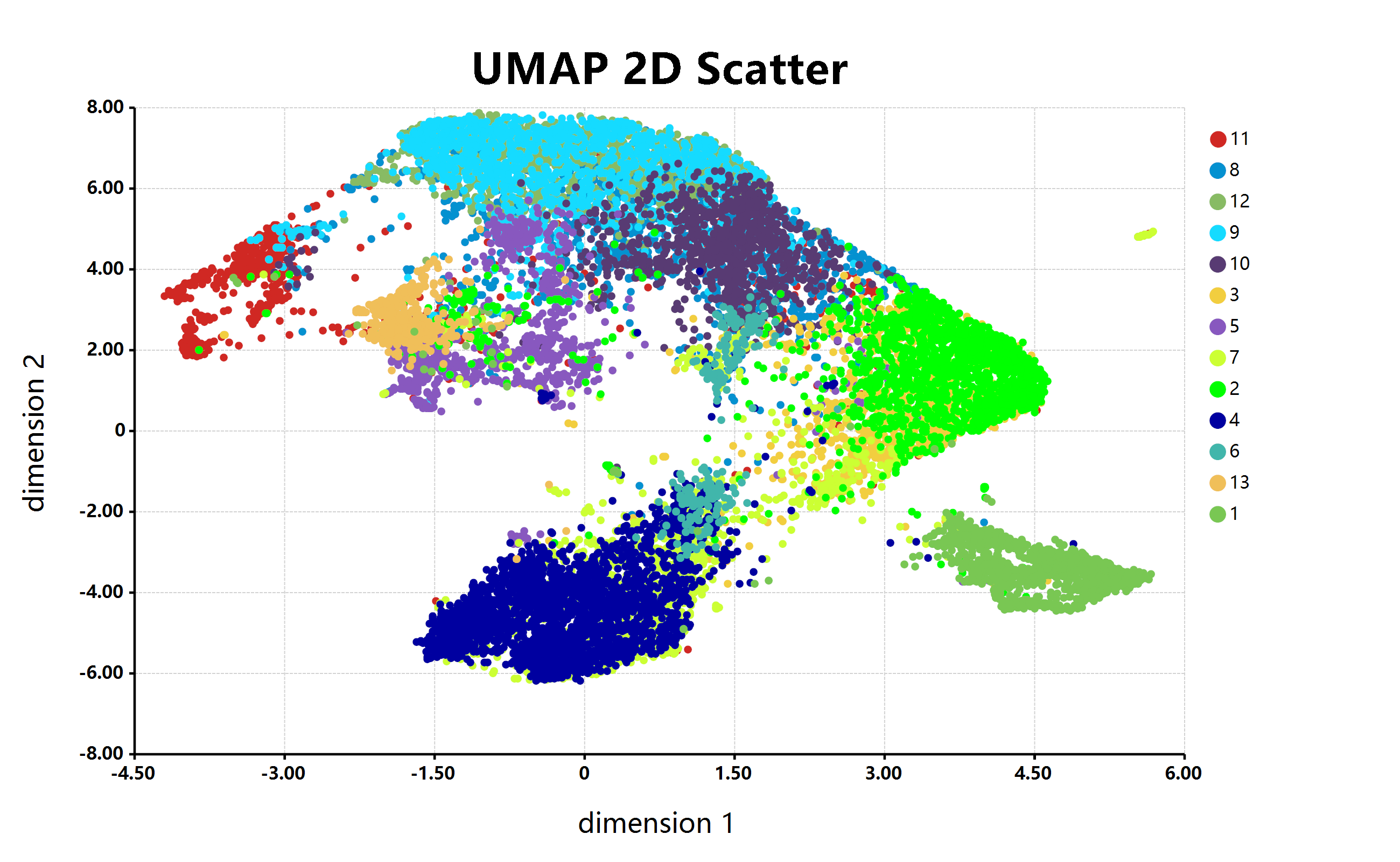
# read scatter point data from a given table file
# and then assign to tuple variables
[ x , y , cluster ] = read.csv( " ./scatter.csv " , row.names = NULL );
# umap scatter with class colors
bitmap( file = " ./scatter.png " ) {
plot( x , y ,
padding = " padding:200px 400px 200px 250px; " ,
class = cluster ,
title = " UMAP 2D Scatter " ,
x.lab = " dimension 1 " ,
y.lab = " dimension 2 " ,
legend.block = 13 ,
colorSet = " paper " ,
grid.fill = " transparent " ,
size = [ 2600 , 1600 ]
);
};R#Le package ggplot2 du langage AR apprécie la grammaire de la bibliothèque graphique pour la programmation en langage R#.
Le langage R# est un autre langage de calcul scientifique conçu pour l'exécution .NET, R# est une évolution du langage R. Il existe une célèbre bibliothèque graphique appelée ggplot2 en langage R, donc reste la même, il existe une bibliothèque graphique appelée ggplot qui a été développée pour le langage R# .
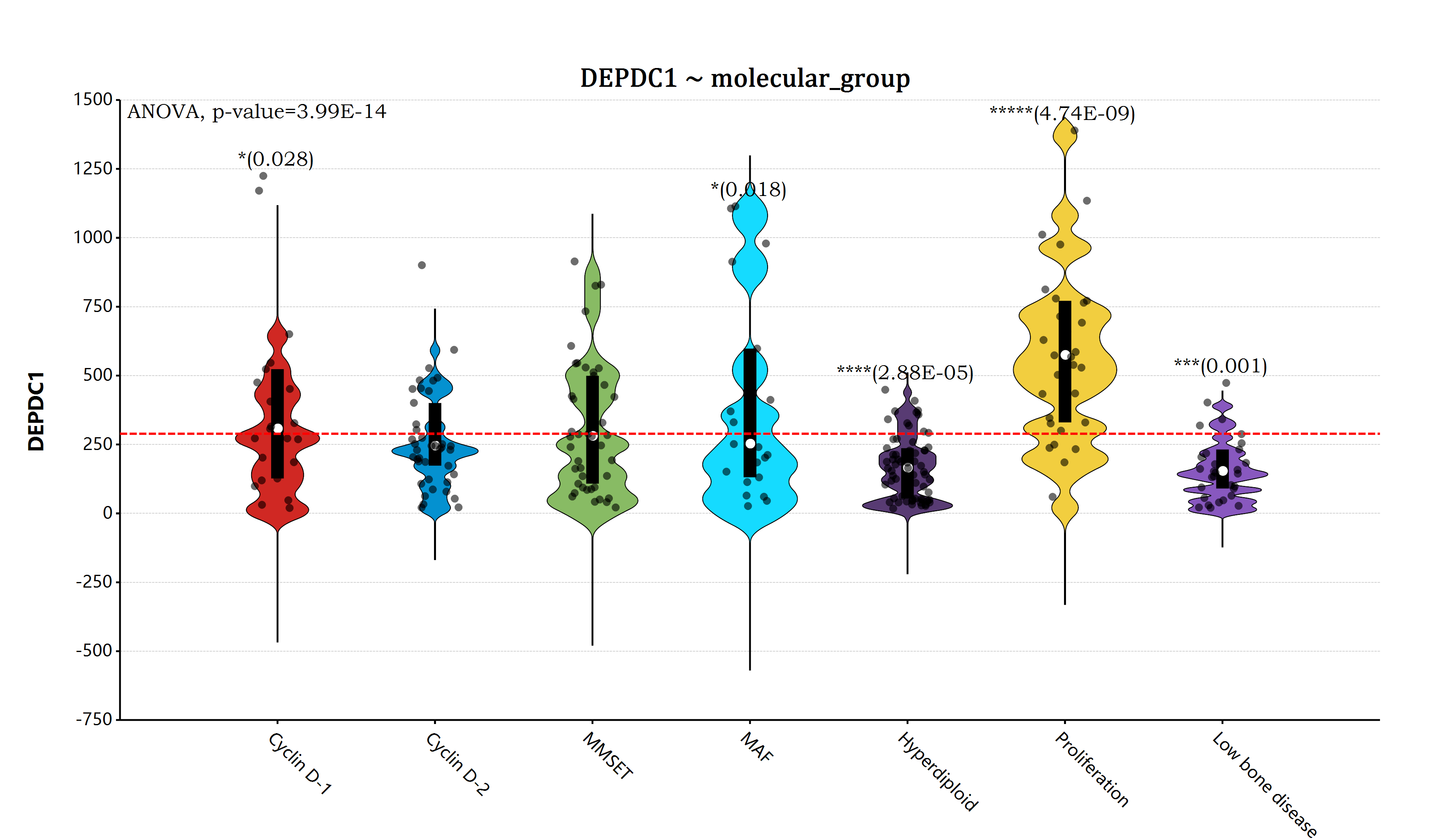
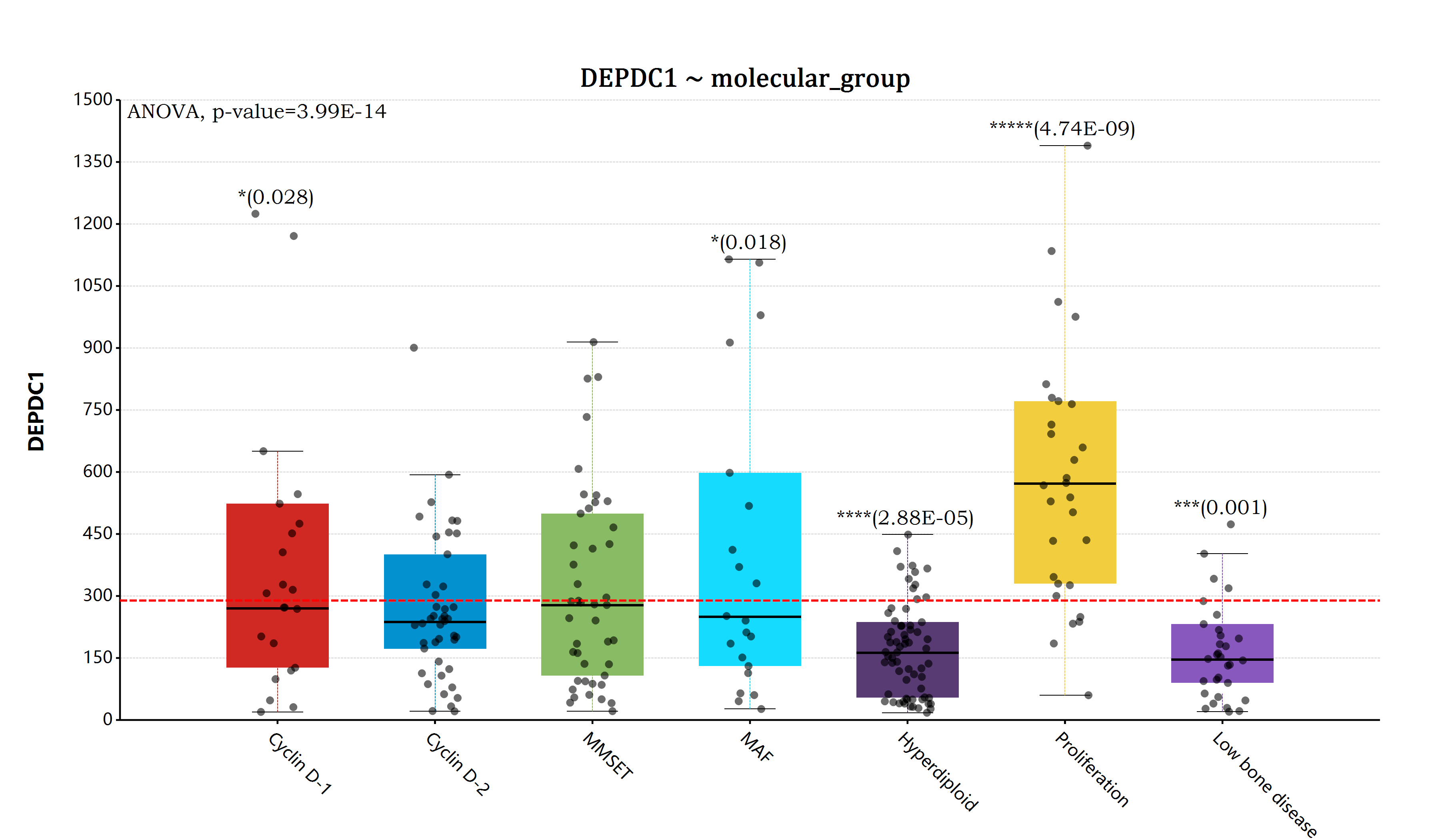
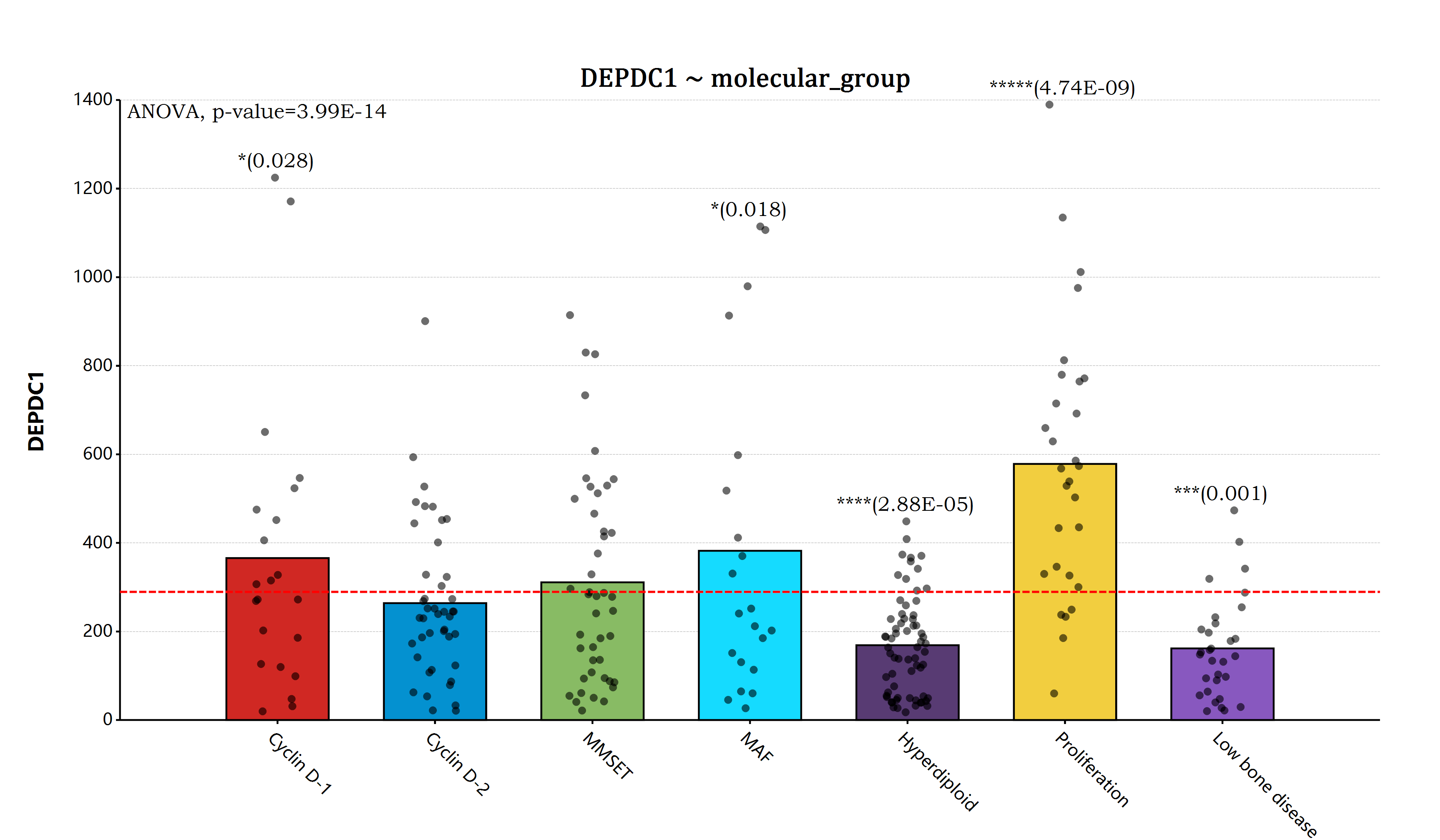
ggplot( myeloma , aes( x = " molecular_group " , y = " DEPDC1 " ))
+ geom_boxplot( width = 0.65 )
+ geom_jitter( width = 0.3 )
# Add horizontal line at base mean
+ geom_hline( yintercept = mean( myeloma $ DEPDC1 ), linetype = " dash " , line.width = 6 , color = " red " )
+ ggtitle( " DEPDC1 ~ molecular_group " )
+ ylab( " DEPDC1 " )
+ xlab( " " )
+ scale_y_continuous( labels = " F0 " )
# Add global annova p-value
+ stat_compare_means( method = " anova " , label.y = 1600 )
# Pairwise comparison against all
+ stat_compare_means( label = " p.signif " , method = " t.test " , ref.group = " .all. " , hide.ns = TRUE )
+ theme(
axis.text.x = element_text( angle = 45 ),
plot.title = element_text( family = " Cambria Math " , size = 16 )
)
; Le système R# ne prend pas seulement en charge le langage R#, il comprend également un moteur de script en langage Python et un moteur de script en langage Julia qui s'exécute sur le runtime R# .
Référence du script python ou du script Julia en langage R# tout comme l'importation d'autres scripts R# :
# imports an external R# script
imports " ./script.R " ;
# imports an external python script in R#
imports " ./script.py " ;
# imports an external julia script in R#
imports " ./script.jl " ;Et vous pouvez également importer un script R en script Python ou Julia :
# example of import R#/julia script in python
# imports an external R# script in python
import "./script.R"
# imports an external julia script in python
import "./script.jl" importer des scripts python et R# dans les scripts Julia reste également simple :
# example of imports R#/python script in julia
# imports an external R# script in julia
include ( " ./script.R " )
# imports an external python script in julia
include ( " ./script.py " ) require ( "GCModeller" ) ;
// load the fastq module from rna-seq package
// inside the GCModeller
import { FastQ } from "rnaseq" ;
// do short reads assembling via SCS algorithm
var assem = FastQ . assemble ( [
"AACAAATGAGACGCTGTGCAATTGCTGA" ,
"AACAAATGAGACGCTGTGCAATTGCAAA" ,
"CAAATGAGACGCTGTGCAATTGCTGAGT" ,
"GCAAATGATACGCTGTGCAATTGCTAGA" ,
"ATGAGACGCTGTGCAATTGCTGAGTACC" ,
"CTGTGCAATTGCTGAGAACAAATGAGAC" ,
"CTGTGCAATTGCTAGAAACAAATGAGAC"
] )
// view the short reads assemble result
console . table ( assem )
// Loading required package: GCModeller
// Loading required package: igraph
// Attaching package: 'igraph'
//
// The following object is masked from 'package:igraph':
//
// eval, class
//
//
//
// GCModeller: genomics CAD(Computer Assistant Design) Modeller System
// author by: [email protected]
//
// (c) 2023 | SMRUCC genomics - GuiLin, China
//
// AssembleResult
// --------------------------------------------------------------------------------------------------------------
// <mode> <string>
// [1, ] "CTGTGCAATTGCTGAGAACAAATGAGACGCTGTGCAATTGCAAATGATACGCTGTGCAATTGCTAGAAACAAATGAGACGCTGTGCAATTGCTGAGTACC"
// [2, ] "...................................................................AACAAATGAGACGCTGTGCAATTGCTGA....."
// [3, ] "................AACAAATGAGACGCTGTGCAATTGCAAA........................................................"
// [4, ] ".....................................................................CAAATGAGACGCTGTGCAATTGCTGAGT..."
// [5, ] ".......................................GCAAATGATACGCTGTGCAATTGCTAGA................................."
// [6, ] "........................................................................ATGAGACGCTGTGCAATTGCTGAGTACC"
// [7, ] "CTGTGCAATTGCTGAGAACAAATGAGAC........................................................................"
// [8, ] "...................................................CTGTGCAATTGCTAGAAACAAATGAGAC....................." R# associéePackages développés pour l'environnement de programmation R# :