Un délicieux outil d'apprentissage automatique qui vous permet de former/adapter, tester et utiliser des modèles sans écrire de code
Note
Je travaille également sur une application de bureau GUI pour igel basée sur les demandes des gens. Vous pouvez le trouver sous Igel-UI.
Table des matières
L'objectif du projet est de fournir un apprentissage automatique à tous , utilisateurs techniques et non techniques.
J'avais parfois besoin d'un outil que je puisse utiliser pour créer rapidement un prototype d'apprentissage automatique. Qu'il s'agisse de construire une preuve de concept, de créer un modèle de brouillon rapide pour prouver un point ou d'utiliser le ML automatique. Je me retrouve souvent coincé à écrire du code passe-partout et à trop réfléchir par où commencer. J'ai donc décidé de créer cet outil.
igel est construit sur d'autres frameworks ML. Il fournit un moyen simple d'utiliser l'apprentissage automatique sans écrire une seule ligne de code . Igel est hautement personnalisable , mais seulement si vous le souhaitez. Igel ne vous oblige pas à rien personnaliser. Outre les valeurs par défaut, igel peut utiliser les fonctionnalités de ml automatique pour déterminer un modèle qui fonctionne parfaitement avec vos données.
Tout ce dont vous avez besoin est un fichier yaml (ou json ), dans lequel vous devez décrire ce que vous essayez de faire. C'est ça!
Igel prend en charge la régression, la classification et le clustering. Igel prend en charge les fonctionnalités de ml automatique telles que ImageClassification et TextClassification
Igel prend en charge les types d'ensembles de données les plus utilisés dans le domaine de la science des données. Par exemple, votre ensemble de données d'entrée peut être un fichier csv, txt, Excel, json ou même html que vous souhaitez récupérer. Si vous utilisez les fonctionnalités de ml automatique, vous pouvez même transmettre des données brutes à igel et il saura comment les gérer. Nous en parlerons plus tard dans les exemples.
$ pip install -U igel Modèles pris en charge par Igel :
+--------------------+----------------------------+-------------------------+
| regression | classification | clustering |
+--------------------+----------------------------+-------------------------+
| LinearRegression | LogisticRegression | KMeans |
| Lasso | Ridge | AffinityPropagation |
| LassoLars | DecisionTree | Birch |
| BayesianRegression | ExtraTree | AgglomerativeClustering |
| HuberRegression | RandomForest | FeatureAgglomeration |
| Ridge | ExtraTrees | DBSCAN |
| PoissonRegression | SVM | MiniBatchKMeans |
| ARDRegression | LinearSVM | SpectralBiclustering |
| TweedieRegression | NuSVM | SpectralCoclustering |
| TheilSenRegression | NearestNeighbor | SpectralClustering |
| GammaRegression | NeuralNetwork | MeanShift |
| RANSACRegression | PassiveAgressiveClassifier | OPTICS |
| DecisionTree | Perceptron | KMedoids |
| ExtraTree | BernoulliRBM | ---- |
| RandomForest | BoltzmannMachine | ---- |
| ExtraTrees | CalibratedClassifier | ---- |
| SVM | Adaboost | ---- |
| LinearSVM | Bagging | ---- |
| NuSVM | GradientBoosting | ---- |
| NearestNeighbor | BernoulliNaiveBayes | ---- |
| NeuralNetwork | CategoricalNaiveBayes | ---- |
| ElasticNet | ComplementNaiveBayes | ---- |
| BernoulliRBM | GaussianNaiveBayes | ---- |
| BoltzmannMachine | MultinomialNaiveBayes | ---- |
| Adaboost | ---- | ---- |
| Bagging | ---- | ---- |
| GradientBoosting | ---- | ---- |
+--------------------+----------------------------+-------------------------+Pour le ML automatique :
La commande help est très utile pour vérifier les commandes prises en charge et les arguments/options correspondants
$ igel --helpVous pouvez également exécuter de l'aide sur les sous-commandes, par exemple :
$ igel fit --helpIgel est hautement personnalisable. Si vous savez ce que vous voulez et souhaitez configurer votre modèle manuellement, consultez les sections suivantes, qui vous guideront sur la façon d'écrire un fichier de configuration yaml ou json. Après cela, il vous suffit de dire à igel quoi faire et où trouver vos données et votre fichier de configuration. Voici un exemple :
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml 'Cependant, vous pouvez également utiliser les fonctionnalités de ml automatique et laisser igel faire tout pour vous. Un bon exemple serait la classification des images. Imaginons que vous disposiez déjà d'un ensemble de données d'images brutes stockées dans un dossier appelé images.
Il ne vous reste plus qu'à exécuter :
$ igel auto-train --data_path ' path_to_your_images_folder ' --task ImageClassificationC'est ça! Igel lira les images du répertoire, traitera l'ensemble de données (conversion en matrices, redimensionnement, fractionnement, etc...) et commencera à former/optimiser un modèle qui fonctionne bien sur vos données. Comme vous pouvez le constater, c'est assez simple, il vous suffit de fournir le chemin d'accès à vos données et la tâche que vous souhaitez effectuer.
Note
Cette fonctionnalité est coûteuse en termes de calcul, car igel essaierait de nombreux modèles différents et comparerait leurs performances afin de trouver le « meilleur ».
Vous pouvez exécuter la commande help pour obtenir des instructions. Vous pouvez également exécuter de l'aide sur les sous-commandes !
$ igel --helpLa première étape consiste à fournir un fichier yaml (vous pouvez également utiliser json si vous le souhaitez)
Vous pouvez le faire manuellement en créant un fichier .yaml (appelé igel.yaml par convention mais vous pouvez le nommer comme vous le souhaitez) et en le modifiant vous-même. Cependant, si vous êtes paresseux (et vous l'êtes probablement, comme moi :D), vous pouvez utiliser la commande igel init pour démarrer rapidement, qui créera un fichier de configuration de base pour vous à la volée.
"""
igel init --help
Example:
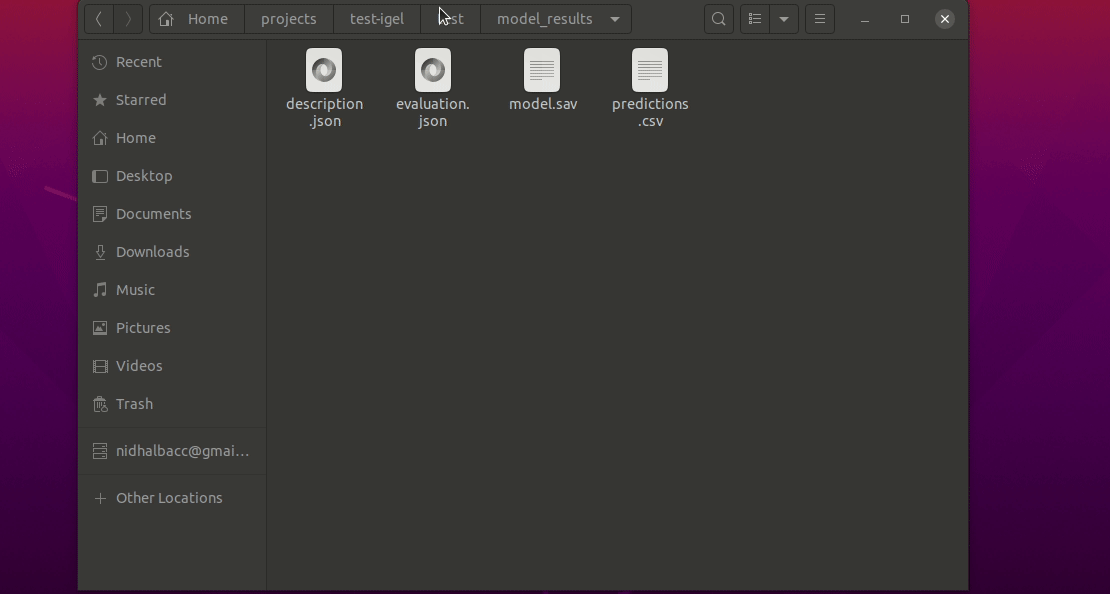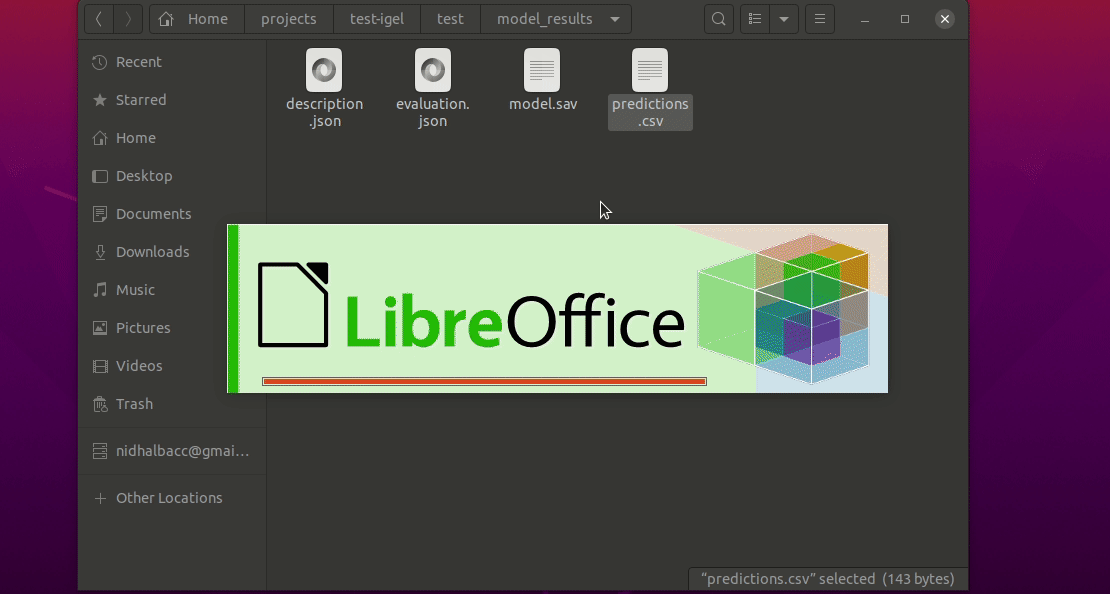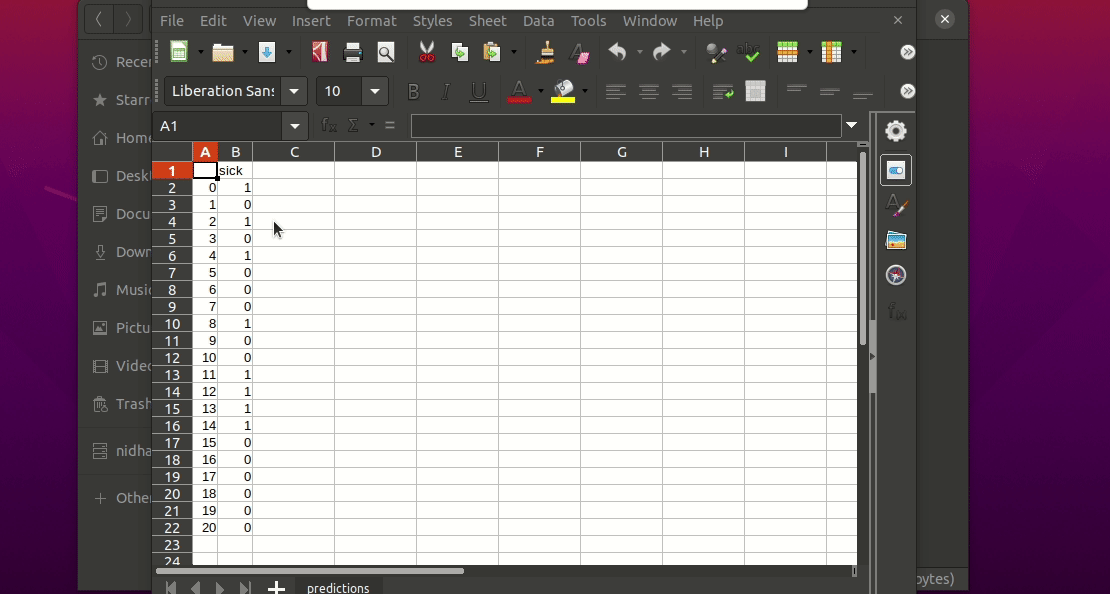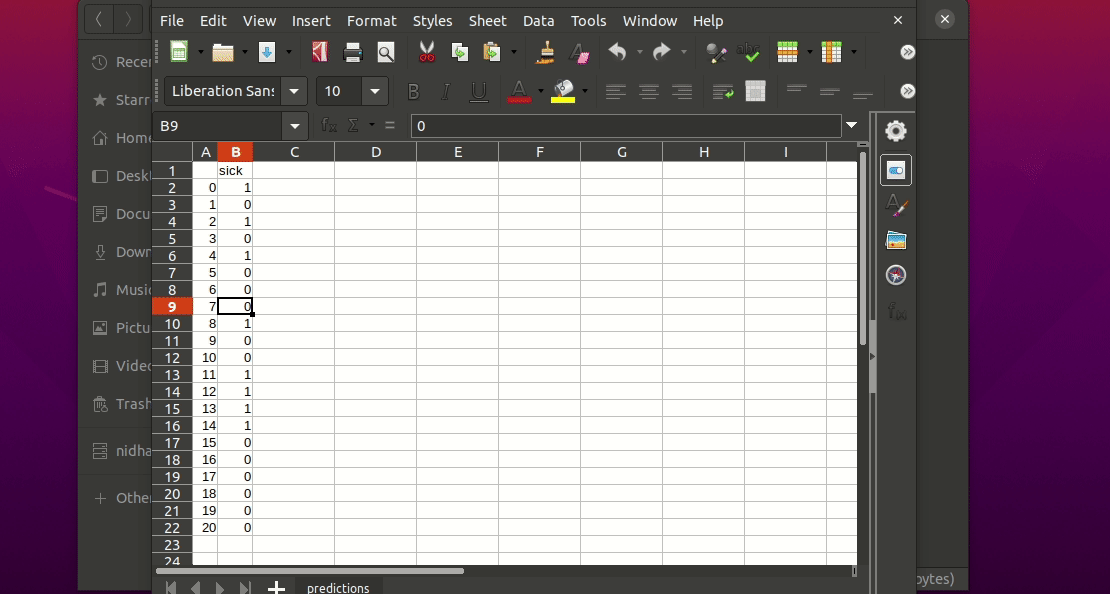
If I want to use neural networks to classify whether someone is sick or not using the indian-diabetes dataset,
then I would use this command to initialize a yaml file n.b. you may need to rename outcome column in .csv to sick:
$ igel init -type " classification " -model " NeuralNetwork " -target " sick "
"""
$ igel initAprès avoir exécuté la commande, un fichier igel.yaml sera créé pour vous dans le répertoire de travail actuel. Vous pouvez le consulter et le modifier si vous le souhaitez, sinon vous pouvez également tout créer à partir de zéro.
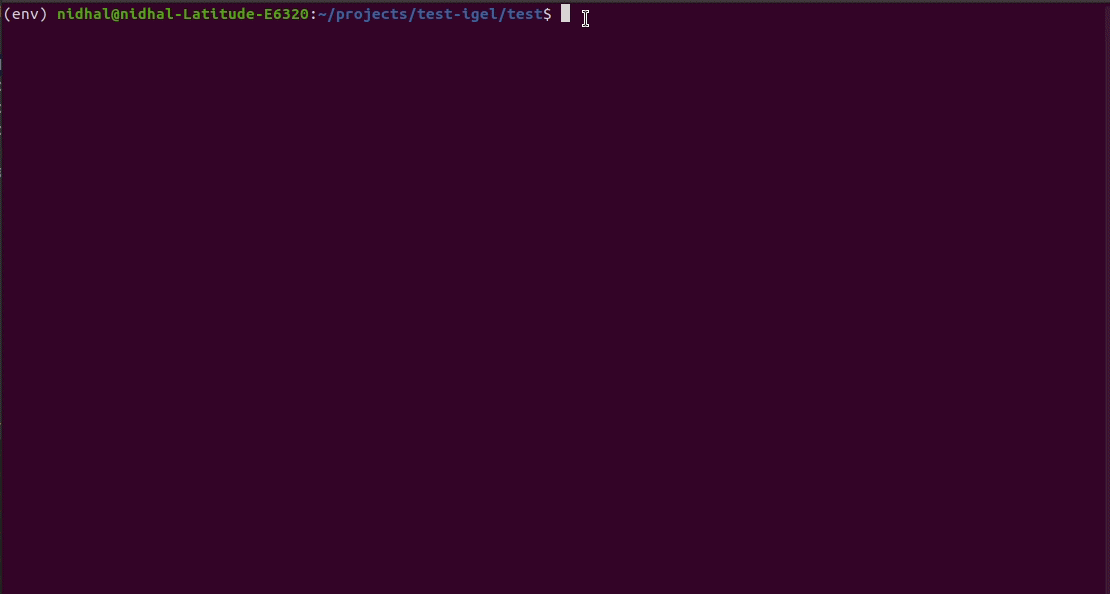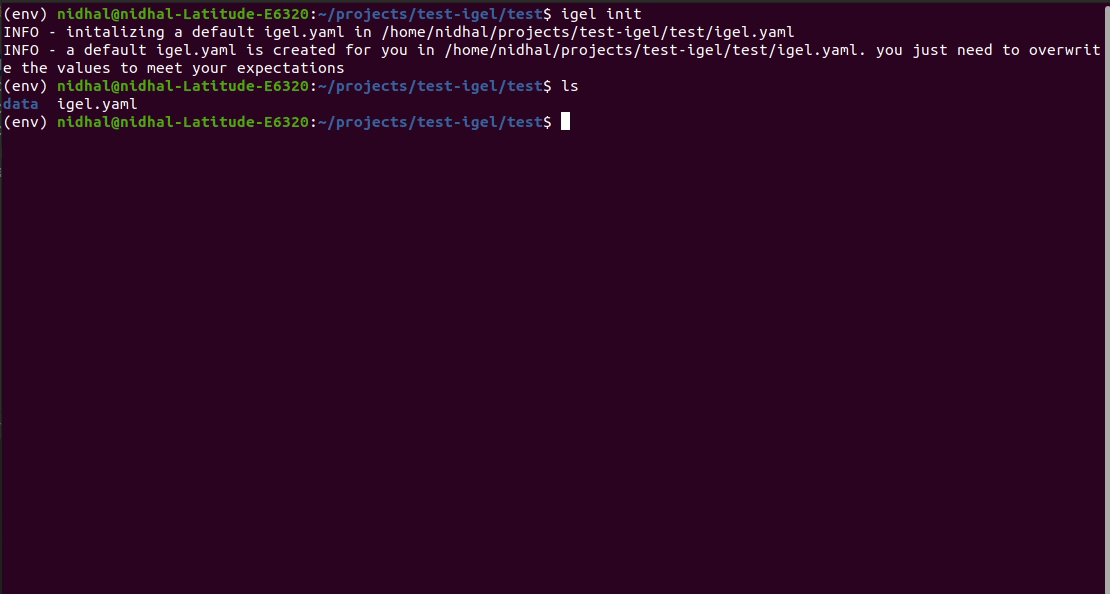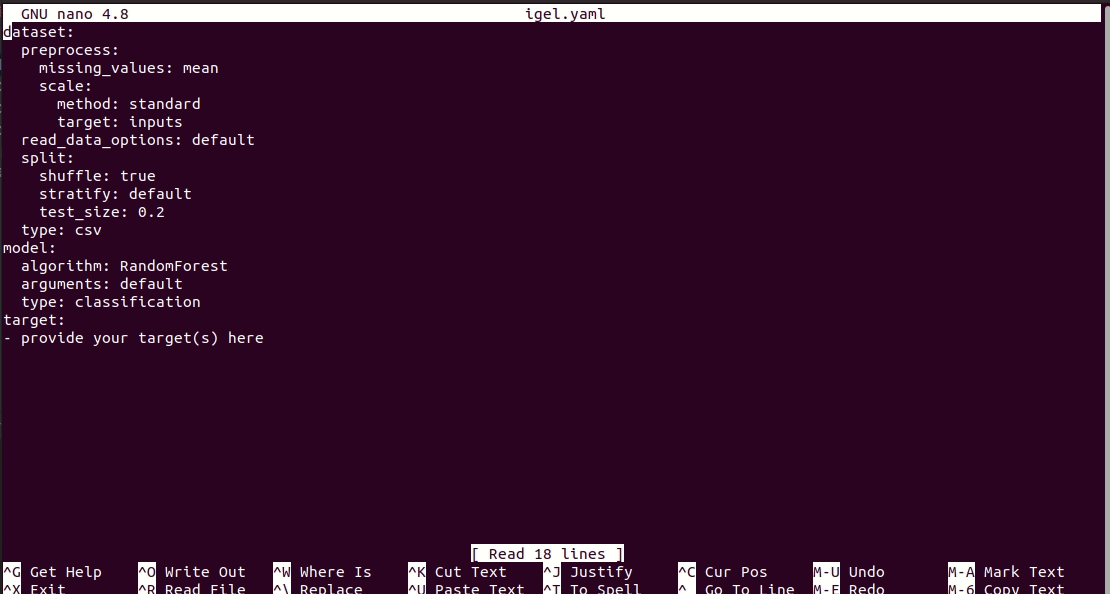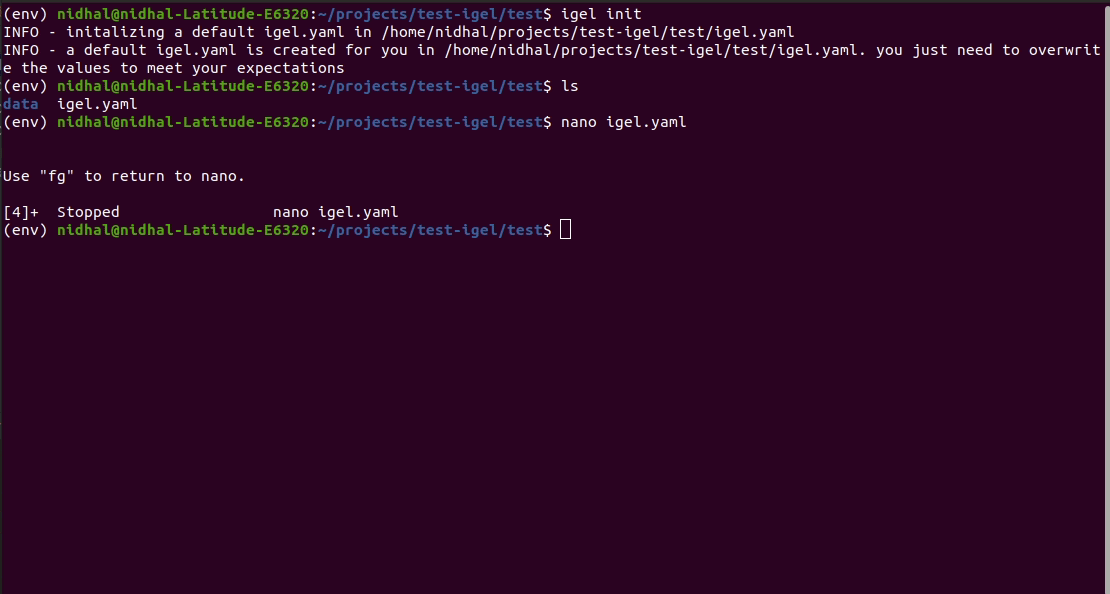
# model definition
model :
# in the type field, you can write the type of problem you want to solve. Whether regression, classification or clustering
# Then, provide the algorithm you want to use on the data. Here I'm using the random forest algorithm
type : classification
algorithm : RandomForest # make sure you write the name of the algorithm in pascal case
arguments :
n_estimators : 100 # here, I set the number of estimators (or trees) to 100
max_depth : 30 # set the max_depth of the tree
# target you want to predict
# Here, as an example, I'm using the famous indians-diabetes dataset, where I want to predict whether someone have diabetes or not.
# Depending on your data, you need to provide the target(s) you want to predict here
target :
- sickDans l'exemple ci-dessus, j'utilise une forêt aléatoire pour classer si une personne est diabétique ou non en fonction de certaines caractéristiques de l'ensemble de données. J'ai utilisé le célèbre diabète indien dans cet exemple d'ensemble de données sur le diabète indien)
Notez que j'ai passé n_estimators et max_depth comme arguments supplémentaires au modèle. Si vous ne fournissez aucun argument, la valeur par défaut sera utilisée. Vous n'avez pas besoin de mémoriser les arguments pour chaque modèle. Vous pouvez toujours exécuter igel models dans votre terminal, ce qui vous amènera en mode interactif, où vous serez invité à saisir le modèle que vous souhaitez utiliser et le type de problème que vous souhaitez résoudre. Igel vous montrera ensuite des informations sur le modèle et un lien que vous pourrez suivre pour voir une liste des arguments disponibles et comment les utiliser.
Exécutez cette commande dans le terminal pour ajuster/entraîner un modèle, où vous fournissez le chemin d'accès à votre ensemble de données et le chemin d'accès au fichier yaml
$ igel fit --data_path ' path_to_your_csv_dataset.csv ' --yaml_path ' path_to_your_yaml_file.yaml '
# or shorter
$ igel fit -dp ' path_to_your_csv_dataset.csv ' -yml ' path_to_your_yaml_file.yaml '
"""
That's it. Your "trained" model can be now found in the model_results folder
(automatically created for you in your current working directory).
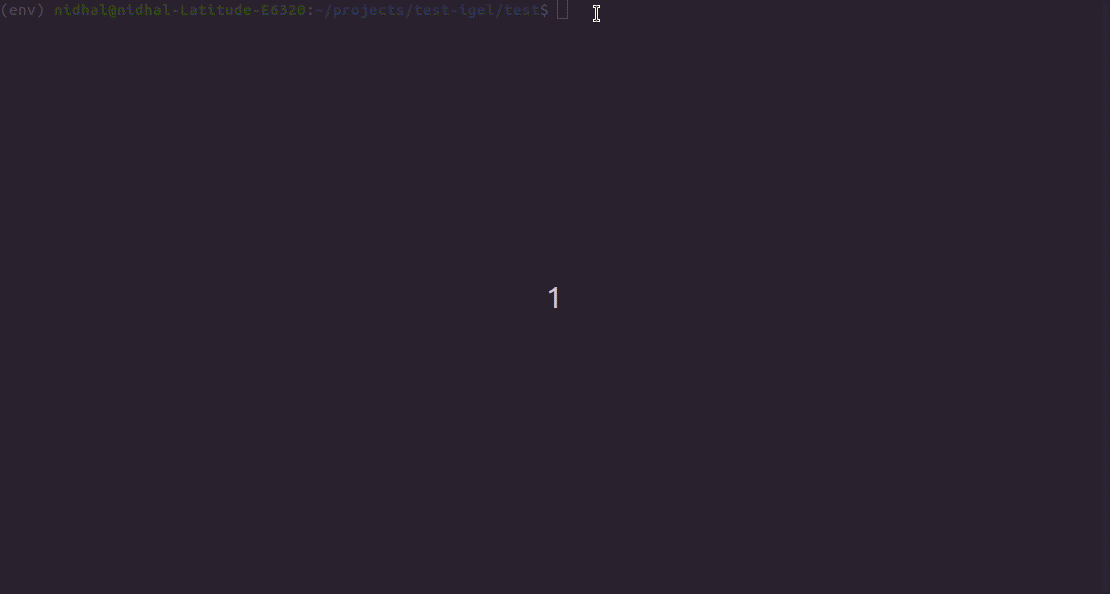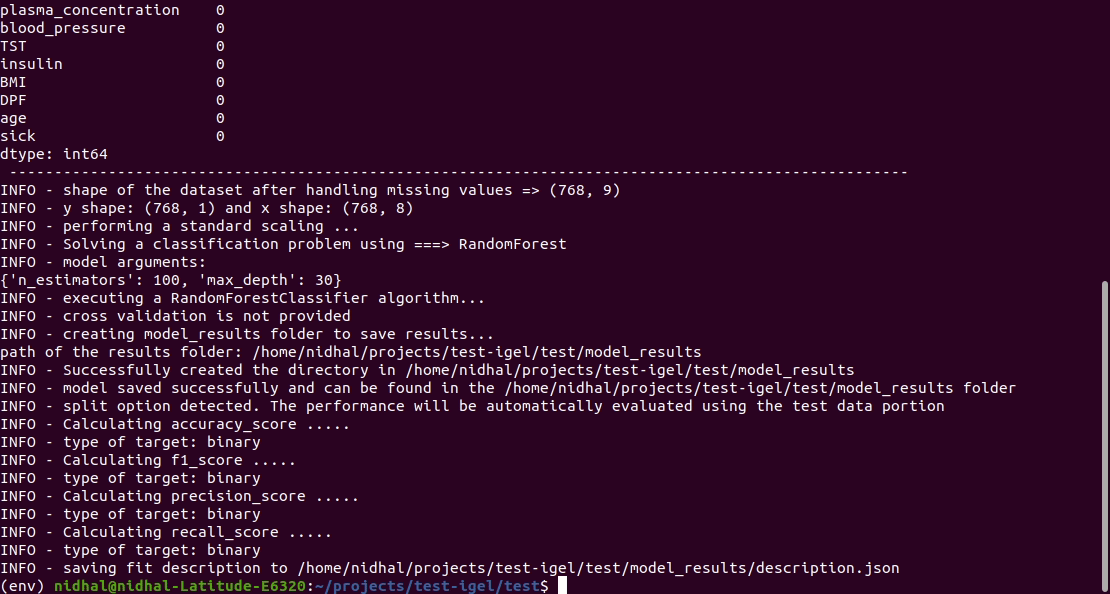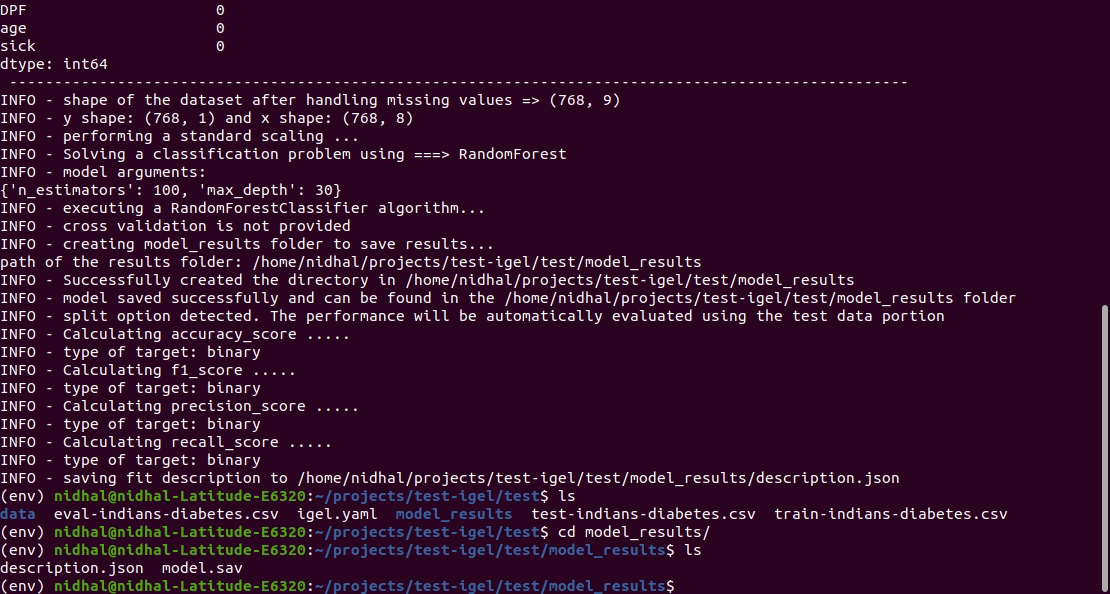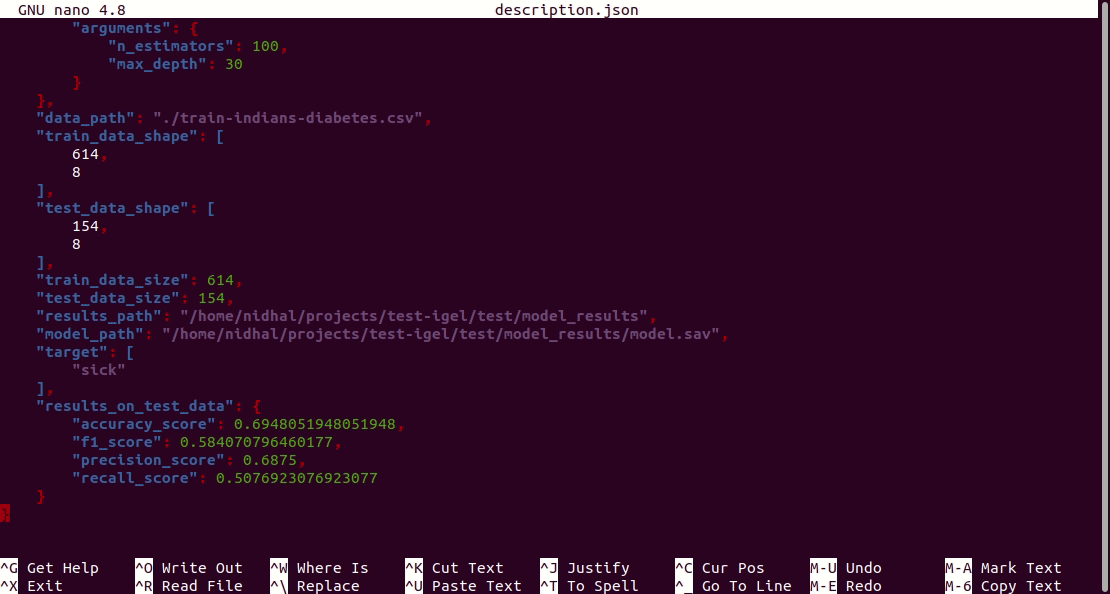
Furthermore, a description can be found in the description.json file inside the model_results folder.
"""
Vous pouvez ensuite évaluer le modèle entraîné/pré-ajusté :
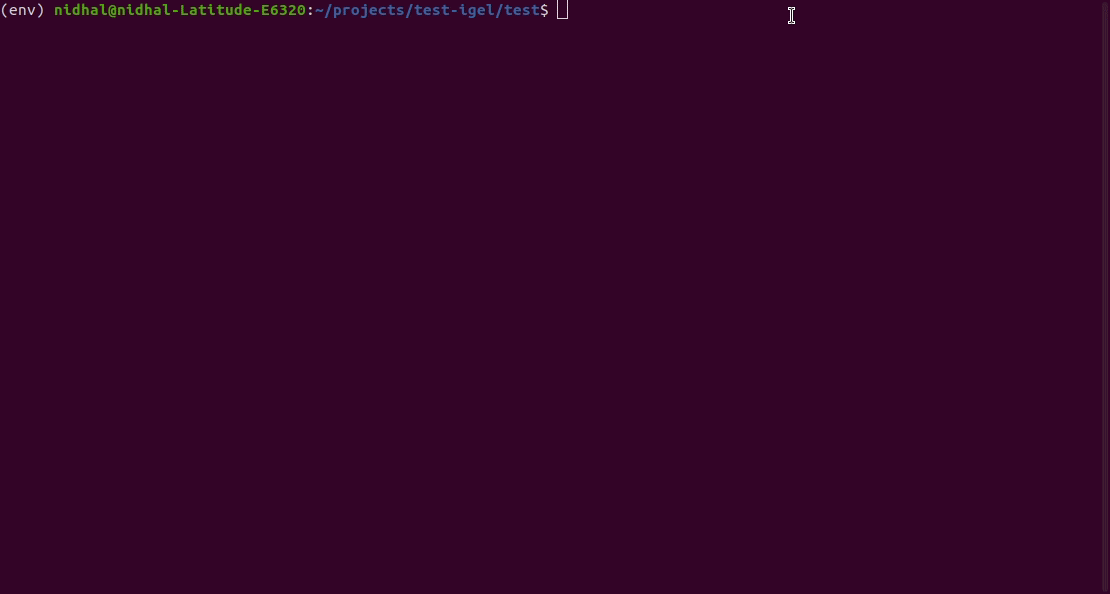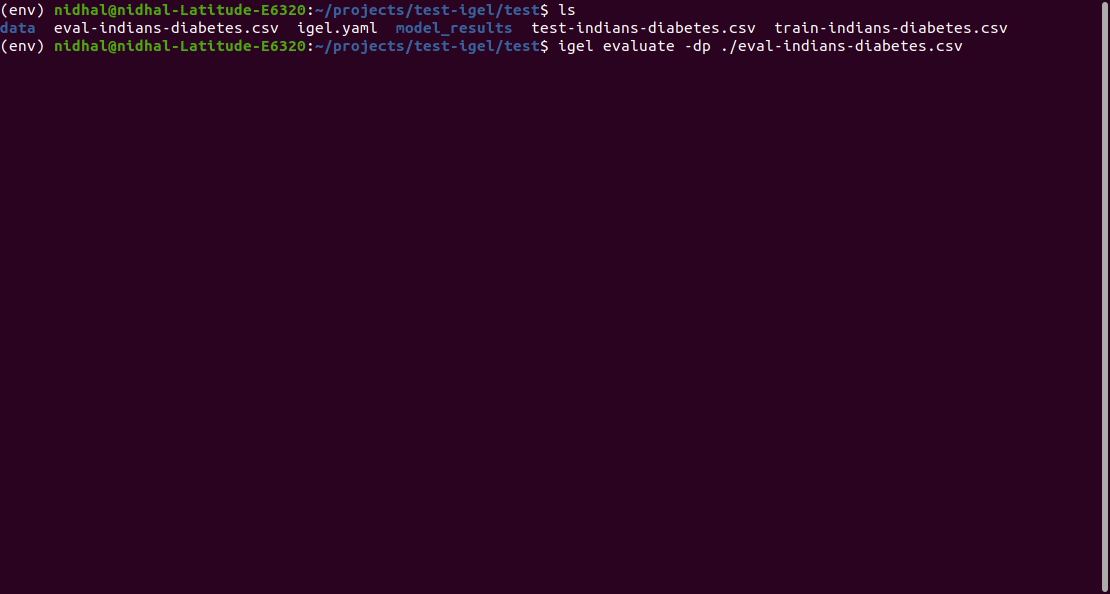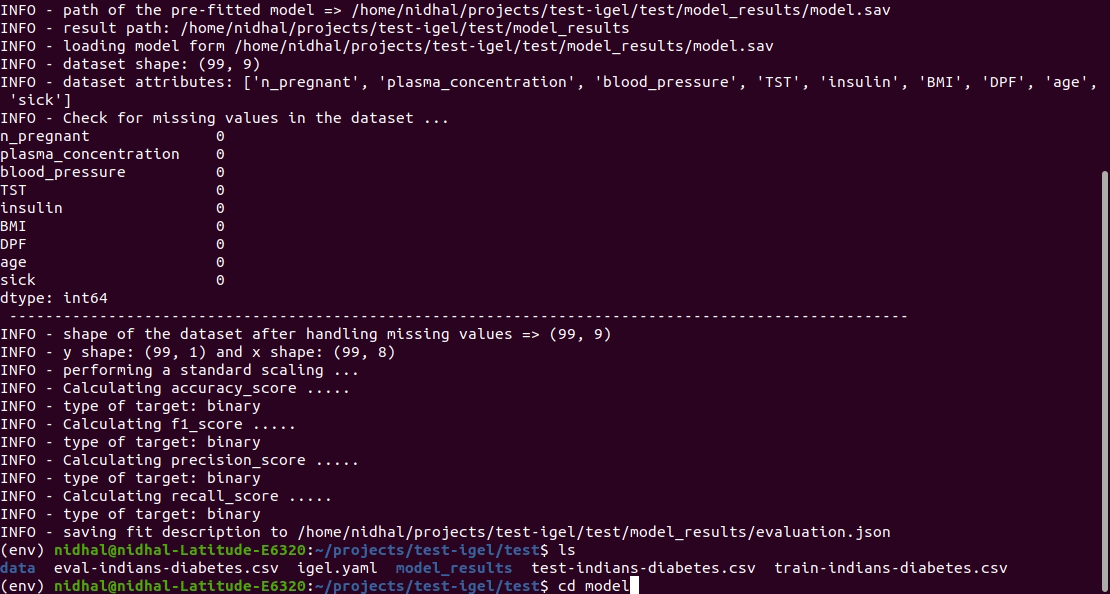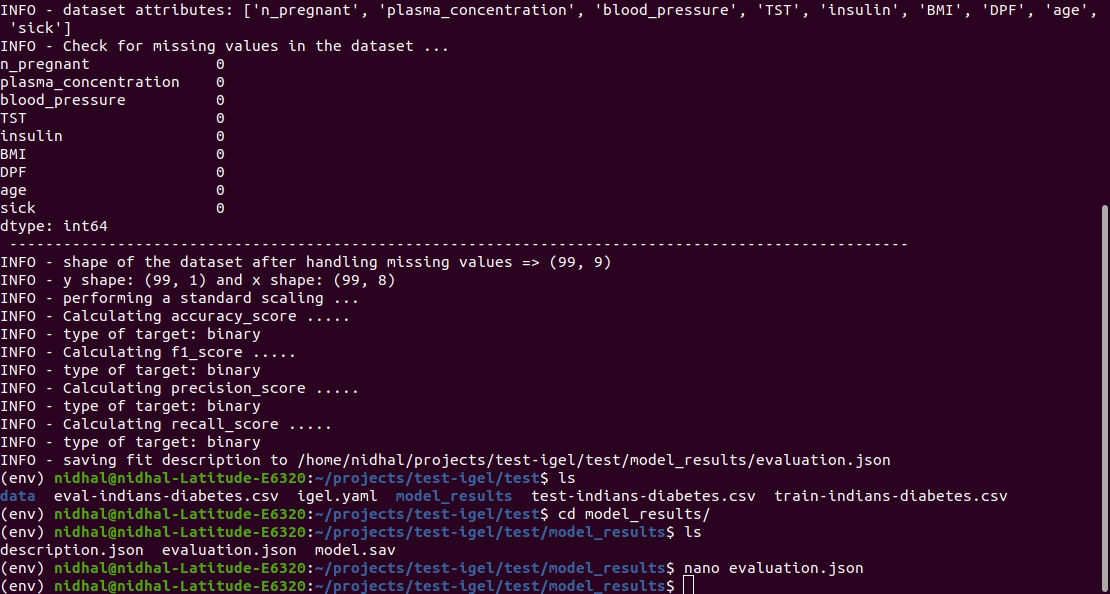
$ igel evaluate -dp ' path_to_your_evaluation_dataset.csv '
"""
This will automatically generate an evaluation.json file in the current directory, where all evaluation results are stored
"""
Enfin, vous pouvez utiliser le modèle entraîné/pré-ajusté pour faire des prédictions si vous êtes satisfait des résultats de l'évaluation :
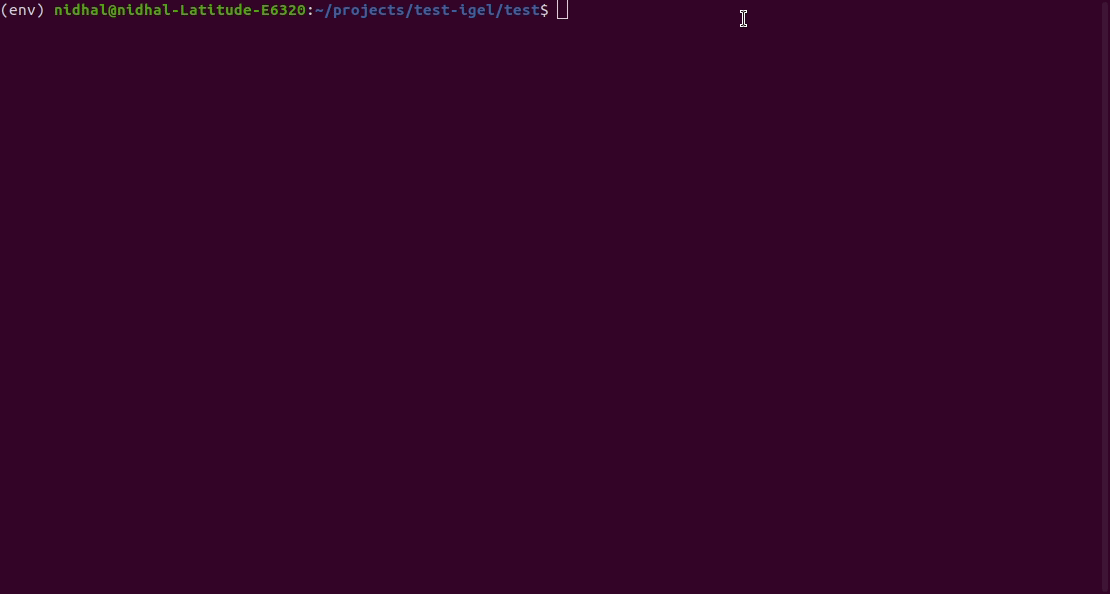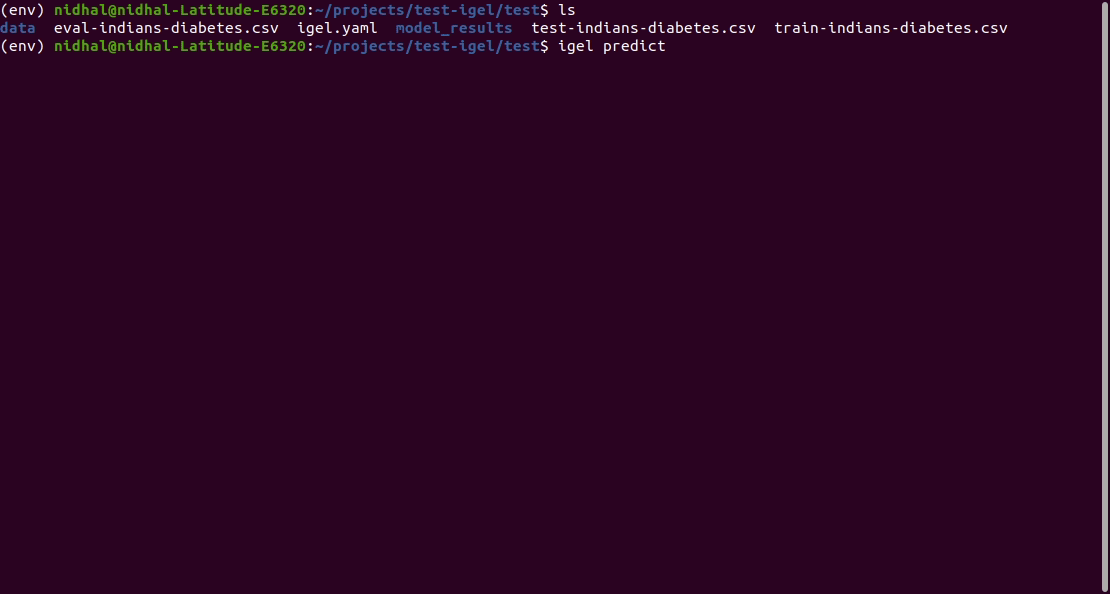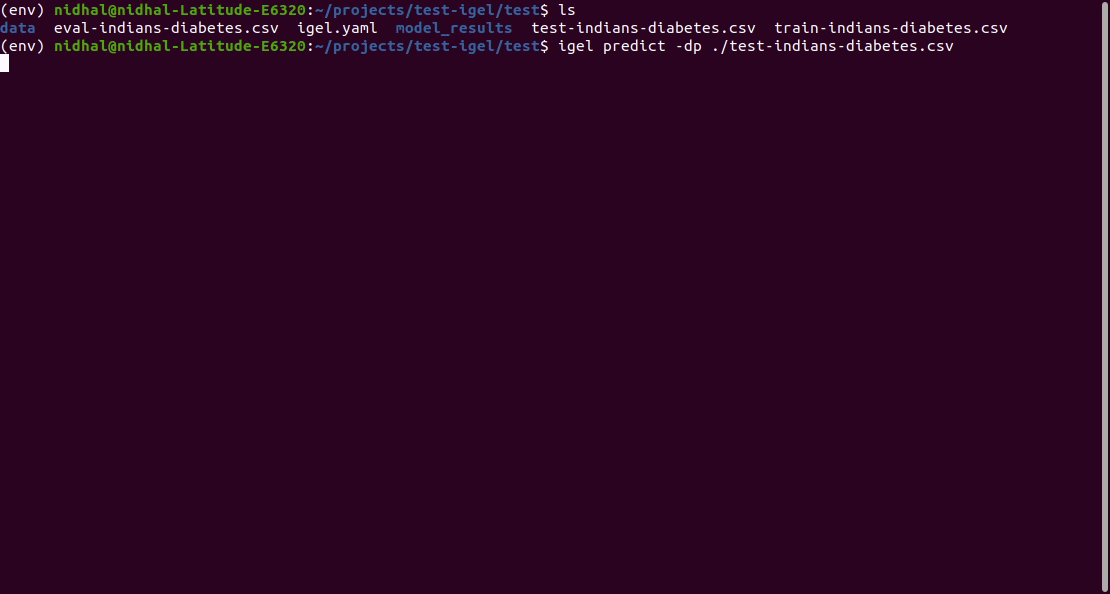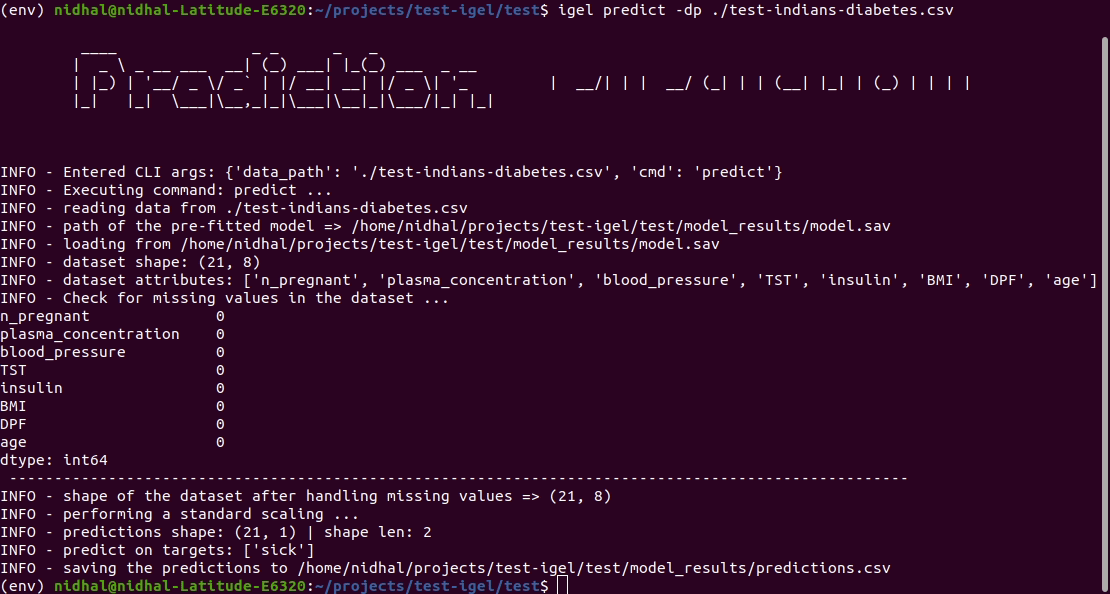
$ igel predict -dp ' path_to_your_test_dataset.csv '
"""
This will generate a predictions.csv file in your current directory, where all predictions are stored in a csv file
"""
Vous pouvez combiner les phases d'entraînement, d'évaluation et de prédiction à l'aide d'une seule commande appelée expérience :
$ igel experiment -DP " path_to_train_data path_to_eval_data path_to_test_data " -yml " path_to_yaml_file "
"""
This will run fit using train_data, evaluate using eval_data and further generate predictions using the test_data
"""
Vous pouvez exporter le modèle sklearn entraîné/pré-ajusté dans ONNX :
$ igel export -dp " path_to_pre-fitted_sklearn_model "
"""
This will convert the sklearn model into ONNX
""" from igel import Igel
Igel ( cmd = "fit" , data_path = "path_to_your_dataset" , yaml_path = "path_to_your_yaml_file" )
"""
check the examples folder for more
"""L'étape suivante consiste à utiliser votre modèle en production. Igel vous aide également dans cette tâche en fournissant la commande serve. L'exécution de la commande serve indiquera à igel de servir votre modèle. Plus précisément, igel construira automatiquement un serveur REST et servira votre modèle sur un hôte et un port spécifiques, que vous pourrez configurer en les transmettant en tant qu'options cli.
Le moyen le plus simple est d'exécuter :
$ igel serve --model_results_dir " path_to_model_results_directory "Notez qu'igel a besoin de l'option cli --model_results_dir ou bientôt -res_dir pour charger le modèle et démarrer le serveur. Par défaut, igel servira votre modèle sur localhost:8000 , cependant, vous pouvez facilement remplacer cela en fournissant des options d'hôte et de port cli.
$ igel serve --model_results_dir " path_to_model_results_directory " --host " 127.0.0.1 " --port 8000Igel utilise FastAPI pour créer le serveur REST, qui est un framework moderne haute performance et uvicorn pour l'exécuter sous le capot.
Cet exemple a été réalisé à l'aide d'un modèle pré-entraîné (créé en exécutant igel init --target malade -type classification) et l'ensemble de données sur le diabète indien sous exemples/données. Les en-têtes des colonnes dans le CSV d'origine sont « preg », « plas », « pres », « skin », « test », « mass », « pedi » et « age ».
BOUCLE:
$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": 1, "plas": 180, "pres": 50, "skin": 12, "test": 1, "mass": 456, "pedi": 0.442, "age": 50} '
Outputs: {"prediction":[[0.0]]}$ curl -X POST localhost:8080/predict --header " Content-Type:application/json " -d ' {"preg": [1, 6, 10], "plas":[192, 52, 180], "pres": [40, 30, 50], "skin": [25, 35, 12], "test": [0, 1, 1], "mass": [456, 123, 155], "pedi": [0.442, 0.22, 0.19], "age": [50, 40, 29]} '
Outputs: {"prediction":[[1.0],[0.0],[0.0]]}Mises en garde/limites :
Exemple d'utilisation du client Python :
from python_client import IgelClient
# the client allows additional args with defaults:
# scheme="http", endpoint="predict", missing_values="mean"
client = IgelClient ( host = 'localhost' , port = 8080 )
# you can post other types of files compatible with what Igel data reading allows
client . post ( "my_batch_file_for_predicting.csv" )
Outputs : < Response 200 > : { "prediction" :[[ 1.0 ],[ 0.0 ],[ 0.0 ]]}L'objectif principal d'igel est de vous fournir un moyen de former/adapter, évaluer et utiliser des modèles sans écrire de code. Au lieu de cela, tout ce dont vous avez besoin est de fournir/décrire ce que vous voulez faire dans un simple fichier yaml.
Fondamentalement, vous fournissez une description ou plutôt des configurations dans le fichier yaml sous forme de paires clé-valeur. Voici un aperçu de toutes les configurations prises en charge (pour l'instant) :
# dataset operations
dataset :
type : csv # [str] -> type of your dataset
read_data_options : # options you want to supply for reading your data (See the detailed overview about this in the next section)
sep : # [str] -> Delimiter to use.
delimiter : # [str] -> Alias for sep.
header : # [int, list of int] -> Row number(s) to use as the column names, and the start of the data.
names : # [list] -> List of column names to use
index_col : # [int, str, list of int, list of str, False] -> Column(s) to use as the row labels of the DataFrame,
usecols : # [list, callable] -> Return a subset of the columns
squeeze : # [bool] -> If the parsed data only contains one column then return a Series.
prefix : # [str] -> Prefix to add to column numbers when no header, e.g. ‘X’ for X0, X1, …
mangle_dupe_cols : # [bool] -> Duplicate columns will be specified as ‘X’, ‘X.1’, …’X.N’, rather than ‘X’…’X’. Passing in False will cause data to be overwritten if there are duplicate names in the columns.
dtype : # [Type name, dict maping column name to type] -> Data type for data or columns
engine : # [str] -> Parser engine to use. The C engine is faster while the python engine is currently more feature-complete.
converters : # [dict] -> Dict of functions for converting values in certain columns. Keys can either be integers or column labels.
true_values : # [list] -> Values to consider as True.
false_values : # [list] -> Values to consider as False.
skipinitialspace : # [bool] -> Skip spaces after delimiter.
skiprows : # [list-like] -> Line numbers to skip (0-indexed) or number of lines to skip (int) at the start of the file.
skipfooter : # [int] -> Number of lines at bottom of file to skip
nrows : # [int] -> Number of rows of file to read. Useful for reading pieces of large files.
na_values : # [scalar, str, list, dict] -> Additional strings to recognize as NA/NaN.
keep_default_na : # [bool] -> Whether or not to include the default NaN values when parsing the data.
na_filter : # [bool] -> Detect missing value markers (empty strings and the value of na_values). In data without any NAs, passing na_filter=False can improve the performance of reading a large file.
verbose : # [bool] -> Indicate number of NA values placed in non-numeric columns.
skip_blank_lines : # [bool] -> If True, skip over blank lines rather than interpreting as NaN values.
parse_dates : # [bool, list of int, list of str, list of lists, dict] -> try parsing the dates
infer_datetime_format : # [bool] -> If True and parse_dates is enabled, pandas will attempt to infer the format of the datetime strings in the columns, and if it can be inferred, switch to a faster method of parsing them.
keep_date_col : # [bool] -> If True and parse_dates specifies combining multiple columns then keep the original columns.
dayfirst : # [bool] -> DD/MM format dates, international and European format.
cache_dates : # [bool] -> If True, use a cache of unique, converted dates to apply the datetime conversion.
thousands : # [str] -> the thousands operator
decimal : # [str] -> Character to recognize as decimal point (e.g. use ‘,’ for European data).
lineterminator : # [str] -> Character to break file into lines.
escapechar : # [str] -> One-character string used to escape other characters.
comment : # [str] -> Indicates remainder of line should not be parsed. If found at the beginning of a line, the line will be ignored altogether. This parameter must be a single character.
encoding : # [str] -> Encoding to use for UTF when reading/writing (ex. ‘utf-8’).
dialect : # [str, csv.Dialect] -> If provided, this parameter will override values (default or not) for the following parameters: delimiter, doublequote, escapechar, skipinitialspace, quotechar, and quoting
delim_whitespace : # [bool] -> Specifies whether or not whitespace (e.g. ' ' or ' ') will be used as the sep
low_memory : # [bool] -> Internally process the file in chunks, resulting in lower memory use while parsing, but possibly mixed type inference.
memory_map : # [bool] -> If a filepath is provided for filepath_or_buffer, map the file object directly onto memory and access the data directly from there. Using this option can improve performance because there is no longer any I/O overhead.
random_numbers : # random numbers options in case you wanted to generate the same random numbers on each run
generate_reproducible : # [bool] -> set this to true to generate reproducible results
seed : # [int] -> the seed number is optional. A seed will be set up for you if you didn't provide any
split : # split options
test_size : 0.2 # [float] -> 0.2 means 20% for the test data, so 80% are automatically for training
shuffle : true # [bool] -> whether to shuffle the data before/while splitting
stratify : None # [list, None] -> If not None, data is split in a stratified fashion, using this as the class labels.
preprocess : # preprocessing options
missing_values : mean # [str] -> other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # [str] -> other possible values: [labelEncoding]
scale : # scaling options
method : standard # [str] -> standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # [str] -> scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification # [str] -> type of the problem you want to solve. | possible values: [regression, classification, clustering]
algorithm : NeuralNetwork # [str (notice the pascal case)] -> which algorithm you want to use. | type igel algorithms in the Terminal to know more
arguments : # model arguments: you can check the available arguments for each model by running igel help in your terminal
use_cv_estimator : false # [bool] -> if this is true, the CV class of the specific model will be used if it is supported
cross_validate :
cv : # [int] -> number of kfold (default 5)
n_jobs : # [signed int] -> The number of CPUs to use to do the computation (default None)
verbose : # [int] -> The verbosity level. (default 0)
hyperparameter_search :
method : grid_search # method you want to use: grid_search and random_search are supported
parameter_grid : # put your parameters grid here that you want to use, an example is provided below
param1 : [val1, val2]
param2 : [val1, val2]
arguments : # additional arguments you want to provide for the hyperparameter search
cv : 5 # number of folds
refit : true # whether to refit the model after the search
return_train_score : false # whether to return the train score
verbose : 0 # verbosity level
# target you want to predict
target : # list of strings: basically put here the column(s), you want to predict that exist in your csv dataset
- put the target you want to predict here
- you can assign many target if you are making a multioutput prediction Note
igel utilise des pandas sous le capot pour lire et analyser les données. Par conséquent, vous pouvez également trouver ces paramètres facultatifs de données dans la documentation officielle des pandas.
Un aperçu détaillé des configurations que vous pouvez fournir dans le fichier yaml (ou json) est donné ci-dessous. Notez que vous n'aurez certainement pas besoin de toutes les valeurs de configuration pour l'ensemble de données. Ils sont facultatifs. En général, igel découvrira comment lire votre ensemble de données.
Cependant, vous pouvez l'aider en fournissant des champs supplémentaires à l'aide de cette section read_data_options. Par exemple, l'une des valeurs utiles à mon avis est le "sep", qui définit la façon dont vos colonnes dans l'ensemble de données CSV sont séparées. Généralement, les ensembles de données CSV sont séparés par des virgules, qui sont également la valeur par défaut ici. Cependant, il peut être séparé par un point-virgule dans votre cas.
Par conséquent, vous pouvez fournir cela dans read_data_options. Ajoutez simplement le sep: ";" sous read_data_options.
| Paramètre | Taper | Explication |
|---|---|---|
| sep | str, par défaut ',' | Délimiteur à utiliser. Si sep vaut None, le moteur C ne peut pas détecter automatiquement le séparateur, mais le moteur d'analyse Python le peut, ce qui signifie que ce dernier sera utilisé et détectera automatiquement le séparateur par l'outil de renifleur intégré de Python, csv.Sniffer. De plus, les séparateurs de plus d'un caractère et différents de 's+' seront interprétés comme des expressions régulières et forceront également l'utilisation du moteur d'analyse Python. Notez que les délimiteurs d’expressions régulières ont tendance à ignorer les données citées. Exemple d'expression régulière : 'rt'. |
| délimiteur | par défaut Aucun | Alias pour septembre. |
| en-tête | int, liste d'int, 'infer' par défaut | Numéro(s) de ligne à utiliser comme noms de colonne et début des données. Le comportement par défaut consiste à déduire les noms de colonnes : si aucun nom n'est transmis, le comportement est identique à header=0 et les noms de colonnes sont déduits de la première ligne du fichier, si les noms de colonnes sont transmis explicitement, alors le comportement est identique à header=None. . Passez explicitement header=0 pour pouvoir remplacer les noms existants. L'en-tête peut être une liste d'entiers qui spécifient les emplacements des lignes pour un multi-index sur les colonnes, par exemple [0,1,3]. Les lignes intermédiaires qui ne sont pas spécifiées seront ignorées (par exemple, 2 dans cet exemple sont ignorées). Notez que ce paramètre ignore les lignes commentées et les lignes vides si skip_blank_lines=True, donc header=0 désigne la première ligne de données plutôt que la première ligne du fichier. |
| noms | en forme de tableau, facultatif | Liste des noms de colonnes à utiliser. Si le fichier contient une ligne d'en-tête, vous devez alors explicitement transmettre header=0 pour remplacer les noms de colonnes. Les doublons dans cette liste ne sont pas autorisés. |
| index_col | int, str, séquence de int/str ou False, par défaut Aucun | Colonne(s) à utiliser comme étiquettes de ligne du DataFrame, soit données sous forme de nom de chaîne, soit d'index de colonne. Si une séquence de int/str est donnée, un MultiIndex est utilisé. Remarque : index_col=False peut être utilisé pour forcer les pandas à ne pas utiliser la première colonne comme index, par exemple lorsque vous avez un fichier mal formé avec des délimiteurs à la fin de chaque ligne. |
| utiliser les cols | sous forme de liste ou appelable, facultatif | Renvoie un sous-ensemble de colonnes. S'ils sont de type liste, tous les éléments doivent être soit positionnels (c'est-à-dire des indices entiers dans les colonnes du document), soit des chaînes qui correspondent aux noms de colonnes fournis soit par l'utilisateur dans les noms, soit déduits de la ou des lignes d'en-tête du document. Par exemple, un paramètre usecols valide de type liste serait [0, 1, 2] ou ['foo', 'bar', 'baz']. L'ordre des éléments est ignoré, donc usecols=[0, 1] est le même que [1, 0]. Pour instancier un DataFrame à partir de données avec l'ordre des éléments préservé, utilisez pd.read_csv(data, usecols=['foo', 'bar'])[['foo', 'bar']] pour les colonnes dans ['foo', 'bar '] commande ou pd.read_csv(data, usecols=['foo', 'bar'])[['bar', 'foo']] pour ['bar', 'foo'] ordre. Si elle est appelable, la fonction appelable sera évaluée par rapport aux noms de colonnes, renvoyant les noms où la fonction appelable est évaluée à True. Un exemple d'argument appelable valide serait lambda x : x.upper() dans ['AAA', 'BBB', 'DDD']. L’utilisation de ce paramètre entraîne un temps d’analyse beaucoup plus rapide et une utilisation moindre de la mémoire. |
| presser | bool, faux par défaut | Si les données analysées ne contiennent qu’une seule colonne, renvoyez une série. |
| préfixe | chaîne, facultatif | Préfixe à ajouter aux numéros de colonnes lorsqu'il n'y a pas d'en-tête, par exemple « X » pour X0, X1, … |
| mangle_dupe_cols | bool, vrai par défaut | Les colonnes en double seront spécifiées comme « X », « X.1 »,… « X.N », plutôt que « X »… « X ». Passer False entraînera l'écrasement des données s'il y a des noms en double dans les colonnes. |
| type | {'c', 'python'}, facultatif | Moteur d'analyseur à utiliser. Le moteur C est plus rapide tandis que le moteur Python est actuellement plus complet en fonctionnalités. |
| convertisseurs | dict, facultatif | Dicté de fonctions pour convertir les valeurs dans certaines colonnes. Les clés peuvent être des entiers ou des étiquettes de colonnes. |
| valeurs_vraies | liste, facultatif | Valeurs à considérer comme vraies. |
| fausses_valeurs | liste, facultatif | Valeurs à considérer comme fausses. |
| sauter l'espace initial | bool, faux par défaut | Ignorer les espaces après le délimiteur. |
| skiprows | de type liste, entier ou appelable, facultatif | Numéros de lignes à sauter (indexés 0) ou nombre de lignes à sauter (int) au début du fichier. Si elle est appelable, la fonction appelable sera évaluée par rapport aux indices de ligne, renvoyant True si la ligne doit être ignorée et False sinon. Un exemple d'argument appelable valide serait lambda x : x dans [0, 2]. |
| saut de pied | int, par défaut 0 | Nombre de lignes en bas du fichier à ignorer (Non pris en charge avec engine='c'). |
| nrows | entier, facultatif | Nombre de lignes du fichier à lire. Utile pour lire des morceaux de fichiers volumineux. |
| na_values | scalaire, str, de type liste ou dict, facultatif | Chaînes supplémentaires à reconnaître comme NA/NaN. Si le dict est réussi, valeurs NA spécifiques par colonne. Par défaut les valeurs suivantes sont interprétées comme NaN : '', '#N/A', '#N/AN/A', '#NA', '-1.#IND', '-1.#QNAN', '-NaN', '-nan', '1.#IND', '1.#QNAN', '<NA>', 'N/A', 'NA', 'NULL', « NaN », « n/a », « nan », « nul ». |
| keep_default_na | bool, vrai par défaut | S'il faut ou non inclure les valeurs NaN par défaut lors de l'analyse des données. Selon que na_values est transmis ou non, le comportement est le suivant : Si keep_default_na est True et que na_values sont spécifiés, na_values est ajouté aux valeurs NaN par défaut utilisées pour l'analyse. Si keep_default_na est True et que na_values ne sont pas spécifiés, seules les valeurs NaN par défaut sont utilisées pour l'analyse. Si keep_default_na est False et que na_values sont spécifiés, seules les valeurs NaN spécifiées na_values sont utilisées pour l'analyse. Si keep_default_na est False et que na_values n'est pas spécifié, aucune chaîne ne sera analysée comme NaN. Notez que si na_filter est transmis comme False, les paramètres keep_default_na et na_values seront ignorés. |
| na_filter | bool, vrai par défaut | Détectez les marqueurs de valeur manquants (chaînes vides et valeur de na_values). Dans les données sans aucun NA, passer na_filter=False peut améliorer les performances de lecture d'un fichier volumineux. |
| verbeux | bool, faux par défaut | Indiquez le nombre de valeurs NA placées dans des colonnes non numériques. |
| skip_blank_lines | bool, vrai par défaut | Si True, sautez les lignes vides plutôt que de les interpréter comme des valeurs NaN. |
| analyser_dates | bool ou liste d'int ou de noms ou liste de listes ou dict, valeur par défaut False | Le comportement est le suivant : booléen. Si True -> essayez d'analyser l'index. liste d'entiers ou de noms. Par exemple, If [1, 2, 3] -> essayez d'analyser les colonnes 1, 2, 3 chacune comme une colonne de date distincte. liste de listes. Par exemple, If [[1, 3]] -> combinez les colonnes 1 et 3 et analysez-les comme une seule colonne de date. dict, par exemple {'foo' : [1, 3]} -> analyser les colonnes 1, 3 comme date et appeler le résultat 'foo' Si une colonne ou un index ne peut pas être représenté comme un tableau de dates et d'heures, par exemple à cause d'une valeur non analysable ou un mélange de fuseaux horaires, la colonne ou l'index sera renvoyé inchangé en tant que type de données objet. |
| infer_datetime_format | bool, faux par défaut | Si True et parse_dates sont activés, les pandas tenteront de déduire le format des chaînes datetime dans les colonnes, et si cela peut être déduit, passeront à une méthode plus rapide pour les analyser. Dans certains cas, cela peut augmenter la vitesse d'analyse de 5 à 10 fois. |
| keep_date_col | bool, faux par défaut | Si True et parse_dates spécifient la combinaison de plusieurs colonnes, conservez les colonnes d'origine. |
| date_parser | fonction, en option | Fonction à utiliser pour convertir une séquence de colonnes de chaîne en un tableau d'instances datetime. La valeur par défaut utilise dateutil.parser.parser pour effectuer la conversion. Les pandas essaieront d'appeler date_parser de trois manières différentes, en passant à la suivante si une exception se produit : 1) Transmettez un ou plusieurs tableaux (tels que définis par parse_dates) comme arguments ; 2) concaténer (par ligne) les valeurs de chaîne des colonnes définies par parse_dates dans un seul tableau et le transmettre ; et 3) appeler date_parser une fois pour chaque ligne en utilisant une ou plusieurs chaînes (correspondant aux colonnes définies par parse_dates) comme arguments. |
| premier jour | bool, faux par défaut | Dates au format JJ/MM, format international et européen. |
| dates_cache | bool, vrai par défaut | Si True, utilisez un cache de dates converties uniques pour appliquer la conversion datetime. Peut produire une accélération significative lors de l’analyse des chaînes de date en double, en particulier celles avec des décalages de fuseau horaire. |
| des milliers | chaîne, facultatif | Séparateur de milliers. |
| décimal | str, par défaut '.' | Caractère à reconnaître comme point décimal (par exemple utiliser ',' pour les données européennes). |
| terminateur de ligne | str (longueur 1), facultatif | Caractère pour diviser le fichier en lignes. Uniquement valable avec l'analyseur C. |
| char d'échappement | str (longueur 1), facultatif | Chaîne d'un caractère utilisée pour échapper d'autres caractères. |
| commentaire | chaîne, facultatif | Indique que le reste de la ligne ne doit pas être analysé. Si elle est trouvée au début d'une ligne, la ligne sera complètement ignorée. |
| codage | chaîne, facultatif | Encodage à utiliser pour UTF lors de la lecture/écriture (ex. 'utf-8'). |
| dialecte | str ou csv.Dialect, facultatif | S'il est fourni, ce paramètre remplacera les valeurs (par défaut ou non) des paramètres suivants : délimiteur, doublequote, escapechar, skipinitialspace, quotechar et quoting. |
| mémoire_basse | bool, vrai par défaut | Traitez le fichier en interne par morceaux, ce qui entraîne une utilisation moindre de la mémoire lors de l'analyse, mais éventuellement une inférence de type mixte. Pour garantir qu'il n'y a pas de types mixtes, définissez False ou spécifiez le type avec le paramètre dtype. Notez que le fichier entier est lu dans un seul DataFrame, |
| carte_mémoire | bool, faux par défaut | mappez l’objet fichier directement sur la mémoire et accédez aux données directement à partir de là. L’utilisation de cette option peut améliorer les performances car il n’y a plus de surcharge d’E/S. |
Une solution complète de bout en bout est fournie dans cette section pour prouver les capacités d' igel . Comme expliqué précédemment, vous devez créer un fichier de configuration yaml. Voici un exemple de bout en bout pour prédire si une personne souffre de diabète ou non en utilisant l'algorithme de l'arbre de décision . L'ensemble de données se trouve dans le dossier des exemples.
model :
type : classification
algorithm : DecisionTree
target :
- sick $ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_fileVoilà, igel va maintenant adapter le modèle pour vous et l'enregistrer dans un dossier model_results de votre répertoire actuel.
Évaluez le modèle pré-ajusté. Igel chargera le modèle pré-ajusté à partir du répertoire model_results et l'évaluera pour vous. Il vous suffit d'exécuter la commande d'évaluation et de fournir le chemin d'accès à vos données d'évaluation.
$ igel evaluate -dp path_to_the_evaluation_datasetC'est ça! Igel évaluera le modèle et stockera les statistiques/résultats dans un fichier évaluation.json dans le dossier model_results
Utilisez le modèle pré-ajusté pour prédire sur de nouvelles données. Ceci est fait automatiquement par igel, il vous suffit de fournir le chemin d'accès à vos données sur lesquelles vous souhaitez utiliser la prédiction.
$ igel predict -dp path_to_the_new_datasetC'est ça! Igel utilisera le modèle pré-ajusté pour faire des prédictions et l'enregistrera dans un fichier prédictions.csv dans le dossier model_results
Vous pouvez également effectuer certaines méthodes de prétraitement ou d'autres opérations en les fournissant dans le fichier yaml. Voici un exemple, où les données sont réparties à 80 % pour la formation et à 20 % pour la validation/les tests. De plus, les données sont mélangées lors du fractionnement.
De plus, les données sont prétraitées en remplaçant les valeurs manquantes par la moyenne (vous pouvez également utiliser la médiane, le mode etc.). consultez ce lien pour plus d'informations
# dataset operations
dataset :
split :
test_size : 0.2
shuffle : True
stratify : default
preprocess : # preprocessing options
missing_values : mean # other possible values: [drop, median, most_frequent, constant] check the docs for more
encoding :
type : oneHotEncoding # other possible values: [labelEncoding]
scale : # scaling options
method : standard # standardization will scale values to have a 0 mean and 1 standard deviation | you can also try minmax
target : inputs # scale inputs. | other possible values: [outputs, all] # if you choose all then all values in the dataset will be scaled
# model definition
model :
type : classification
algorithm : RandomForest
arguments :
# notice that this is the available args for the random forest model. check different available args for all supported models by running igel help
n_estimators : 100
max_depth : 20
# target you want to predict
target :
- sickEnsuite, vous pouvez ajuster le modèle en exécutant la commande igel comme indiqué dans les autres exemples
$ igel fit -dp path_to_the_dataset -yml path_to_the_yaml_filePour évaluation
$ igel evaluate -dp path_to_the_evaluation_datasetPour la production
$ igel predict -dp path_to_the_new_dataset Dans le dossier d'exemples du référentiel, vous trouverez un dossier de données, où sont stockés les célèbres ensembles de données sur le diabète indien, l'iris et les ensembles de données linnerud (de sklearn). De plus, il y a des exemples de bout en bout dans chaque dossier, où se trouvent des scripts et des fichiers yaml qui vous aideront à démarrer.
Le dossier indian-diabetes-example contient deux exemples pour vous aider à démarrer :
Le dossier iris-example contient un exemple de régression logistique , où un prétraitement (un encodage à chaud) est effectué sur la colonne cible pour vous montrer davantage les capacités d'igel.
De plus, l'exemple multi-sorties contient un exemple de régression multi-sorties . Enfin, l'exemple cv contient un exemple utilisant le classificateur Ridge utilisant la validation croisée.
Vous pouvez également trouver des exemples de validation croisée et de recherche d'hyperparamètres dans le dossier.
Je vous suggère de jouer avec les exemples et igel cli. Cependant, vous pouvez également exécuter directement fit.py, évalue.py et prédire.py si vous le souhaitez.
Tout d'abord, créez ou modifiez un ensemble de données d'images classées en sous-dossiers en fonction de l'étiquette/classe de l'image. Par exemple, si vous avez des images de chiens et de chats, vous aurez besoin de 2 sous-dossiers :
En supposant que ces deux sous-dossiers sont contenus dans un dossier parent appelé images, transmettez simplement les données à igel :
$ igel auto-train -dp ./images --task ImageClassificationIgel s'occupera de tout, du prétraitement des données à l'optimisation des hyperparamètres. A la fin, le meilleur modèle sera stocké dans le répertoire de travail actuel.
Tout d'abord, créez ou modifiez un ensemble de données texte classé en sous-dossiers en fonction de l'étiquette/classe de texte. Par exemple, si vous disposez d'un ensemble de données texte contenant des commentaires positifs et négatifs, vous aurez besoin de 2 sous-dossiers :
En supposant que ces deux sous-dossiers soient contenus dans un dossier parent appelé textes, transmettez simplement les données à igel :
$ igel auto-train -dp ./texts --task TextClassificationIgel s'occupera de tout, du prétraitement des données à l'optimisation des hyperparamètres. A la fin, le meilleur modèle sera stocké dans le répertoire de travail actuel.
Vous pouvez également exécuter l'interface utilisateur igel si vous n'êtes pas familier avec le terminal. Installez simplement igel sur votre machine comme mentionné ci-dessus. Ensuite, exécutez cette seule commande dans votre terminal
$ igel guiCela ouvrira l'interface graphique, qui est très simple à utiliser. Vérifiez des exemples de l'apparence de l'interface graphique et comment l'utiliser ici : https://github.com/nidhaloff/igel-ui
Vous pouvez d'abord extraire l'image du hub Docker
$ docker pull nidhaloff/igelAlors utilisez-le :
$ docker run -it --rm -v $( pwd ) :/data nidhaloff/igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv 'Vous pouvez exécuter igel à l'intérieur de Docker en créant d'abord l'image :
$ docker build -t igel .Et puis exécutez-le et attachez votre répertoire actuel (il n'est pas nécessaire que ce soit le répertoire igel) en tant que /data (le répertoire de travail) à l'intérieur du conteneur :
$ docker run -it --rm -v $( pwd ) :/data igel fit -yml ' your_file.yaml ' -dp ' your_dataset.csv ' Si vous rencontrez des problèmes, n'hésitez pas à ouvrir un problème. De plus, vous pouvez prendre contact avec l'auteur pour plus d'informations/questions.
Aimez-vous Igel? Vous pouvez toujours aider au développement de ce projet en :
Vous pensez que ce projet est utile et vous souhaitez apporter de nouvelles idées, de nouvelles fonctionnalités, des corrections de bugs, étendre la doc ?
Les contributions sont toujours les bienvenues. Assurez-vous de lire d'abord les directives
Licence MIT
Copyright (c) 2020-présent, Nidhal Baccouri