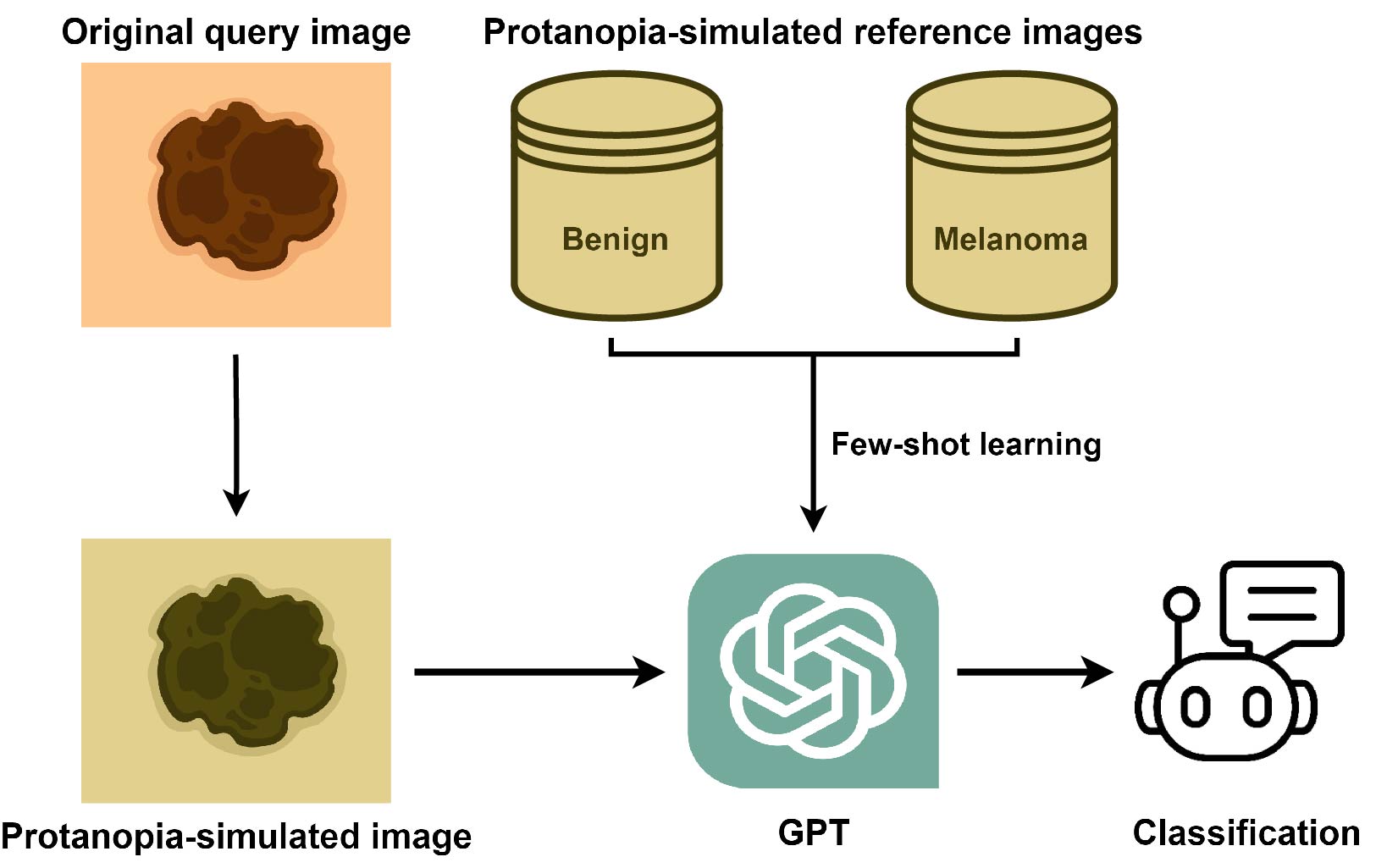
Ce référentiel contient du code et des données pour effectuer une classification dermoscopique accommodante de la vision des couleurs avec GPT-4V.
Lire le journal
Veuillez trouver la réponse détaillée du modèle GPT Formulaire Mendeley Data
nombant
Pilote
daltonlens
json
Openai
base64
pandas
. ├── data # Contains subdirectories for processed data │ ├── all │ ├── all_resized │ ├── all_resized_brettel_protan_1 │ ├── all_resized_brettel_deutan_1 │ ├── all_resized_brettel_tritan_1 │ ├── bn │ ├── bn_resized │ ├── bn_resized_label │ ├── bn_resized_label_brettel_protan_1 │ ├── bn_resized_label_brettel_deutan_1 │ ├── bn_resized_label_brettel_tritan_1 │ ├── mm │ ├── mm_resized │ ├── mm_resized_label │ ├── mm_resized_label_brettel_protan_1 │ ├── mm_resized_label_brettel_deutan_1 │ ├── mm_resized_label_brettel_tritan_1 │ └── selected_images.xlsx # Image names ├── RAW # Contains raw data downloaded from ISCI Archive ├── result # Results from running API_calling will be stored here │ ├── 2_shot_brettel_protan_1 │ │ ├──rep 1 │ │ └── ... │ ├── 2_shot_brettel_deutan_1 │ └── ... ├── CVD_classification_GPT.py # Call OpenAI API for classification ├── CVD_convertor.py # Convert original image to CVD simulated image ├── data_labeling.py # Add label to the image for reference ├── data_resizing.py # Resize the data with the original aspect ratio ├── data_selection.py # Select data from RAW └── README.md
Toutes les images dermoscopiques sont téléchargées à partir de l'archive ISIC.
Ce projet est uniquement à des fins de recherche académique. Le code de ce référentiel est publié sous la licence MIT. Si vous utilisez les données fournies, veuillez citer les archives ISIC.S
Pour la simulation CVD, nous avons choisi le package Daltonlens-Python. Il a couvert une variété des algorithmes actuellement disponibles de la simulation de daltonisme. Le blog de l'auteur vaut également la peine d'être lu.
Nous avons sélectionné Brettel et al. 1997 en tant que méthode de simulation CVD et définissez la gravité à 1, qui peut être ajustée avec le code fourni au besoin.
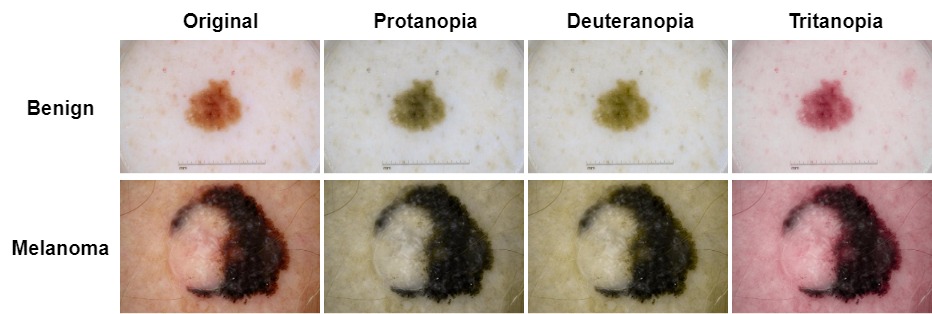
Exemple bénin: ISIC_0012656
Exemple de mélanome: ISIC_0046725
Téléchargez les données brutes de l'archive ISIC
Sélectionnez des données:
Exécutez python data_selection.py - [Options]
Données de processus:
Exécutez python data_resizeing.py - [Options]
Exécutez Python Data_Labeling.py - [Options]
Convertir les données:
Exécutez Python CVD_Convertor.py - [Options]
Appel API:
Exécutez python cvd_classification_gpt.py - [Options]
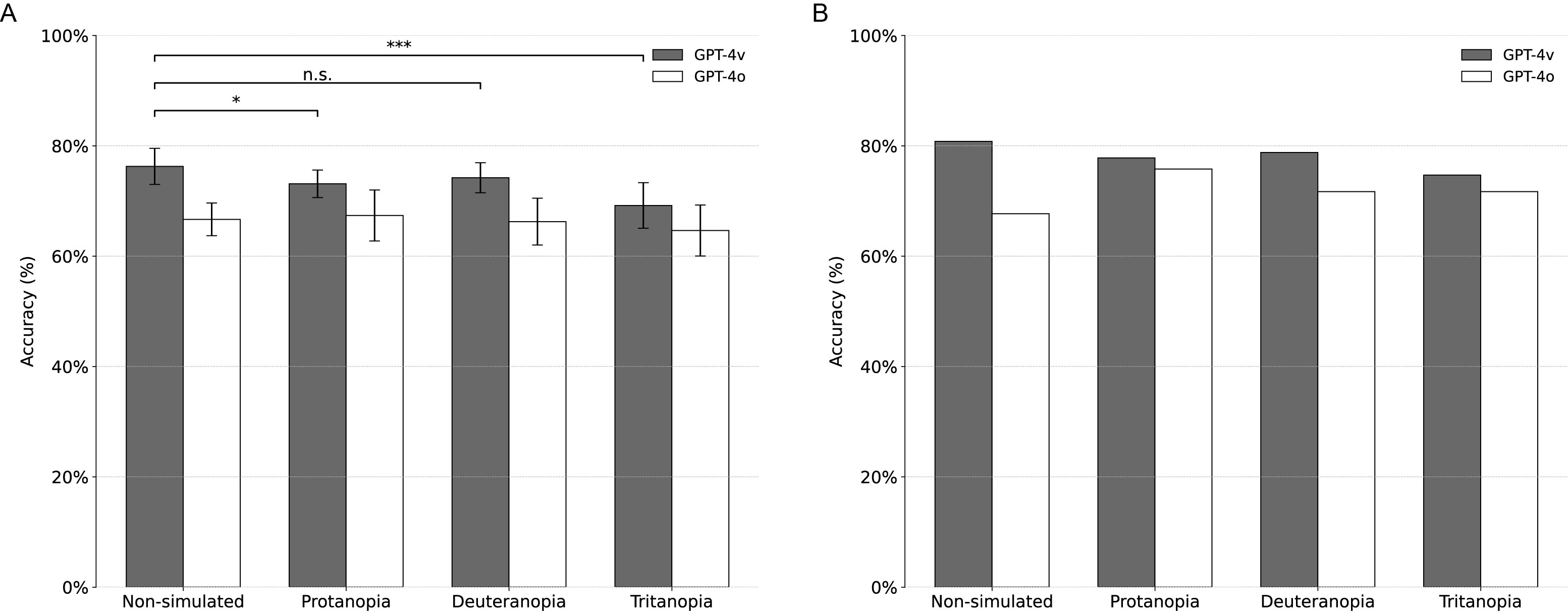
(A) Précision de classification moyenne de GPT-4V pour les images non simulées et simulées par CVD (protanopie, deutéanopie, tritanopie). Barres d'erreur: écarts-types. *: P <0,05; **: P <0,01 (test t; bilatéral). NS: Pas significatif. Toutes les expériences étaient dans dix répétitions. (B) Précision de classification de GPT-4V suite à l'application de la stratégie de consensus à travers les dix répétitions pour chaque requête d'image. GPT-4O inclus pour comparaison.
GPT-4V a adapté son interprétation sur les résultats basés sur les couleurs à différentes simulations de MCV. Plus précisément, il n'a pas mentionné les couleurs rouges ou roses dans les simulations Protanope et Deuteranope, mais a associé ces couleurs à une prédiction de mélanome dans des images non simulées. Dans les simulations de Tritanopie - confrontées à des teintes rosées répandues - le GPT-4V n'est plus traité rose comme une caractéristique de mélanome.
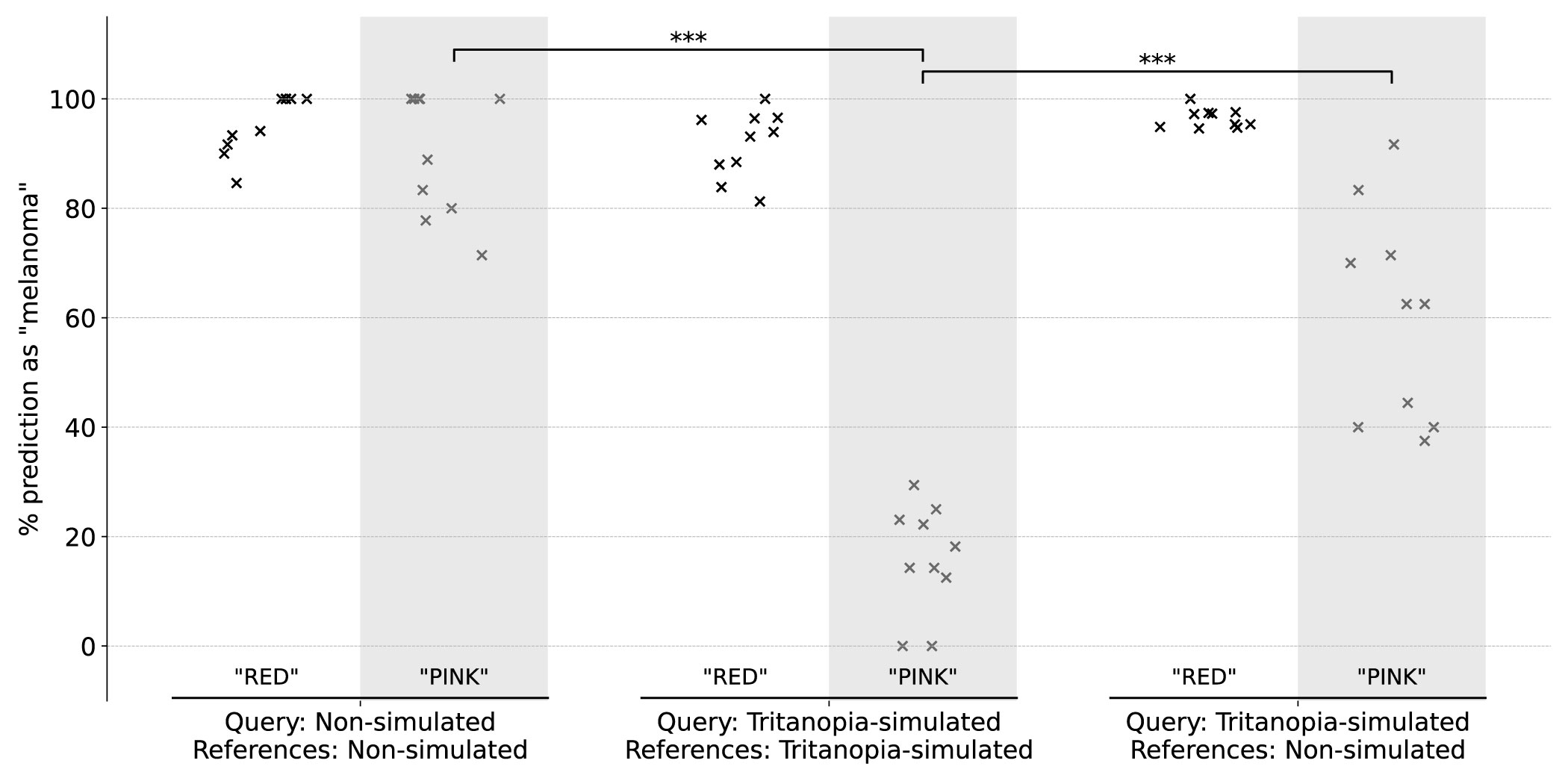
Pour chacune des conditions non simulées et simululées par Tritanopia, deux groupes de demandes d'image ont été identifiés sur la base des explications de GPT-4V pour ses prédictions. Le premier groupe, étiqueté «rouge», comprend des descriptions où la couleur rouge est mentionnée pour décrire l'image de la requête, quelle que soit la couleur rose. Le deuxième groupe, étiqueté «rose», comprend des descriptions mentionnant exclusivement le rose sans rouge. L'axe y affiche le pourcentage de prédictions de «mélanome» de chaque réplique. Par défaut, les images de requête et de référence étaient non simulées ou simulées dans la même condition CVD. Un test d'ablation (les deux dernières colonnes) a été effectué pour la simulation Tritanopia en échantillonnant les références d'images non simulées. ***: p <0,001 (test t; bilatéral).