Ini adalah gudang resmi untuk "Satu Model untuk Mengatur Semuanya: Menuju Segmentasi Universal untuk Gambar Medis dengan Perintah Teks"
Ini adalah model segmentasi universal yang ditingkatkan pengetahuan yang dibangun berdasarkan pengumpulan data yang belum pernah ada sebelumnya (72 kumpulan data segmentasi medis 3D publik), yang dapat mensegmentasi 497 kelas dari 3 modalitas berbeda (MR, CT, PET) dan 8 wilayah tubuh manusia, yang diminta oleh teks (anatomi). terminologi).
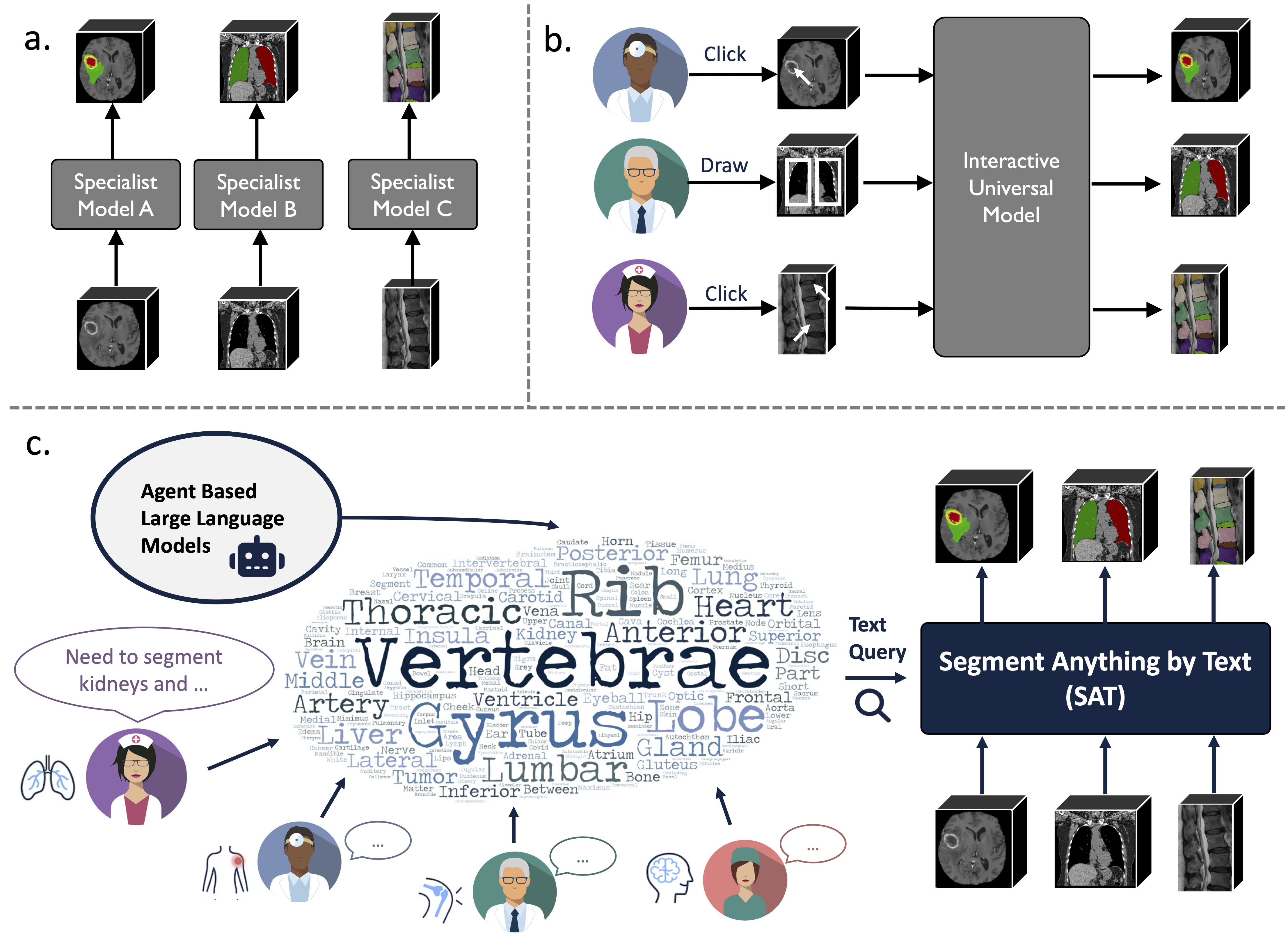
Hal ini bisa menjadi lebih kuat dan efisien daripada melatih dan menerapkan serangkaian model khusus. Temukan lebih banyak di situs web atau makalah kami.
2024.08 ? Berdasarkan SAT dan model bahasa besar, kami membangun kumpulan data interpretasi CT dada 3D yang komprehensif, berskala besar, dan dipandu wilayah. Ini berisi segmentasi tingkat organ untuk 196 kategori, dan laporan multi-perincian, di mana setiap kalimat didasarkan pada segmentasi yang sesuai. Periksa di wajah pelukan.
2024.06 ? Kami telah merilis kode untuk membuat SAT-DS , kumpulan 72 kumpulan data segmentasi publik, berisi lebih dari 22 ribu gambar 3D, 302 ribu masker segmentasi, dan 497 kelas dari 3 modalitas berbeda (MRI, CT, PET) dan 8 wilayah tubuh manusia, yang menjadi dasar pembuatannya. kami membangun SAT. Kami juga menawarkan tautan pengunduhan pintasan untuk kumpulan data 42/72, yang telah kami proses sebelumnya dan dikemas demi kenyamanan Anda, siap untuk segera digunakan setelah pengunduhan dan ekstraksi. Periksa repo ini untuk detailnya.
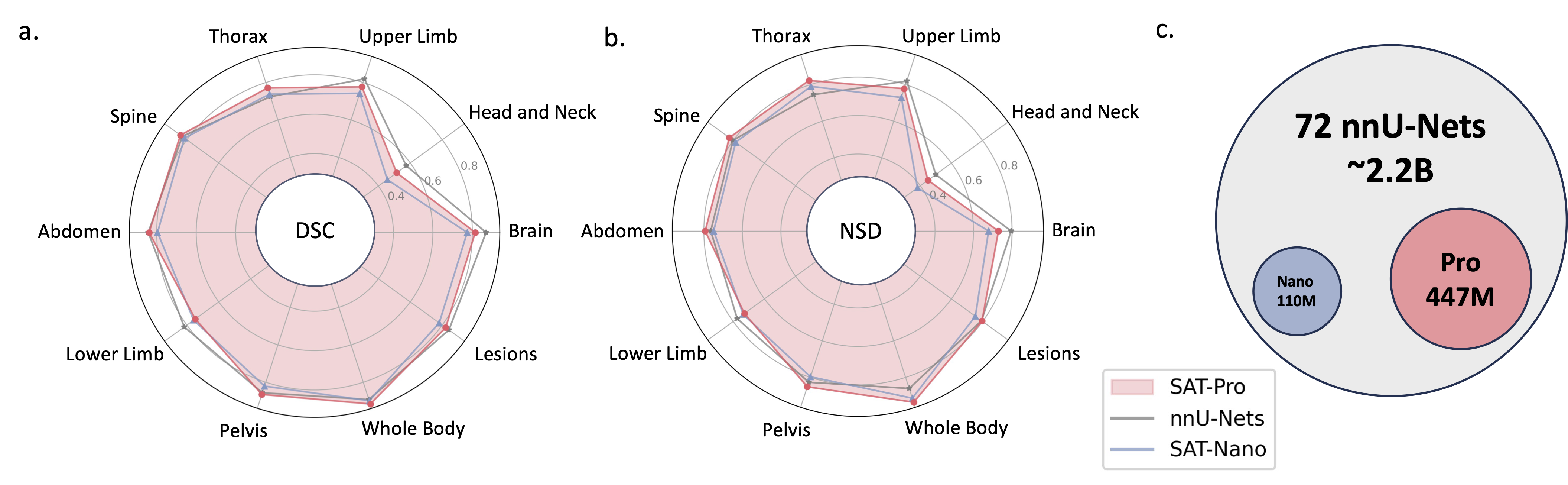
2024.05? Kami melatih SAT versi baru dengan ukuran model lebih besar ( SAT-Pro ) dan lebih banyak kumpulan data ( 72 ), dan sekarang mendukung 497 kelas! Kami juga memperbarui SAT-Nano, dan merilis beberapa varian SAT-Nano, berdasarkan tulang punggung visual yang berbeda (U-Mamba dan SwinUNETR) dan encoder teks (MedCPT dan BERT-Base). Untuk detail lebih lanjut tentang pembaruan ini, lihat makalah baru kami.
Implementasi U-Net bergantung pada versi arsitektur jaringan dinamis yang disesuaikan, untuk menginstalnya:
cd model
pip install -e dynamic-network-architectures-main
Beberapa persyaratan utama lainnya:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
Anda juga perlu menginstal mamba_ssm jika Anda menginginkan SAT-Nano varian U-Mamba
S1. Bangun lingkungan dengan mengikuti requirements.txt .
S2. Unduh pos pemeriksaan SAT dan Encoder Teks dari huggingface.
S3. Siapkan data dalam file jsonl. Periksa demo di data/inference_demo/demo.jsonl .
image (jalur ke gambar), labe (nama target segmentasi), dataset (dataset mana yang dimiliki sampel) dan modality (ct, mri atau pet) diperlukan untuk setiap sampel yang akan disegmentasi. Modalitas dan kelas yang didukung SAT dapat ditemukan di Tabel 12 makalah ini.
orientation_code (orientasi) adalah RAS secara default, yang sesuai dengan sebagian besar gambar dalam bidang aksial. Untuk gambar pada bidang sagital (misalnya pemeriksaan tulang belakang), setel ke ASR . Gambar masukan harus berbentuk H,W,D Kode proses data kami akan menormalkan gambar masukan dalam hal orientasi, intensitas, jarak, dan sebagainya. Dua gambar yang berhasil diproses dapat ditemukan di demoprocessed_data , pastikan normalisasi dilakukan dengan benar untuk menjamin kinerja SAT.
S4. Mulai inferensi dengan SAT-Pro?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d adalah ukuran batch patch gambar masukan, dan perlu disesuaikan berdasarkan memori GPU (periksa tabel di bawah); --max_queries disarankan untuk menyetel lebih besar dari kelas dalam kumpulan data inferensi, kecuali memori GPU Anda sangat terbatas;
| Model | ukuran batch_3d | Memori GPU |
|---|---|---|
| SAT-Pro | 1 | ~ 34GB |
| SAT-Pro | 2 | ~ 62GB |
| SAT-Nano | 1 | ~ 24GB |
| SAT-Nano | 2 | ~ 36GB |
S5. Periksa --rcd_dir untuk keluaran. Hasil disusun berdasarkan kumpulan data. Untuk setiap kasus, gambar masukan, hasil segmentasi gabungan, dan folder yang berisi segmentasi setiap kelas akan ditemukan. Semua keluaran disimpan sebagai file nifiti. Anda dapat memvisualisasikannya menggunakan ITK-SNAP.
Jika Anda ingin menggunakan SAT-Nano yang dilatih pada 72 kumpulan data, cukup ubah --vision_backbone menjadi 'UNET', lalu ubah --checkpoint dan --text_encoder_checkpoint sesuai kebutuhan.
Untuk varian SAT-Nano lainnya (dilatih pada 49 kumpulan data):
UNET-Milik kita: setel --vision_backbone 'UNET' dan --text_encoder 'ours' ;
UNET-CPT: setel --vision_backbone 'UNET' dan --text_encoder 'medcpt' ;
UNET-BB: setel --vision_backbone 'UNET' dan --text_encoder 'basebert' ;
UMamba-CPT: setel --vision_backbone 'UMamba' dan --text_encoder 'medcpt' ;
SwinUNETR-CPT: setel --vision_backbone 'SwinUNETR' dan --text_encoder 'medcpt' ;
Beberapa persiapan sebelum memulai pelatihan:
sh/ untuk memulai proses pelatihan. Ambil SAT-Pro misalnya: sbatch sh/train_sat_pro.sh
Ini juga memerlukan pembuatan data pengujian setelah repo ini. Anda dapat merujuk ke skrip slurm sh/evaluate_sat_pro.sh untuk memulai proses evaluasi:
sbatch sh/evaluate_sat_pro.sh
Jika Anda menggunakan kode ini untuk penelitian atau proyek Anda, harap kutip:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}