
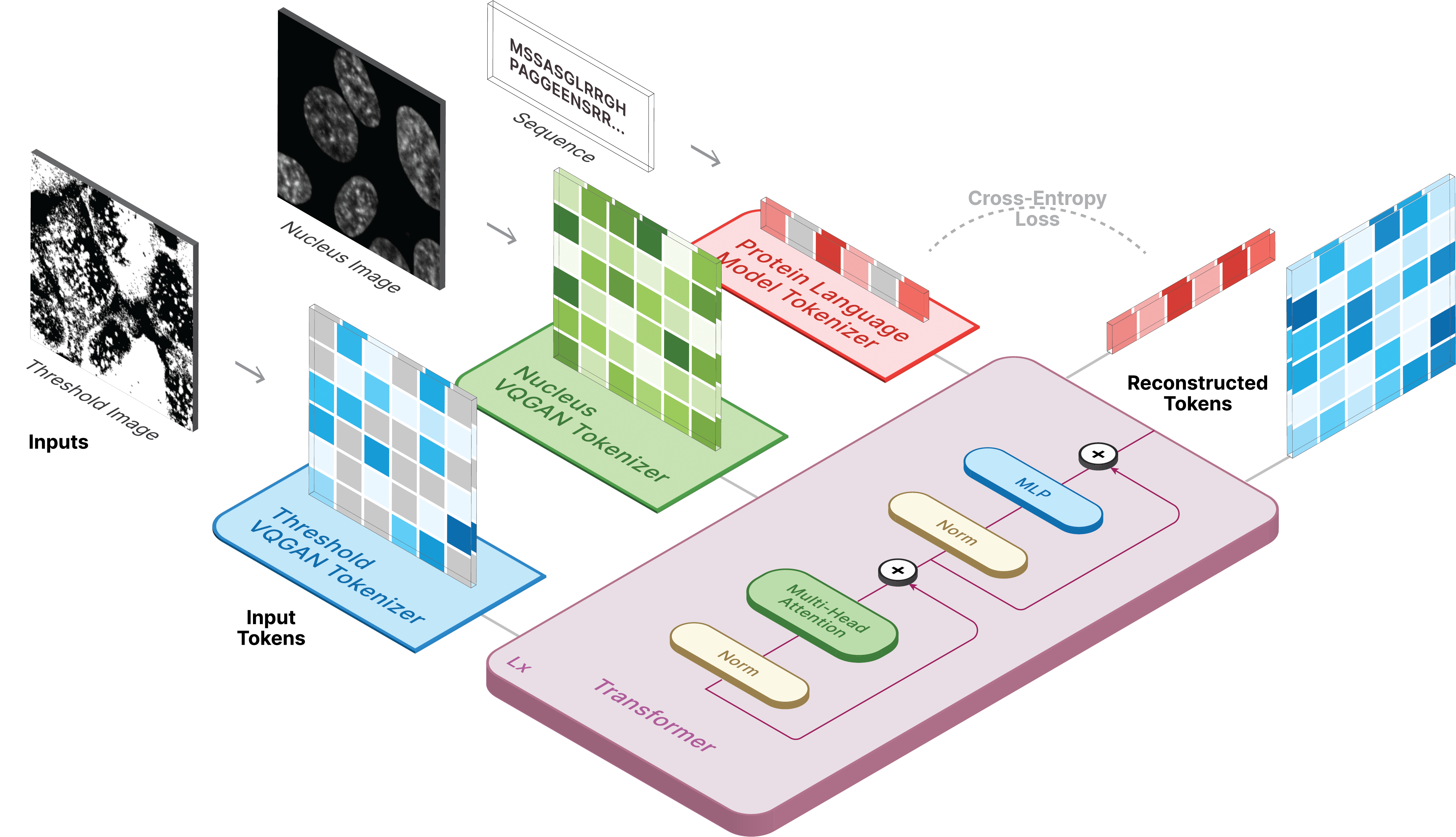
Repositori ini adalah implementasi resmi dari CELL-E 2: Menerjemahkan Protein ke Gambar dan Kembali dengan Transformator Teks-ke-Gambar Dua Arah.
Buat lingkungan virtual dan instal paket yang diperlukan melalui:
pip install -r requirements.txt
Selanjutnya install torch = 2.0.0 dengan versi CUDA yang sesuai
Model tersedia di Hugging Face.
Kami juga memiliki dua ruang yang tersedia di mana Anda dapat menjalankan prediksi pada data Anda sendiri!
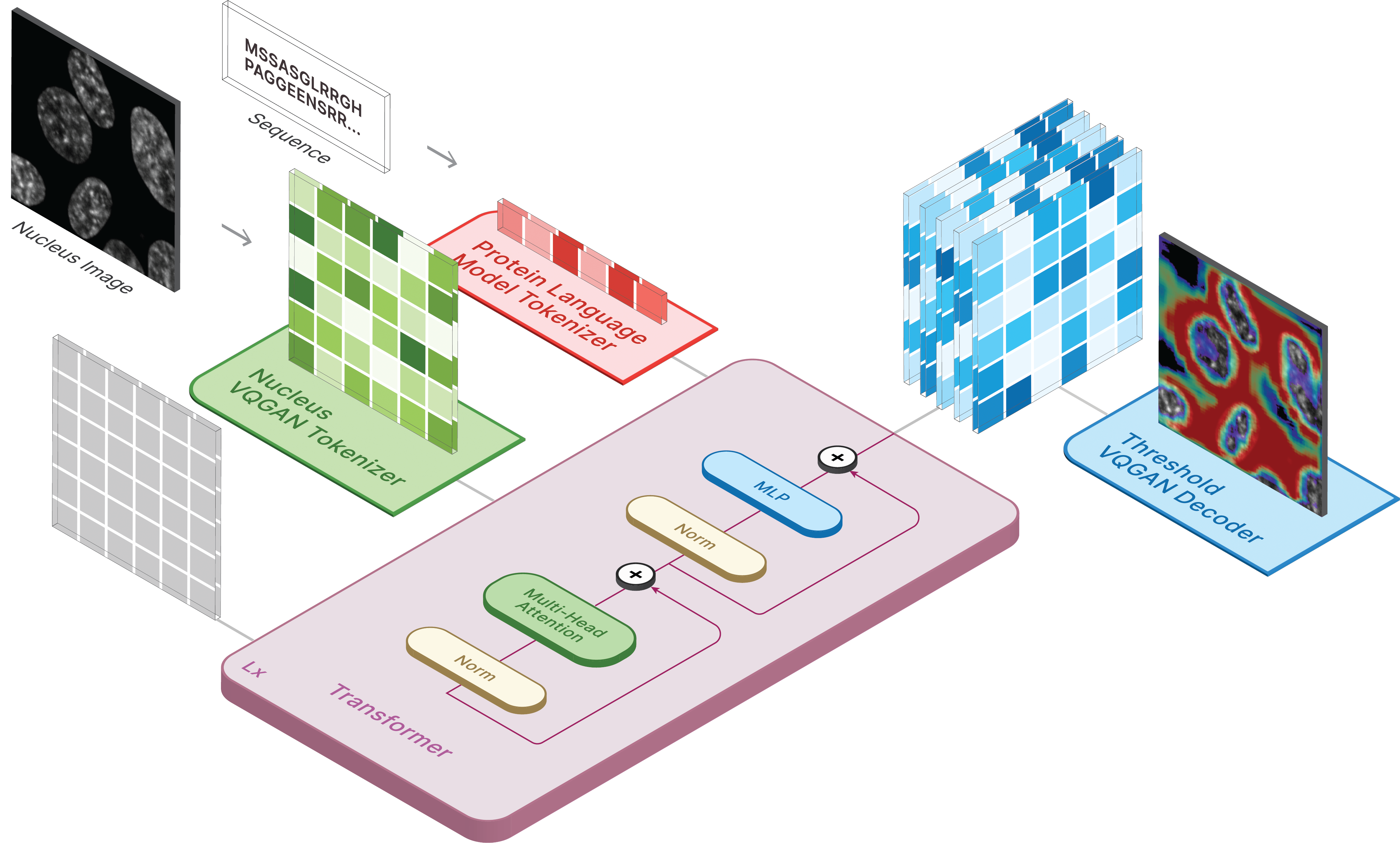
Untuk menghasilkan gambar, atur model yang disimpan sebagai ckpt_path. Metode ini mungkin tidak stabil, jadi lihat Demo.ipynb untuk melihat cara memuat lainnya.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )Pelatihan untuk CELL-E terjadi dalam 3 tahap:
Jika menggunakan gambar ambang batas protein, tetapkan threshold: True untuk kumpulan data.
Kami menggunakan versi kode penjinak transformator yang sedikit dimodifikasi.
Untuk melatih, jalankan skrip berikut:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Silakan merujuk ke repo asli untuk mengetahui tanda tambahan, seperti --gpus .
Kami menyediakan script untuk mendownload gambar Human Protein Atlas dan OpenCell di folder scripts. data_csv diperlukan untuk pemuat data. Anda harus membuat file csv yang berisi nucleus_image_path , protein_image_path , metadata_path , split (train atau val), dan sequence (opsional). Diasumsikan bahwa file ini ada dalam folder data umum yang sama dengan file gambar dan metadata.
Metadata adalah JSON yang harus menyertai setiap rangkaian protein. Jika suatu urutan tidak muncul di data_csv , maka harus muncul di metadata.json dengan kunci bernama protein_sequence .
Menambahkan lebih banyak informasi di sini dapat berguna untuk menanyakan protein individu. Mereka dapat diambil melalui retrieve_metadata , yang membuat variabel self.metadata dalam objek kumpulan data.
Untuk melatih, jalankan skrip berikut:
python celle_main.py --base configs/celle.yaml -t True
Tentukan --gpus dalam format yang sama dengan VQGAN.
CELL-E berisi opsi berikut:
ckpt_path : Melanjutkan pelatihan CELL-E 2 sebelumnya. Model tersimpan dengan state_dictvqgan_model_path : Model gambar protein yang disimpan (dengan state_dict) untuk pembuat enkode gambar proteinvqgan_config_path : Model gambar protein yaml yang disimpancondition_model_path : Model kondisi (inti) yang disimpan (dengan state_dict) untuk pembuat enkode gambar proteincondition_config_path : Model kondisi (inti) tersimpan yamlnum_images : 1 jika hanya menggunakan encoder gambar protein, 2 jika menyertakan encoder gambar kondisiimage_key : nucleus , target , atau thresholddim : Dimensi penyematan model bahasanum_text_tokens : jumlah total token dalam model bahasa (33 untuk ESM-2)text_seq_len : Jumlah total asam amino yang dipertimbangkandepth : Kedalaman model transformator, lebih dalam biasanya lebih baik dengan mengorbankan VRAMheads : jumlah kepala yang digunakan dalam perhatian multi-kepaladim_head : ukuran kepala perhatianattn_dropout : Tingkat Dropout Perhatian dalam pelatihanff_dropout : Tingkat Dropout Feed-Forward dalam pelatihanloss_img_weight : Pembobotan diterapkan pada rekonstruksi gambar. berat teks = 1loss_text_weight : Pembobotan diterapkan pada kondisi rekonstruksi gambar.stable : Bobot norma (saat ledakan gradien terjadi)learning_rate : Kecepatan pembelajaran untuk pengoptimal Adammonitor : Param digunakan untuk menyimpan model Silakan kutip kami jika Anda memutuskan untuk menggunakan kode kami untuk bagian mana pun dari penelitian Anda.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}