Selamat datang di repositori InstaDeep Github ini, yang menampilkan:
Kami sangat senang dapat menjadikan karya-karya ini menjadi sumber terbuka dan memberi komunitas akses ke kode dan bobot terlatih untuk sembilan model bahasa genomik dan 2 model segmentasi ini. Model dari Proyek nucleotide transformer dikembangkan bekerja sama dengan Nvidia dan TUM, dan model tersebut dilatih pada node DGX A100 di Cambridge-1. Model dari proyek nucleotide transformer Agro dikembangkan bekerja sama dengan Google, dan model tersebut dilatih pada akselerator TPU-v4.
Secara keseluruhan, karya kami memberikan wawasan baru terkait dengan pelatihan awal dan penerapan model dasar bahasa, serta pelatihan model yang menggunakannya sebagai encoder tulang punggung, hingga genomik dengan banyak peluang penerapannya di lapangan.
Di repositori ini, Anda akan menemukan yang berikut ini:
Dibandingkan dengan pendekatan lain, model kami tidak hanya mengintegrasikan informasi dari genom referensi tunggal, namun juga memanfaatkan rangkaian DNA dari lebih dari 3.200 genom manusia yang beragam, serta 850 genom dari berbagai spesies, termasuk organisme model dan non-model. Melalui evaluasi yang kuat dan ekstensif, kami menunjukkan bahwa model besar ini memberikan prediksi fenotip molekuler yang sangat akurat dibandingkan dengan metode yang ada.
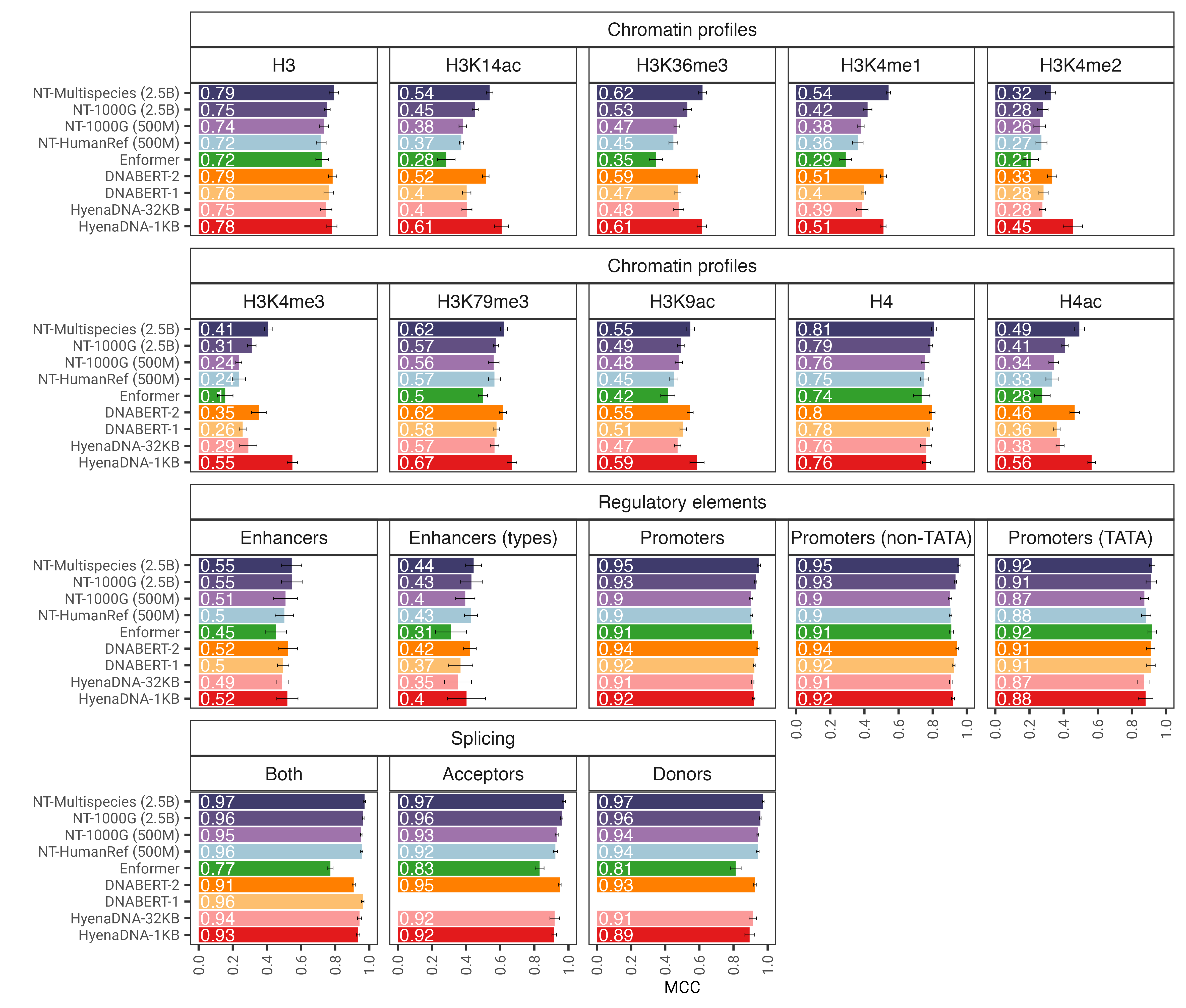
Gambar 1: Model nucleotide transformer secara akurat memprediksi beragam tugas genomik setelah penyesuaian. Kami menunjukkan hasil kinerja di seluruh tugas hilir untuk model transformator yang disempurnakan. Bilah kesalahan mewakili 2 SD yang diperoleh dari validasi silang 10 kali lipat.
Dalam karya ini kami menyajikan model bahasa besar dasar baru yang dilatih pada genom referensi dari 48 spesies tanaman dengan fokus utama pada spesies tanaman. Kami menilai kinerja AgroNT di beberapa tugas prediksi mulai dari fitur regulasi, pemrosesan RNA, dan ekspresi gen, dan menunjukkan bahwa AgroNT dapat memperoleh kinerja canggih.
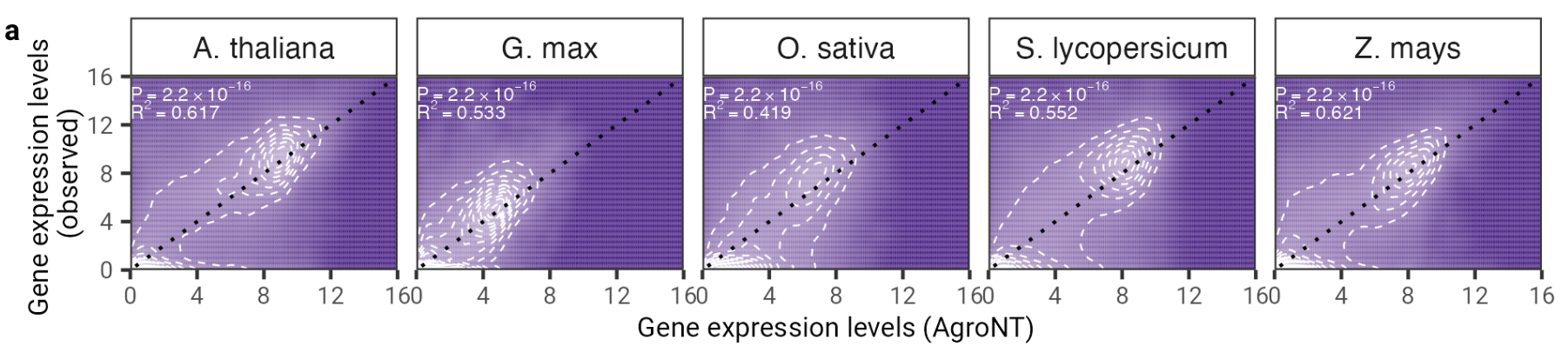
Gambar 2: AgroNT memberikan prediksi ekspresi gen pada berbagai spesies tanaman. Prediksi ekspresi gen pada gen yang bertahan di semua jaringan berkorelasi dengan tingkat ekspresi gen yang diamati. Koefisien determinasi (R 2 ) dari model linier dan nilai P yang terkait antara nilai prediksi dan nilai observasi ditampilkan.
Untuk menggunakan kode dan model terlatih, cukup:
pip install . .Anda kemudian dapat mengunduh dan melakukan inferensi dengan salah satu dari sembilan model kami hanya dalam beberapa baris kode:
import haiku as hk
import jax
import jax . numpy as jnp
from nucleotide_transformer . pretrained import get_pretrained_model
# Get pretrained model
parameters , forward_fn , tokenizer , config = get_pretrained_model (
model_name = "500M_human_ref" ,
embeddings_layers_to_save = ( 20 ,),
max_positions = 32 ,
)
forward_fn = hk . transform ( forward_fn )
# Get data and tokenize it
sequences = [ "ATTCCGATTCCGATTCCG" , "ATTTCTCTCTCTCTCTGAGATCGATCGATCGAT" ]
tokens_ids = [ b [ 1 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens_str = [ b [ 0 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens = jnp . asarray ( tokens_ids , dtype = jnp . int32 )
# Initialize random key
random_key = jax . random . PRNGKey ( 0 )
# Infer
outs = forward_fn . apply ( parameters , random_key , tokens )
# Get embeddings at layer 20
print ( outs [ "embeddings_20" ]. shape )Nama model yang didukung adalah:
Anda juga dapat menjalankan model kami dan menemukan lebih banyak contoh kode di google colab
Kode ini berjalan pada GPU dan TPU berkat Jax!
Model nucleotide transformer v2 versi kedua kami mencakup serangkaian perubahan arsitektur yang terbukti lebih efisien: alih-alih menggunakan penyematan posisi yang dipelajari, kami menggunakan Penyematan Putar yang digunakan di setiap lapisan perhatian dan Unit Linier Berpagar dengan aktivasi desir tanpa bias. Model yang ditingkatkan ini juga menerima urutan hingga 2.048 token yang mengarah ke jendela konteks yang lebih panjang yaitu 12kbp. Terinspirasi oleh undang-undang penskalaan Chinchilla, kami juga melatih model NT-v2 pada kumpulan data multi-spesies kami untuk durasi yang lebih lama (300 miliar token untuk model 50 juta dan 100 juta; 1 ton token untuk model 250 juta dan 500 juta) dibandingkan dengan model v1 (token 300 miliar untuk keempat model).
Lapisan transformator memiliki indeks 1, yang berarti memanggil get_pretrained_model dengan argumen model_name="500M_human_ref" dan embeddings_layers_to_save=(1, 20,) akan menghasilkan ekstraksi embeddings setelah lapisan transformator pertama dan ke-20. Untuk transformator yang menggunakan kepala Roberta LM, praktik umum adalah mengekstraksi embeddings akhir setelah norma lapisan pertama kepala LM, bukan setelah blok transformator terakhir. Oleh karena itu, jika get_pretrained_model dipanggil dengan argumen berikut embeddings_layers_to_save=(24,) , embeddings tidak akan diekstraksi setelah lapisan transformator terakhir melainkan setelah norma lapisan pertama kepala LM.
Model SegmenNT memanfaatkan transformator nucleotide transformer (NT) yang kepala model bahasanya kami hilangkan dan diganti dengan kepala segmentasi U-Net 1 dimensi untuk memprediksi lokasi beberapa jenis elemen genomik dalam urutan pada resolusi nukleotida tunggal. Kami menyajikan dua varian model berbeda pada 14 kelas elemen genom manusia yang berbeda dalam urutan input hingga 30kb. Ini termasuk gen (gen pengkode protein, lncRNA, 5'UTR, 3'UTR, ekson, intron, akseptor sambungan dan situs donor) dan pengatur (sinyal poliA, promotor dan peningkat invarian jaringan dan spesifik jaringan, serta terikat CTCF situs) elemen. SegmenNT mencapai kinerja yang unggul dibandingkan arsitektur segmentasi U-Net yang canggih, memanfaatkan bobot NT yang telah dilatih sebelumnya, dan mendemonstrasikan generalisasi zero-shot hingga 50kbp.
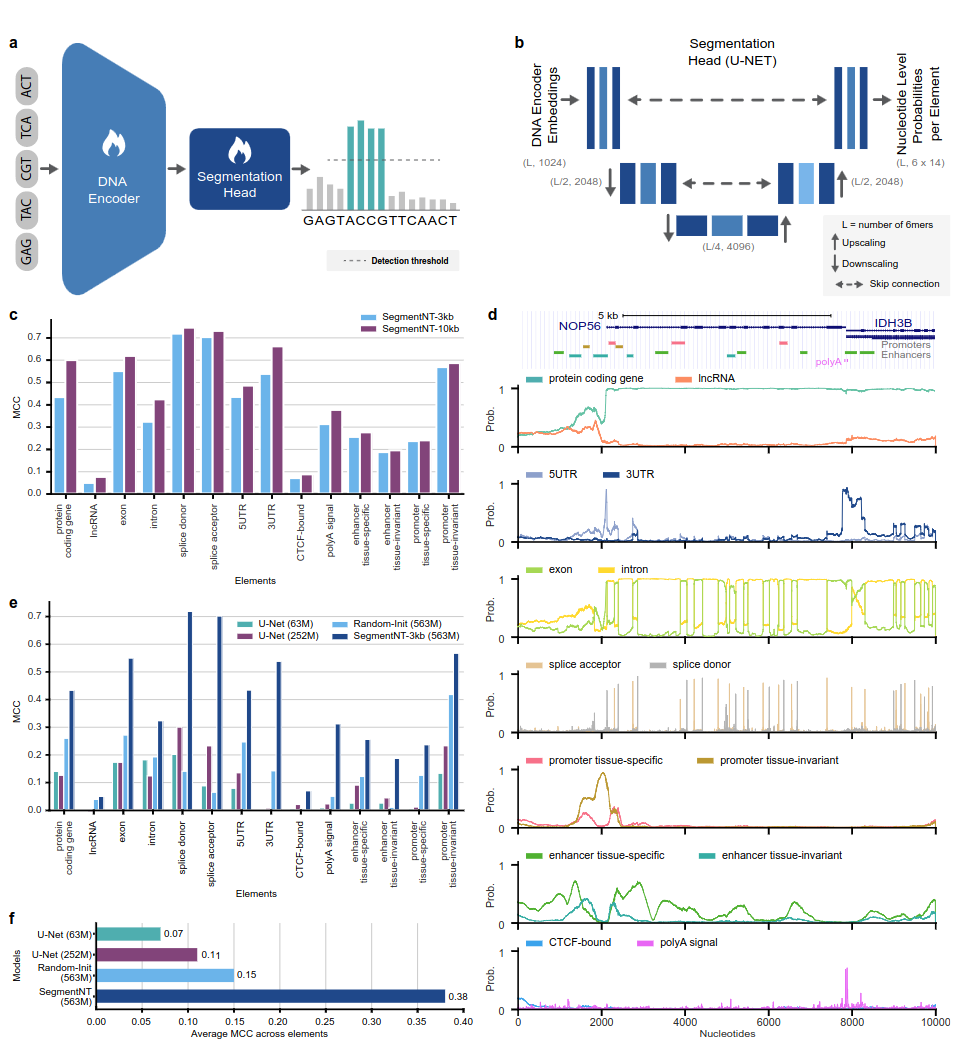
Gambar 1: SegmentNT melokalisasi elemen genomik pada resolusi nukleotida.
Untuk menggunakan kode dan model terlatih, cukup:
pip install . .Anda kemudian dapat mengunduh dan menyimpulkan secara berurutan dengan salah satu model kami hanya dalam beberapa baris kode:
rescaling factor diatur ke faktor yang digunakan selama pelatihan. Jika Anda perlu menyimpulkan urutan antara 30kbp dan 50kbp, pastikan untuk meneruskan argumen rescaling_factor dalam fungsi get_pretrained_segment_nt_model dengan nilai rescaling_factor = max_num_nucleotides / max_num_tokens_nt di mana num_dna_tokens_inference adalah jumlah token pada inferensi (yaitu 6669 untuk urutan basis 40008 berpasangan) dan max_num_tokens_nt adalah jumlah maksimum token tempat transformator nukleotida tulang punggung dilatih, yaitu 2048 .
? examples/inference_segment_nt.ipynb menampilkan cara menyimpulkan urutan 50kb dan memplot probabilitas untuk mereproduksi Gambar.3 makalah.
? Model SegmentNT tidak menangani "N" apa pun dalam rangkaian masukan karena setiap nukleotida perlu diberi token sebagai 6-mer, yang tidak dapat dilakukan jika menggunakan rangkaian yang mengandung satu atau beberapa pasangan basa "N".
import haiku as hk
import jax
import jax . numpy as jnp
from nucleotide_transformer . pretrained import get_pretrained_segment_nt_model
# Initialize CPU as default JAX device. This makes the code robust to memory leakage on
# the devices.
jax . config . update ( "jax_platform_name" , "cpu" )
backend = "cpu"
devices = jax . devices ( backend )
num_devices = len ( devices )
print ( f"Devices found: { devices } " )
# The number of DNA tokens (excluding the CLS token prepended) needs to be dividible by
# 2 to the power of the number of downsampling block, i.e 4.
max_num_nucleotides = 8
assert max_num_nucleotides % 4 == 0 , (
"The number of DNA tokens (excluding the CLS token prepended) needs to be dividible by"
"2 to the power of the number of downsampling block, i.e 4." )
parameters , forward_fn , tokenizer , config = get_pretrained_segment_nt_model (
model_name = "segment_nt" ,
embeddings_layers_to_save = ( 29 ,),
attention_maps_to_save = (( 1 , 4 ), ( 7 , 10 )),
max_positions = max_num_nucleotides + 1 ,
)
forward_fn = hk . transform ( forward_fn )
apply_fn = jax . pmap ( forward_fn . apply , devices = devices , donate_argnums = ( 0 ,))
# Get data and tokenize it
sequences = [ "ATTCCGATTCCGATTCCAACGGATTATTCCGATTAACCGATTCCAATT" , "ATTTCTCTCTCTCTCTGAGATCGATGATTTCTCTCTCATCGAACTATG" ]
tokens_ids = [ b [ 1 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens_str = [ b [ 0 ] for b in tokenizer . batch_tokenize ( sequences )]
tokens = jnp . asarray ( tokens_ids , dtype = jnp . int32 )
random_key = jax . random . PRNGKey ( seed = 0 )
keys = jax . device_put_replicated ( random_key , devices = devices )
parameters = jax . device_put_replicated ( parameters , devices = devices )
tokens = jax . device_put_replicated ( tokens , devices = devices )
# Infer on the sequence
outs = apply_fn ( parameters , keys , tokens )
# Obtain the logits over the genomic features
logits = outs [ "logits" ]
# Transform them in probabilities
probabilities = jnp . asarray ( jax . nn . softmax ( logits , axis = - 1 ))[..., - 1 ]
print ( f"Probabilities shape: { probabilities . shape } " )
print ( f"Features inferred: { config . features } " )
# Get probabilities associated with intron
idx_intron = config . features . index ( "intron" )
probabilities_intron = probabilities [..., idx_intron ]
print ( f"Intron probabilities shape: { probabilities_intron . shape } " )Nama model yang didukung adalah:
Kode ini berjalan pada GPU dan TPU berkat Jax!
Model dilatih pada urutan dengan panjang hingga 1000 token, termasuk token <CLS> yang ditambahkan secara otomatis ke awal urutan. Tokenizer memulai tokenisasi dari kiri ke kanan dengan mengelompokkan huruf "A", "C", "G" dan "T" dalam 6-mer. Huruf "N" dipilih untuk tidak dikelompokkan di dalam k-mers, oleh karena itu setiap kali tokenizer menemukan "N", atau jika jumlah nukleotida dalam urutan tersebut bukan kelipatan 6, maka nukleotida akan diberi tokenisasi tanpa pengelompokan. mereka. Contohnya diberikan di bawah ini:
dna_sequence_1 = "ACGTGTACGTGCACGGACGACTAGTCAGCA"
tokenized_dna_sequence_1 = [ < CLS > , < ACGTGT > , < ACGTGC > , < ACGGAC > , < GACTAG > , < TCAGCA > ]
dna_sequence_2 = "ACGTGTACNTGCACGGANCGACTAGTCTGA"
tokenized_dna_sequence_2 = [ < CLS > , < ACGTGT > , < A > , < C > , < N > , < TGCACG > , < G > , < A > , < N > , < CGACTA > , < GTCTGA > ]Oleh karena itu, semua transformator v1 dan v2 dapat mengambil urutan masing-masing hingga 5994 dan 12282 nukleotida jika tidak ada "N" di dalamnya.
Koleksi model yang disajikan dalam repositori ini tersedia di ruang pelukan Instadeep di sini: Ruang nucleotide transformer dan ruang nucleotide transformer Agro!
Kami berterima kasih kepada Maša Roller, serta anggota Rostlab, khususnya Tobias Olenyi, Ivan Koludarov, dan Burkhard Rost atas diskusi konstruktif yang membantu mengidentifikasi arah penelitian yang menarik. Selain itu, kami mengucapkan terima kasih kepada semua pihak yang menyimpan data eksperimen dalam database publik, kepada mereka yang memelihara database tersebut, dan kepada mereka yang menyediakan metode analitis dan prediktif secara bebas. Kami juga berterima kasih kepada tim pengembangan Jax.
Jika Anda merasa repositori ini berguna dalam pekerjaan Anda, harap tambahkan kutipan yang relevan ke salah satu makalah kami yang terkait:
Kertas nucleotide transformer :
@article { dalla2023nucleotide ,
title = { The nucleotide transformer : Building and Evaluating Robust Foundation Models for Human Genomics } ,
author = { Dalla-Torre, Hugo and Gonzalez, Liam and Mendoza Revilla, Javier and Lopez Carranza, Nicolas and Henryk Grywaczewski, Adam and Oteri, Francesco and Dallago, Christian and Trop, Evan and Sirelkhatim, Hassan and Richard, Guillaume and others } ,
journal = { bioRxiv } ,
pages = { 2023--01 } ,
year = { 2023 } ,
publisher = { Cold Spring Harbor Laboratory }
}Kertas nucleotide transformer :
@article { mendoza2024foundational ,
title = { A foundational large language model for edible plant genomes } ,
author = { Mendoza-Revilla, Javier and Trop, Evan and Gonzalez, Liam and Roller, Ma{v{s}}a and Dalla-Torre, Hugo and de Almeida, Bernardo P and Richard, Guillaume and Caton, Jonathan and Lopez Carranza, Nicolas and Skwark, Marcin and others } ,
journal = { Communications Biology } ,
volume = { 7 } ,
number = { 1 } ,
pages = { 835 } ,
year = { 2024 } ,
publisher = { Nature Publishing Group UK London }
}Kertas segmenNT
@article { de2024segmentnt ,
title = { SegmentNT: annotating the genome at single-nucleotide resolution with DNA foundation models } ,
author = { de Almeida, Bernardo P and Dalla-Torre, Hugo and Richard, Guillaume and Blum, Christopher and Hexemer, Lorenz and Gelard, Maxence and Pandey, Priyanka and Laurent, Stefan and Laterre, Alexandre and Lang, Maren and others } ,
journal = { bioRxiv } ,
pages = { 2024--03 } ,
year = { 2024 } ,
publisher = { Cold Spring Harbor Laboratory }
}Jika Anda memiliki pertanyaan atau masukan tentang kode dan model, jangan ragu untuk menghubungi kami.
Terima kasih atas minat Anda pada pekerjaan kami!