Paket ini menyediakan model U-net terlatih untuk segmentasi paru-paru. Untuk saat ini, empat model tersedia:
U-net(R231): Model ini dilatih pada kumpulan data yang besar dan beragam yang mencakup berbagai variabilitas visual. Model melakukan segmentasi pada irisan individu, mengekstraksi paru-paru kanan-kiri secara terpisah meliputi kantong udara, tumor dan efusi. Trakea tidak akan dimasukkan dalam segmentasi paru-paru. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): Model ini dilatih pada subset dari dataset LTRC. Model ini melakukan segmentasi lobus paru-paru individu tetapi menghasilkan kinerja yang terbatas ketika terdapat patologi yang padat atau ketika celah tidak terlihat di setiap irisan.
U-net(LTRCLobes_R231): Ini akan menjalankan model R231 dan LTRCLobes dan menggabungkan hasilnya. Negatif palsu dari LTRCLobes akan diisi dengan prediksi R231 dan dipetakan ke label tetangga. Positif palsu dari LTRCLobe akan dihapus. Proses peleburan memerlukan komputasi yang intensif dan, bergantung pada data dan hasil, dapat memakan waktu hingga beberapa menit per volume.
U-net(R231CovidWeb)
Contoh kedua model yang diterapkan . Kiri: U-net (R231), akan membedakan paru kiri dan kanan dan mencakup area yang sangat padat seperti efusi (baris ketiga), tumor, atau fibrosis parah (baris keempat). Kanan: U-net (LTRLobes), akan membedakan lobus paru-paru namun tidak mencakup area yang sangat padat. LTRCLobes_R231 akan menggabungkan hasil LTRCLobes dan R231. R231CovidWeb dilatih dengan data tambahan COVID-19.
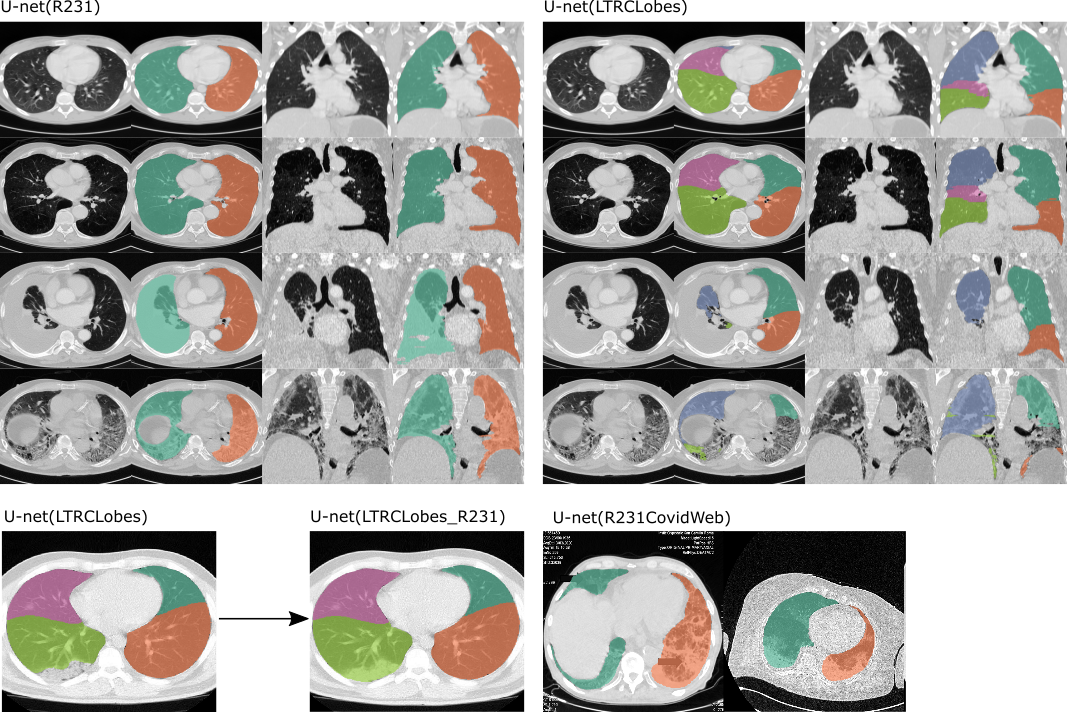
Semantik keluaran :
Dua model label (Kiri-Kanan):
1 = Paru-paru kanan
2 = Paru-paru kiri
Lima model label (lobus paru-paru):
1 = Lobus kiri atas
2 = Lobus kiri bawah
3 = Lobus kanan atas
4 = Lobus tengah kanan
5 = Lobus kanan bawah
Untuk penelitian lebih menarik mengenai data CT paru-paru, lihat situs web kelompok penelitian kami: https://www.cir.meduniwien.ac.at/research/lung/
Jika Anda menggunakan kode ini atau salah satu model terlatih dalam pekerjaan Anda, silakan merujuk ke:
Hofmanninger, J., Doa, F., Pan, J. dkk. Segmentasi paru-paru otomatis dalam pencitraan rutin pada dasarnya adalah masalah keragaman data, bukan masalah metodologi. Euro Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Makalah ini berisi penjelasan rinci tentang dataset yang digunakan, evaluasi menyeluruh terhadap model U-net (R231), dan perbandingan dengan metode referensi.
pip install lungmask
atau
pip install git+https://github.com/JoHof/lungmask
Di Windows, bergantung pada pengaturan Anda, mungkin perlu menginstal torch terlebih dahulu: https://pytorch.org
Waktu proses antara inferensi yang didukung CPU saja dan GPU sangat bervariasi. Menggunakan GPU, memproses volume hanya membutuhkan beberapa detik, menggunakan CPU saja akan memakan waktu beberapa menit. Untuk menggunakan GPU, pastikan instalasi obor Anda memiliki dukungan CUDA. Jika terjadi kesalahan cuda out of memory, kurangi ukuran batch menjadi 1 dengan argumen opsional --batchsize 1
lungmask INPUT OUTPUT
Jika INPUT menunjuk ke suatu file, maka file tersebut akan diproses. Jika INPUT mengarah ke suatu direktori, maka direktori tersebut akan dicari seri DICOM. Volume terbesar yang ditemukan (dalam hal jumlah voxel) akan digunakan untuk menghitung lungmask. OUTPUT adalah nama file keluaran. Semua format ITK didukung.
Pilih model:
U-net (R231) akan digunakan sebagai default. Namun, Anda dapat menentukan model alternatif seperti LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Untuk opsi tambahan ketik:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image harus berupa objek SimpleITK.
Muat model alternatif seperti ini:
inferer = LMInferer(modelname="R231CovidWeb")
Untuk menggunakan kemampuan peleburan model (misalnya LTRCLobes_R231) gunakan:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
Pada versi 0.2.9, array numpy didukung sebagai volume input. Mode ini mengasumsikan array input numpy memiliki format berikut untuk setiap sumbu:
Model ini hanya berfungsi pada irisan penuh. Irisan untuk diproses harus memperlihatkan paru-paru secara utuh dan paru-paru harus dikelilingi oleh jaringan agar dapat tersegmentasi. Namun, model tersebut cukup stabil untuk kasus dengan bidang pandang yang terpotong selama paru-paru dikelilingi oleh jaringan.
lungmask INPUT OUTPUT --modelname R231CovidWeb
Model U-net(R231) reguler bekerja sangat baik untuk CT scan COVID-19. Namun, kumpulan potongan dan laporan kasus dari web sering kali dipotong, diberi anotasi, atau dikodekan dalam format gambar biasa sehingga nilai unit hounsfield (HU) asli hanya dapat diperkirakan. Data pelatihan model U-net (R231CovidWeb) ditambah dengan irisan COVID-19 yang dipetakan kembali dari format pencitraan biasa ke HU. Data dikumpulkan dan disiapkan oleh MedSeg (http://medicalsegmentation.com/covid19/). Meskipun U-net biasa (R231) menunjukkan hasil yang sangat baik untuk gambar-gambar ini, mungkin ada kasus di mana model ini akan menghasilkan segmentasi yang sedikit lebih baik. Perhatikan bahwa Anda harus memetakan gambar kembali ke HU saat menggunakan gambar dari web. Posting blog ini menjelaskan bagaimana Anda dapat melakukan itu. Alternatifnya, Anda dapat menyetel tag --noHU . 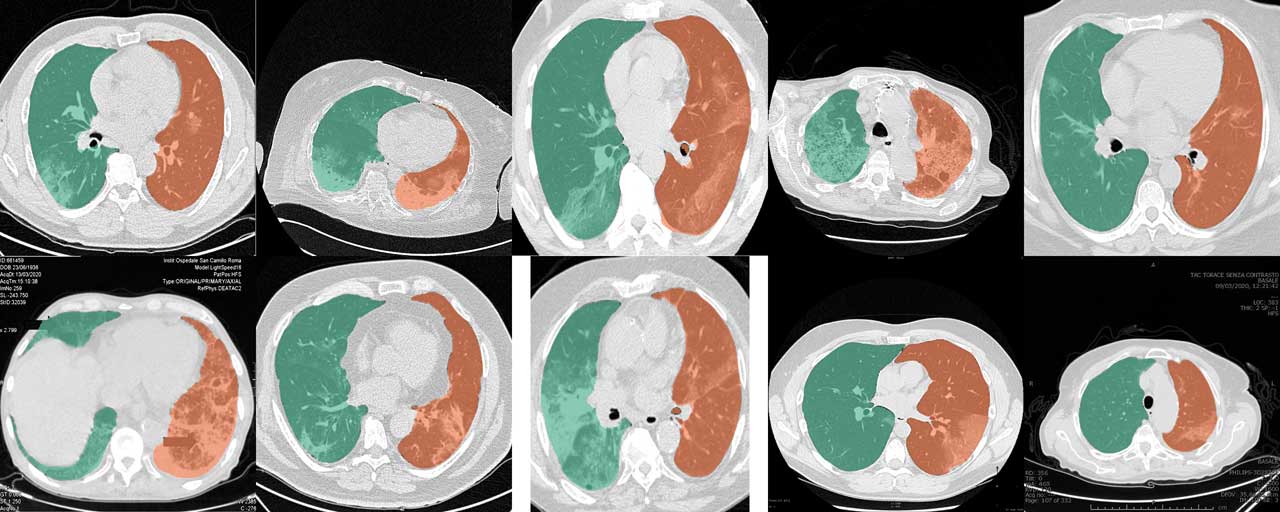
Fitur ini hanya tersedia dalam versi antara 0.2.5 dan 0.2.14 Pada versi 0.2.5 gambar ini didukung. Gunakan tag --noHU jika Anda memproses gambar yang tidak dikodekan dalam HU. Perlu diingat bahwa model dilatih menggunakan CT scan yang tepat dan dikodekan dalam HU. Hasil pada gambar yang dipotong, diberi anotasi, pergeseran intensitas sangat tinggi dan sangat rendah mungkin tidak terlalu dapat diandalkan. Saat menggunakan tag --noHU hanya satu irisan yang dapat diproses.