MedSegDiff kerangka kerja berbasis Diffusion Probabilistic Model (DPM) untuk Segmentasi Citra Medis. Algoritme ini diuraikan dalam makalah kami MedSegDiff: Segmentasi Citra Medis dengan Model Probabilistik Difusi dan MedSegDiff-V2: Segmentasi Citra Medis berbasis Difusi dengan Transformer.
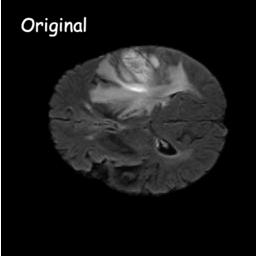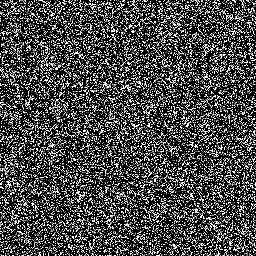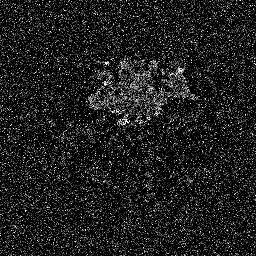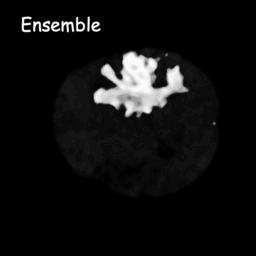 Model Difusi bekerja dengan menghancurkan data pelatihan melalui penambahan derau Gaussian secara berturut-turut, dan kemudian belajar memulihkan data dengan membalikkan proses derau ini. Setelah pelatihan, kita dapat menggunakan Model Difusi untuk menghasilkan data hanya dengan meneruskan sampel kebisingan secara acak melalui proses denoising yang dipelajari. Dalam proyek ini, kami memperluas ide ini ke segmentasi citra medis. Kami memanfaatkan gambar asli sebagai kondisi dan menghasilkan beberapa peta segmentasi dari noise acak, kemudian melakukan ensembling untuk mendapatkan hasil akhir. Pendekatan ini menangkap ketidakpastian dalam citra medis dan mengungguli metode sebelumnya dalam beberapa tolok ukur.
Model Difusi bekerja dengan menghancurkan data pelatihan melalui penambahan derau Gaussian secara berturut-turut, dan kemudian belajar memulihkan data dengan membalikkan proses derau ini. Setelah pelatihan, kita dapat menggunakan Model Difusi untuk menghasilkan data hanya dengan meneruskan sampel kebisingan secara acak melalui proses denoising yang dipelajari. Dalam proyek ini, kami memperluas ide ini ke segmentasi citra medis. Kami memanfaatkan gambar asli sebagai kondisi dan menghasilkan beberapa peta segmentasi dari noise acak, kemudian melakukan ensembling untuk mendapatkan hasil akhir. Pendekatan ini menangkap ketidakpastian dalam citra medis dan mengungguli metode sebelumnya dalam beberapa tolok ukur.
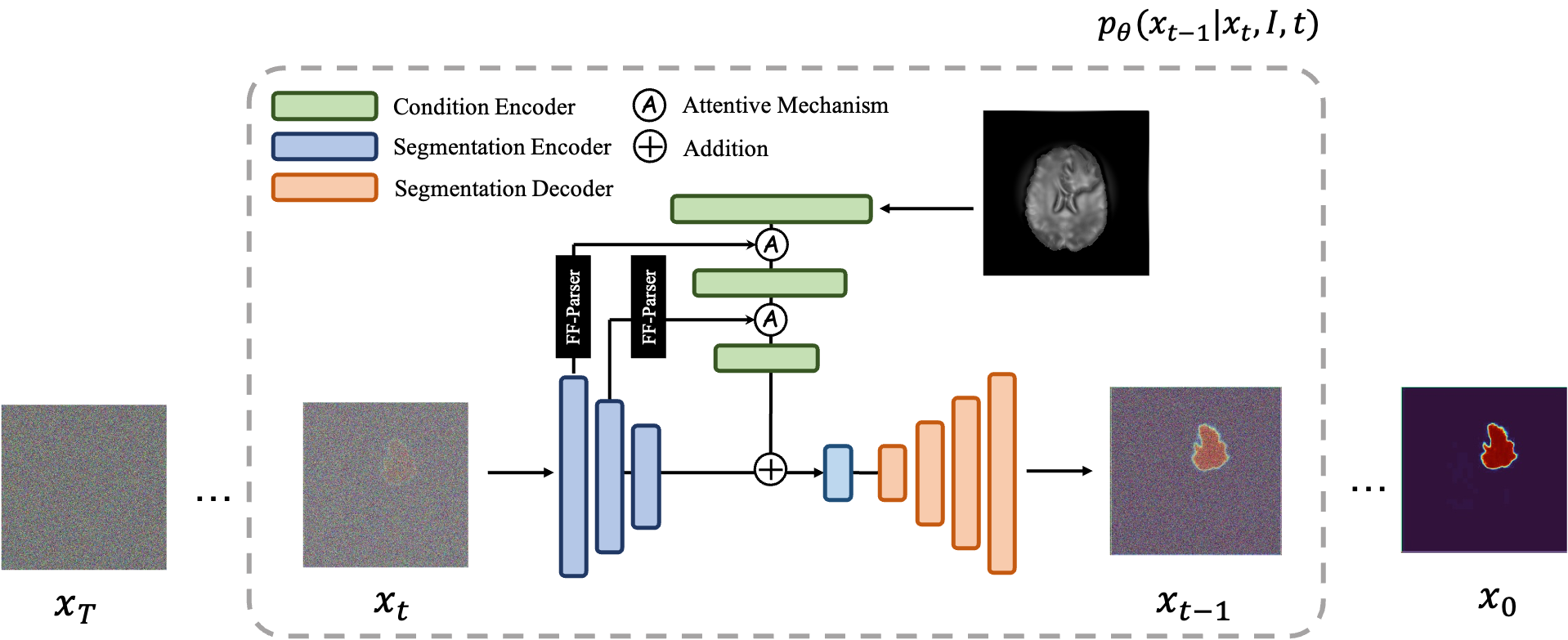 | 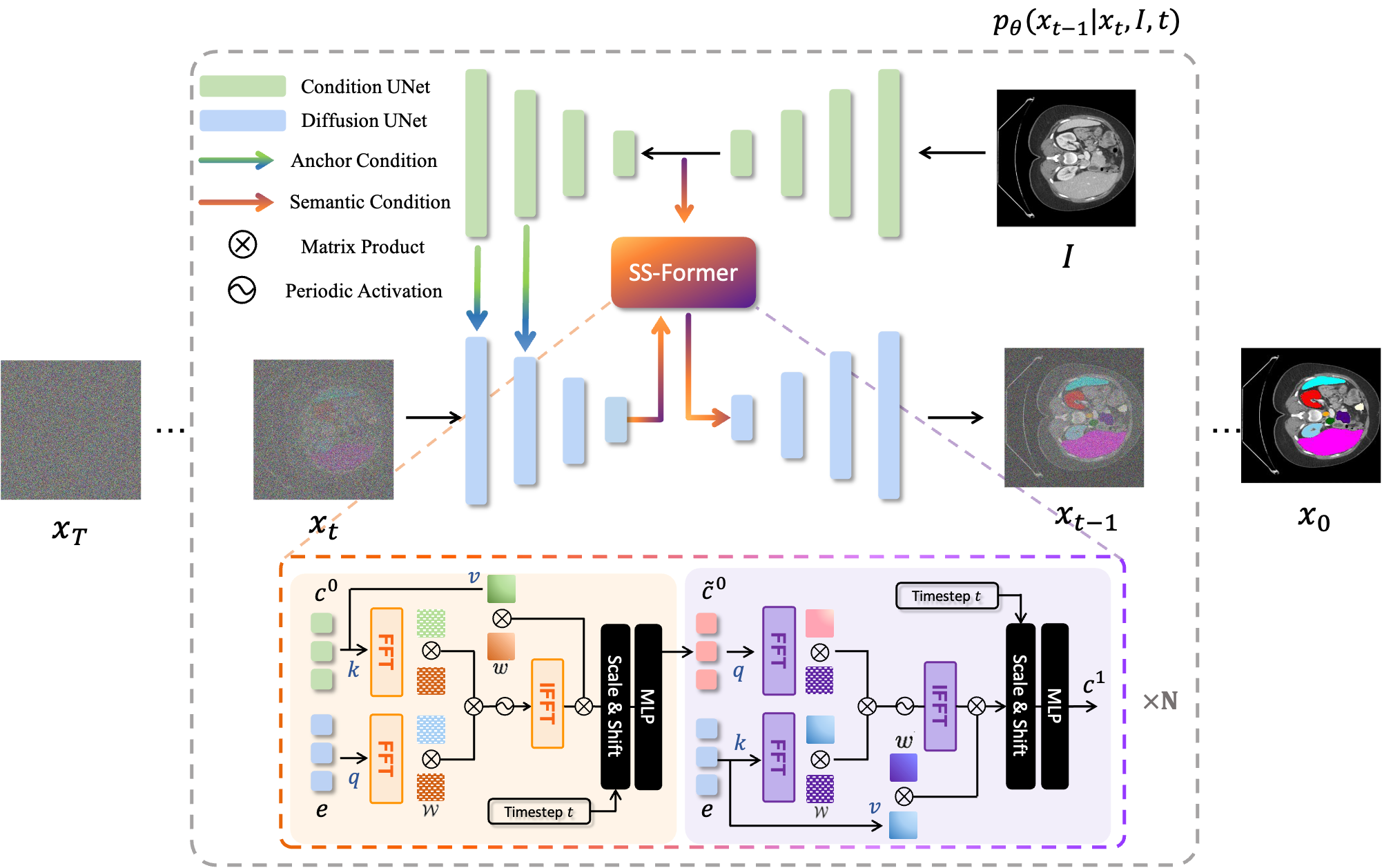 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Untuk pelatihan, jalankan: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Untuk pengambilan sampel, jalankan: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Untuk evaluasi, jalankan python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
Secara default, sampel akan disimpan di ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Untuk pelatihan, jalankan: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Untuk pengambilan sampel, jalankan: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
Sangat mudah untuk menjalankan MedSegDiff pada kumpulan data lainnya. Cukup tulis file pemuat data lain berikut ./guided_diffusion/isicloader.py atau ./guided_diffusion/bratsloader.py . Selamat datang di masalah terbuka jika Anda menemui masalah apa pun. Akan sangat kami hargai jika Anda dapat menyumbangkan ekstensi kumpulan data Anda. Tidak seperti gambar alam, gambar medis sangat bervariasi tergantung pada tugas yang berbeda. Memperluas generalisasi suatu metode memerlukan upaya semua orang.
Untuk melatih model yang bagus, yaitu MedSegDiff-B di makalah, atur hyperparameter model sebagai:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
hyperparameter difusi sebagai:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Untuk mempercepat pengambilan sampel:
--diffusion_steps 50 --dpm_solver True
dijalankan pada beberapa GPU:
--multi-gpu 0,1,2 (for example)
melatih hyperparameter sebagai:
--lr 5e-5 --batch_size 8
dan atur --num_ensemble 5 dalam pengambilan sampel.
Jalankan sekitar 100.000 langkah dalam pelatihan akan dikonvergensi di sebagian besar kumpulan data. Perhatikan bahwa meskipun kerugian tidak berkurang pada sebagian besar langkah selanjutnya, kualitas hasilnya masih meningkat. Proses serupa juga diamati pada aplikasi DPM lainnya, seperti pembuatan gambar. Semoga seseorang yang pintar dapat memberi tahu saya alasannya?.
Saya akan segera mempublikasikan kinerjanya dalam ukuran batch yang lebih kecil (cocok untuk dijalankan pada GPU 24GB) untuk kebutuhan perbandingan?.
Pengaturan untuk mengeluarkan semua potensinya adalah (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Kemudian latih dengan ukuran batch --batch_size 64 dan sampel dengan nomor ansambel --num_ensemble 25 .
Selamat berkontribusi pada MedSegDiff. Teknik apa pun yang dapat meningkatkan kinerja atau mempercepat algoritme sangat kami hargai. Saya menulis MedSegDiff V2, ditujukan untuk publikasi seperti jurnal Nature/CVPR. Saya senang mencantumkan kontributor sebagai rekan penulis saya?.
Kode banyak disalin dari openai/difusi yang ditingkatkan, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Segmentasi berbasis Difusi, hojonathanho/difusi, difusi terpandu, bigmb/Unet-Segmentasi-Pytorch-Nest -of-Unet, nnUnet, lucidrains/vit-pytorch
Silakan kutip
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu