
Harap dicatat bahwa Kode Poseidon baru saja ditransfer ke NextFlow sehingga mungkin masih ada beberapa bug. Jangan ragu untuk melaporkan masalah!
Di sini kami menyajikan Poseidon, pipa untuk mendeteksi situs yang dipilih positif secara signifikan dan kemungkinan peristiwa rekombinasi dalam penyelarasan beberapa urutan pengkodean protein. Situs-situs yang menjalani seleksi positif memberikan wawasan dalam sejarah evolusi urutan Anda, misalnya menunjukkan hot spot mutasi yang penting, diakumulasikan sebagai hasil ras lengan virus-host selama evolusi.
Poseidon bergantung pada berbagai alat pihak ketiga yang berbeda (lihat di bawah). Tapi jangan khawatir, kami merangkum setiap alat dalam wadah Docker sendiri dan menghubungkannya di sistem manajemen alur kerja NextFlow.
Pergi langsung ke contoh kecil output poseidon untuk protein spike SARS-COV-2 dibandingkan dengan studi baru-baru ini dari Zhou et al . 2020.
Anda hanya perlu NextFlow (Versi 20.+) dan Docker diinstal untuk menjalankan pipa. Semua dependensi akan ditarik secara otomatis.
Entah run poseidon dengan mengkloning repositori ini:
git clone https://github.com/hoelzer/poseidon.gitcd poseidon nextflow run poseidon.nf --help
Atau biarkan NextFlow melakukan tarikan
NextFlow Tarik Hoelzer/Poseidon
Kami sarankan menggunakan rilis spesifik Poseidon via
#pullnextflow tarik hoelzer/poseidon -r v1.0.1#runnextflow run hoelzer/poseidon -r v1.0.1 --help
Bergantung pada prosedur instalasi Anda, perbarui pipa melalui git pull atau nextflow pull hoelzer/poseidon .
Penting: Poseidon membutuhkan urutan nukleotida dengan bingkai bacaan terbuka yang benar sebagai input. Selain itu, hasilnya sangat tergantung pada pemilihan sekuens Anda, dengan demikian, Anda dapat mempertimbangkan untuk menjalankan pipa beberapa kali dengan sampel yang berbeda dari urutan input Anda. Juga pipa tidak dapat bekerja dengan terlalu banyak urutan karena dalam intinya Poseidon menggunakan codeml dari suite PAML yang tidak diselesaikan untuk> 100 urutan. Silakan temukan deskripsi terperinci tentang parameter input dan pengaturan di bawah ini.
NextFlow dapat dengan mudah dieksekusi di lingkungan yang berbeda seperti mesin lokal Anda, cluster berkinerja tinggi atau cloud. -profile yang berbeda digunakan untuk memberi tahu NextFlow sistem mana yang harus digunakan. Untuk eksekusi lokal -profile local,docker harus digunakan (dan juga default). Anda juga dapat menjalankan Poseidon pada HPC menggunakan Singularity via -profile lsf,singularity , -profile slurm,singularity atau -profile sge,singularity . Dalam kasus seperti itu, harap juga pertimbangkan untuk menyesuaikan --cachedir untuk menunjukkan di mana menyimpan gambar singularitas di cluster Anda. Parameter --workdir mungkin juga membantu untuk menyesuaikan di mana harus menyimpan direktori kerja sementara (misalnya penggunaan /scratch alih -alih /tmp tergantung pada konfigurasi HPC Anda.)
Sekarang mari kita asumsikan Anda menggunakan NextFlow untuk menarik kode Poseidon dan Anda menjalankan pipa pada mesin lokal menggunakan profil default -profile local,docker .
# Tampilkan Bantuan NextFlow Jalankan Hoelzer/Poseidon ---help # jalankan contoh kecil pada mesin lokal dengan # (pertama kali ini membutuhkan waktu lebih karena wadah Docker diunduh) NextFlow Run Hoelzer/Poseidon -R V1.0.1 - -Fasta ~/.nextflow/aset/hoelzer/poseidon/test_data/bats_mx1_small.fast --Cores 4# Lanjutkan RunNextFlow Run Hoelzer/Poseidon -R V1.0.1 -Fasta ~/.nextflow/Assets/Hoelzer/Poseidon/test_data/Bats_mx1_small.fasta --CORES 4 -RESUME# Alih -alih menggunakan semua core yang tersedia hanya menggunakan jumlah maksimum pada MachinenextFlow lokal Run hooLZER/POSEIDON -R V1.0.1 ---Fasta ~/.nextflow/aset/hoelzer/poseidon/test_data/bats_mx1_small.fasta --max_cores 8 --Cores 4
Untuk mereproduksi hasil seleksi positif yang dilaporkan dalam Fuchs et al . (2017), Journal of Virology Run:
NextFlow Jalankan hoelzer/poseidon -r v1.0.0 ---fasta ~/.nextflow/aset/hoelzer/poseidon/test_data/bats_mx1.fasta --cores 4 --kh-outgroup "pteropus_alecto, eidolon_helvum, rousettus_aegyptiacus, hypsignatus_monstrosus" ---referensi "myotis_daubentonii"
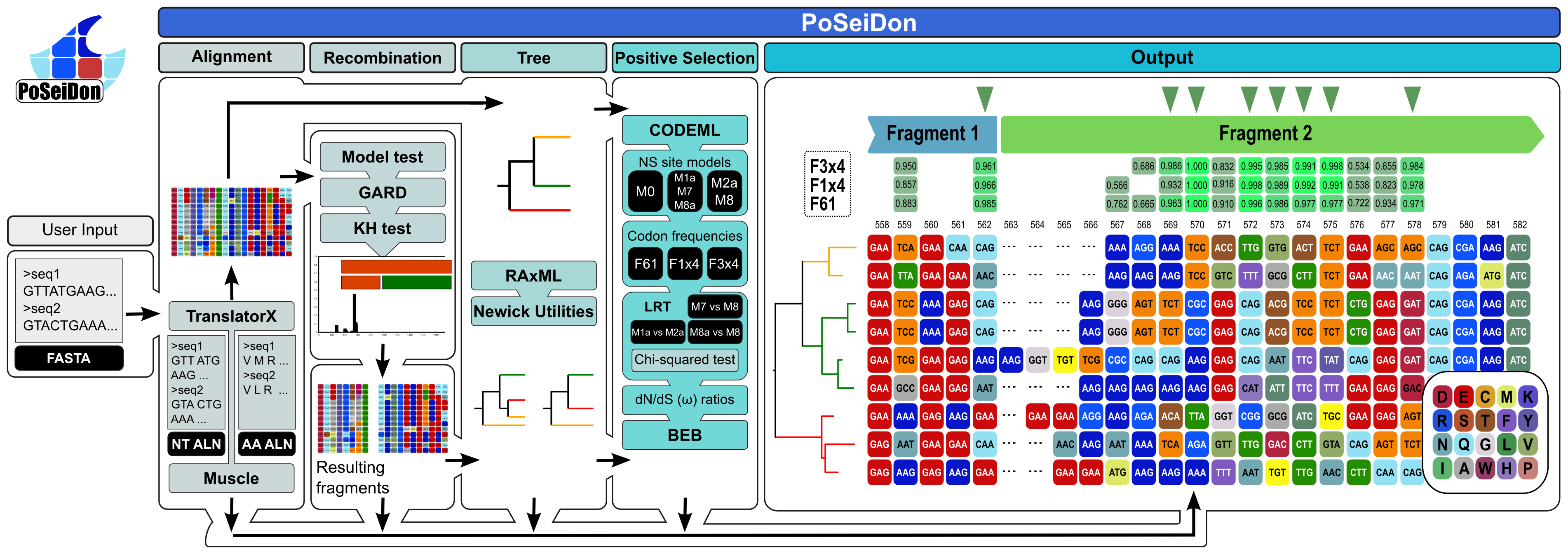
Pipa Poseidon terdiri dari penyelarasan dalam urutan pengkodean protein homolog, deteksi peristiwa rekombinasi yang diduga dan breakpoint evolusi, rekonstruksi filogenetik dan deteksi situs yang dipilih secara positif dalam penyelarasan penuh dan semua fragmen yang mungkin. Akhirnya, semua hasil digabungkan dan divisualisasikan dalam halaman web HTML yang ramah pengguna dan jelas. Fragmen penyelarasan yang dihasilkan ditunjukkan dengan batang berwarna dalam output HTML.
Translatorx (V1.1), Abascal et al . (2010); 20435676
Muscle (v3.8.31), Edgar (2004); 15034147
Raxml (v8.0.25), Stamatakis (2014); 24451623
Newick Utilities (v1.6), Junier dan Zdobnov (2010); 20472542
Modeltest, Posada dan Crandall (1998); 9918953
Hyphy (v2.2), Pond et al . (2005); 15509596
Gard, Pond et al. (2006); 17110367
PAML/CODEML (v4.8), Yang (2007); 17483113
Ruby (v2.3.1)
Inkscape (v1.0)
PDFTEX (v3.14)
Sebagian besar parameter Poseidon adalah opsional dan dijelaskan di bawah ini secara rinci.
--fasta
Wajib. File input FASTA Anda harus mengikuti format:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA... >Myotis_davidii Mx1 Gene ATGGCGGTCGAGATAAGATACGTT...
Semua urutan harus memiliki bingkai bacaan terbuka yang benar, hanya diizinkan mengandung karakter nukleotida [a | c | g | t] dan tidak ada kodon stop internal.
ID urutan harus unik sampai kemunculan pertama ruang.
--reference
Opsional. Default: Gunakan ID urutan pertama sebagai referensi. Anda dapat mendefinisikan satu ID spesies dari beberapa file fasta Anda sebagai spesies referensi. Situs yang dipilih secara positif dan asam amino yang sesuai akan ditarik sehubungan dengan spesies ini. ID harus cocok dengan tajuk FASTA sampai kejadian ruang pertama. Misalnya, jika Anda menginginkan Myotis lucifugus karena spesies referensi Anda dan file FASTA Anda berisi:
>Myotis_lucifugus Mx1 Gene ATGGCGATCGAGATACGATACGTA...
menggunakan
--reference "Myotis_lucifugus"
sebagai parameter untuk mengatur spesies referensi. Per default ID pertama yang terjadi di beberapa file fasta akan digunakan.
--outgroup
Opsional. Default: Pohon tidak root. Anda dapat mendefinisikan satu atau beberapa ID spesies (koma terpisah) sebagai outgroup. Semua pohon filogenetik akan berakar sesuai dengan spesies ini. Misalnya, jika file fasta ganda Anda berisi
ATGGCGATCGAGATACGATACGTA...
>Myotis_davidii Mx1 Gene
ATGGCGGTCGAGATAAGATACGTT...
>Pteropus_vampyrus Mx1 Gene
ATGGCCGTAGAGATTAGATACTTT...
>Eidolon_helvum Mx1 Gene
ATGCCCGTAGAGAATAGATACTTT...Anda dapat mendefinisikan:
--outgroup "Pteropus_vampyrus,Eidolon_helvum"
untuk root semua pohon dalam kaitannya dengan dua spesies ini.
--kh
Opsional. Default: Salah. Dengan parameter ini Anda dapat memutuskan apakah breakpoint yang tidak signifikan harus diperhitungkan. Semua breakpoint diuji untuk ketidaksesuaian topologi yang signifikan menggunakan uji Kashino Hasegawa (KH) Kishino, H. dan Hasegawa, M. (1989). Breakpoint yang tidak signifikan KH paling sering muncul dari variasi panjang cabang antar segmen. Namun demikian, dengan mempertimbangkan breakpoint yang tidak signifikan KH bisa menarik, karena kami sudah mengamati situs yang dipilih secara positif dalam fragmen tanpa ketidaksesuaian topologis yang signifikan. Fragmen yang tidak signifikan KH ditandai dalam output akhir, karena mungkin tidak terjadi dari peristiwa rekombinasi nyata.
Per default hanya breakpoint signifikan yang digunakan untuk perhitungan lebih lanjut.
Harap juga diingat bahwa menggunakan juga breakpoint yang tidak signifikan dapat memperpanjang waktu berjalan Poseidon dari menit ke jam, tergantung pada jumlah breakpoint yang terdeteksi.
Silakan lihat --help untuk parameter lain (gard, raxml, ...) dan beri tahu kami jika Anda membutuhkan lebih banyak kustomisasi!
Jika Poseidon membantu Anda mengutip:
Martin Hölzer dan Manja Marz, "Poseidon: Pipa NextFlow untuk mendeteksi peristiwa rekombinasi evolusi dan seleksi positif", OUP Bioinformatika (2020)