Kami dengan senang hati mengumumkan bahwa ukuran indeks pada disk turun 8 kali dan dalam memori 4 kali karena pindah ke hanya satu jenis fm-index (2bit.64 bukannya 2bit.64 dan 8bit.32) dan kompresi 8x array sufiks. Misalnya, untuk genom manusia, ukuran indeks pada disk turun menjadi ~ 10GB dari ~ 80GB dan jejak memori turun menjadi ~ 10GB dari ~ 40GB. Ada pengurangan substansial dalam waktu IO indeks karena pengurangan dan hampir tidak ada dampak kinerja pada pemetaan baca. Karena perubahan dalam struktur indeks ini (dalam komit #4B59796, 10 Oktober 2020), Anda perlu membangun kembali indeks.
Menambahkan flag MC dalam file SAM output di Commit A591E22. Output harus cocok dengan versi BWA-MEM asli 0.7.17.
Saat komit E0AC59E, kami memiliki Safestringlib Git Submodule. Untuk mendapatkannya, gunakan -Rekursif saat mengkloning atau menggunakan "Git Submodule Init" dan "Git Submodule Update" di repositori yang sudah dikloning (lihat di bawah untuk detail lebih lanjut).
# Gunakan binari yang dikompilasi (disarankan) curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2 | Tar JXF - BWA-MEM2-2.2.1_X64-LINUX/BWA-MEM2 INDEX REF.FA BWA-MEM2-2.2.1_X64-LINUX/BWA-MEM2 MEM REF.FA Read1.fq read2.fq> out.sam# kompilasi dari sumber (tidak disarankan untuk pengguna umum)# Dapatkan SourceGit Clone --Recursive https: // github.com/bwa-mem2/bwa-mem2cd bwa-mem2# orgit clone https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 Git Submodule Init Pembaruan submodule git# kompilasi dan runmake ./bwa-mem2
Alat BWA-MEM2 adalah versi berikutnya dari algoritma BWA-MEM di BWA. Ini menghasilkan perataan yang identik dengan BWA dan ~ 1.3-3.1x lebih cepat tergantung pada kasus penggunaan, dataset, dan mesin yang sedang berjalan.
BWA asli dikembangkan oleh Heng Li (@LH3). Peningkatan kinerja di BWA-MEM2 terutama dilakukan oleh Vasimuddin MD (@yuk12) dan Sanchit Misra (@Sanchit-Misra) dari lab komputasi paralel, Intel. BWA-MEM2 didistribusikan di bawah lisensi MIT.
Untuk pengguna umum, disarankan untuk menggunakan binari yang dikompilasi dari halaman rilis. Binari ini dikompilasi dengan kompiler Intel dan berjalan lebih cepat dari biner yang dikompilasi GCC. Binari yang dikompilasi juga secara tidak langsung mendukung pengiriman CPU. Biner bwa-mem2 dapat secara otomatis memilih implementasi yang paling efisien berdasarkan set instruksi SIMD yang tersedia di mesin yang sedang berjalan. Binari yang dikompilasi dihasilkan pada mesin Centos7 menggunakan baris perintah berikut:
buat cxx = ICPC multi
Penggunaannya persis sama dengan alat BWA Mem asli. Ini sinopsy singkat. Jalankan ./BWA-MEM2 untuk perintah yang tersedia.
# Mengindeks Urutan Referensi (membutuhkan memori 28n GB di mana n adalah ukuran urutan referensi) ../ indeks BWA-MEM2 [-P awalan] <in.fast> di mana <in.fast> adalah jalur ke file fasta urutan referensi dan <drefix> adalah awalan nama file yang menyimpan indeks yang dihasilkan. Default adalah in.fast. # Pemetaan # run "./bwa-mem2 mem" untuk mendapatkan semua opsi./bwa-mem2 mem -t <um_threads> <drefix> <creads.fq/fa>> out.sam Di mana <drefix> adalah awalan yang ditentukan saat membuat indeks atau jalur ke file fasta referensi jika tidak ada awalan yang disediakan.
Dataset:
Genom Referensi: Human_G1K_V37.fasta
| Alias | Sumber Dataset | Jumlah pembacaan | Bacalah panjang |
|---|---|---|---|
| D1 | Institut Broad | 2 x 2.5m bp | 151bp |
| D2 | SRA: SRR7733443 | 2 x 2.5m bp | 151bp |
| D3 | SRA: SRR9932168 | 2 x 2.5m bp | 151bp |
| D4 | SRA: SRX699918 | 2 x 2.5m bp | 151bp |
Detail Mesin:
Prosesor: Intel (R) Xeon (R) 8280 CPU @ 2.70GHz
OS: Rilis Centos Linux 7.6.1810
Memori: 100GB
Kami mengikuti langkah -langkah di bawah ini untuk mengumpulkan hasil kinerja:
A. Langkah Unduh Data:
Unduh SRA Toolkit dari https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global
TAR XFZV Sratoolkit.2.10.5-Centos_linux64.tar.gz
Unduh D2: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-file srr7733443
Unduh D3: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-file srr9932168
Unduh D4: sratoolkit.2.10.5-centos_linux64/bin/fastq-dump --split-file srx699918
B. Langkah Alignment:
Git Clone https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (menggunakan Intel C/C ++ Compiler)
atau make (menggunakan gcc compiler)
./bwa-mem2 index <Ref.fa>
./bwa-mem2 mem [-t <#threads>] <ref.
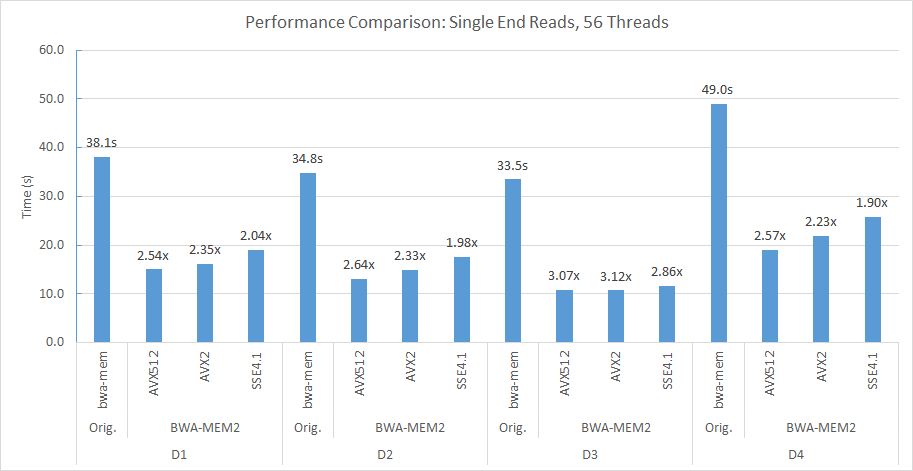
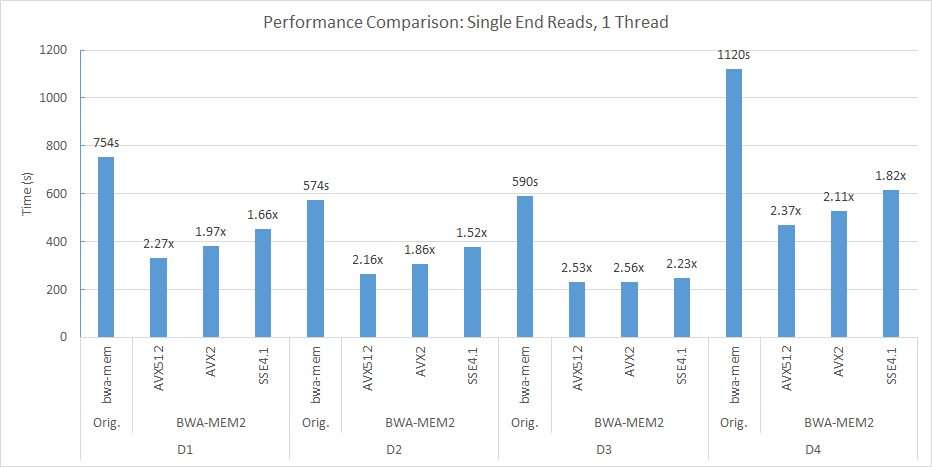
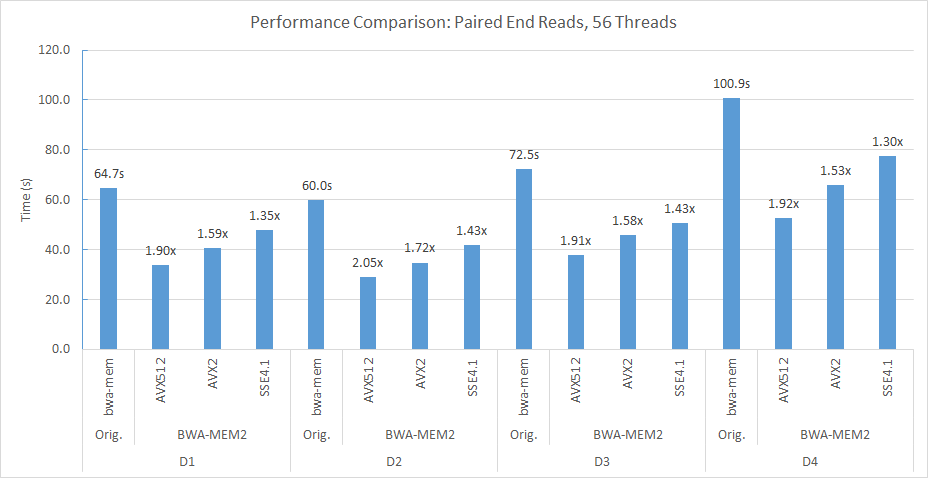
Misalnya, di soket ganda kami (masing -masing 56 utas) dan node NUMA Double, kami menggunakan baris perintah berikut untuk menyelaraskan D2 ke Human_G1K_V37.FASTA Referensi Genome.
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-LISA adalah versi yang dipercepat dari BWA-MEM2 di mana kami menerapkan indeks terpelajar pada fase penyemaian. Cabang BWA-MEM2-LISA berisi kode sumber implementasi. Berikut ini adalah fitur BWA-MEM2-Lisa:
Output yang sama persis dengan BWA-MEM2.
Semua garis perintah untuk membuat indeks dan pemetaan baca persis sama dengan BWA-MEM2.
BWA-MEM2-LISA mempercepat fase penyemaian (salah satu hambatan utama di BWA-MEM2) hingga 4,5x dibandingkan dengan BWA-MEM2.
Jejak memori indeks BWA-MEM2-LISA adalah ~ 120GB untuk genom manusia.
Kode ini hadir dalam cabang BWA-MEM2-LISA: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
Cabang ERT dari repositori BWA-MEM2 berisi basis kode dari akselerasi berbasis pohon radix yang diaktifkan dari BWA-MEM2. Kode ERT dibangun di atas BWA-MEM2 (berkat kerja keras oleh @Arun-Sub). Berikut ini adalah sorotan dari alat BWA-MEM2 berbasis ERT:
Output yang sama persis dengan BWA-MEM (2)
Alat ini memiliki dua bendera tambahan untuk memungkinkan penggunaan solusi ERT (untuk pembuatan indeks dan pemetaan), kalau tidak ia berjalan dalam mode vanilla BWA-MEM2
Ini menggunakan 1 flag tambahan untuk membuat indeks ERT (berbeda dari indeks BWA-MEM2) dan 1 flag tambahan untuk menggunakan indeks ERT itu (silakan lihat readme of ert cabang)
Solusi ERT adalah 10% -30% lebih cepat (diuji pada konfigurasi mesin di atas) dibandingkan dengan vanilla BWA -MEM2 -pengguna disarankan untuk menggunakan opsi -K 1000000 untuk melihat speedups
Cetakan kaki memori dari indeks ERT adalah ~ 60GB
Kode ini ada di cabang ERT: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng LI, Srinivas Aluru. Akselerasi BWA-MEM yang efisien untuk sistem multicore. IEEE Paralel dan Simposium Pemrosesan Terdistribusi (IPDPS), 2019. 10.1109/IPDPS.2019.00041