ANCOM Code Archive
Third release of ANCOM
このリポジトリはアーカイブのみを目的としていることに注意してください。このリポジトリ内のスタンドアロン ANCOM 機能は維持されなくなりました。 ANCOMBC R パッケージのancomまたはancombc関数を使用することを強くお勧めします。バグレポートについては、ANCOMBC リポジトリにアクセスしてください。
現在のコードは、共変量の使用を許可しながら、断面および縦断データセットに ANCOM を実装します。 R コードを実行するには、次のライブラリを含める必要があります。
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )微分存在量分析を実行する前に、さまざまなタイプのゼロを処理するための前処理ステップとして ANCOM-II の方法論を採用しました。
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : 行 ( rownames ) に分類群、列 ( colnames ) にサンプルを含む観測された OTU/SV テーブルを表すデータ フレームまたは行列。これは絶対的な存在量テーブルであり、相対的な存在量テーブル (列の合計が 1 に等しい) に変換しないことに注意してください。meta_data : 対象となるすべての変数および共変量のデータ フレームまたは行列。sample_var : 文字。サンプル ID を格納する列の名前。group_var : 文字。グループインジケーターの名前。 group_var構造ゼロと外れ値を検出するために必要です。さまざまなゼロ (構造ゼロ、外れ値ゼロ、サンプリング ゼロ) の定義については、ANCOM-II を参照してください。out_cut 0 と 1 の間の数値の小数。分類群ごとに、混合分布の割合がout_cutより小さい観測値は外れ値ゼロとして検出されます。一方、混合分布の割合が1 - out_cutより大きい観測値は外れ値として検出されます。zero_cut : 0 と 1 の間の数値の小数。ゼロの割合がzero_cutより大きい分類群は分析に含まれません。lib_cut : 数値。ライブラリ サイズがlib_cut未満のサンプルは分析に含まれません。neg_lb : 論理的。 TRUE は、漸近下限を使用して分類群が対応する実験グループ内の構造ゼロとして分類されることを示します。より具体的には、 neg_lb = TRUE構造ゼロを検出するために ANCOM-II のセクション 3.2 に記載されている両方の基準を使用していることを示します。それ以外の場合、 neg_lb = FALSE構造ゼロの宣言に ANCOM-II のセクション 3.2 の式 1 のみを使用します。 feature_table : 前処理された OTU テーブルのデータ フレーム。meta_data : 前処理されたメタデータのデータ フレーム。structure_zeros : 行列は 0 と 1 で構成され、1 は分類群が対応するグループ内で構造ゼロとして識別されることを示します。ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : 行 ( rownames ) に分類群、列 ( colnames ) にサンプルを含む OTU/SV テーブルを表すデータ フレーム。これは、 feature_table_pre_processからの出力値である可能性があります。これは絶対的な存在量テーブルであり、相対的な存在量テーブル (列の合計が 1 に等しい) に変換しないことに注意してください。meta_data : 変数のデータフレーム。 feature_table_pre_processからの出力値を指定できます。struc_zero : 行列は 0 と 1 で構成され、1 は分類群が対応するグループ内の構造ゼロとして識別されることを示します。 feature_table_pre_processからの出力値を指定できます。main_var : 文字。対象となる主な変数の名前。 ANCOM v2.1 は現在、カテゴリカルmain_varをサポートしています。p_adjust_method : 文字。多重比較の p 値を調整する方法を指定します。デフォルトは「BH」(Benjamini-Hochberg 手順)です。alpha : 重要度。デフォルトは 0.05 です。adj_formula : 調整式を表す文字列(例を参照)。rand_formula : lmeの変量効果の計算式を表す文字列。詳細については、 ?lmeを参照してください。lme_control : lme Fit の制御値を指定するリスト。詳細については、 ?lmeControlを参照してください。 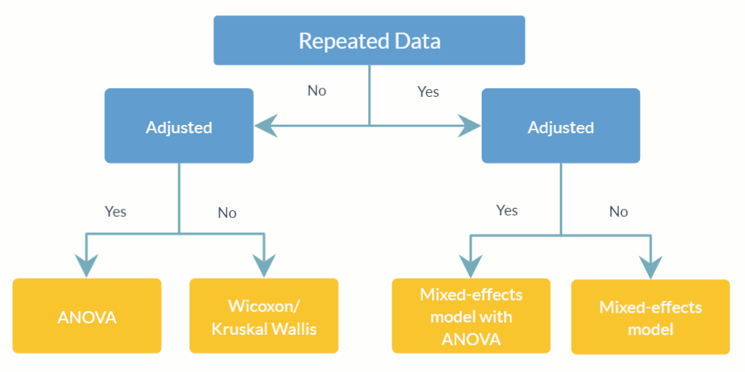
out : 各分類群のW統計値と後続の列を含むデータ フレーム。これらは、OTU または分類群が一連のカットオフ (0.9、0.8、0.7、および 0.6) の下で差次的に豊富であるかどうかを示す論理指標です。 detected_0.7が一般的に使用されます。ただし、結果をより保守的にしたい場合 (FDR が小さい場合)、 detected_0.8 detected_0.9選択するか、より多くの発見を調査したい場合 (検出力が大きい場合)、 detected_0.6使用できます。
fig : volcano プロットのggplotオブジェクト。
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var指定して構造ゼロを識別します。ここでは、さまざまなdeliveryレベルにわたって構造上のゼロがあるかどうかを知りたいと考えています。 library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var指定して構造ゼロを識別します。ここでは、さまざまなdeliveryレベルにわたって構造上のゼロがあるかどうかを知りたいと考えています。 library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var指定して構造ゼロを識別します。ここでは、さまざまなdeliveryレベルにわたって構造上のゼロがあるかどうかを知りたいと考えています。 library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )