このパッケージは、肺セグメンテーション用のトレーニング済み U-net モデルを提供します。現時点では、次の 4 つのモデルが利用可能です。
U-net(R231): このモデルは、広範囲の視覚的変動をカバーする大規模で多様なデータセットでトレーニングされました。モデルは個々のスライスでセグメンテーションを実行し、空気ポケット、腫瘍、胸水を含む左右の肺を別々に抽出します。気管は肺のセグメンテーションには含まれません。 https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): このモデルは、LTRC データセットのサブセットでトレーニングされました。このモデルは個々の肺葉のセグメンテーションを実行しますが、高密度の病変が存在する場合、またはすべてのスライスで亀裂が表示されない場合、パフォーマンスは制限されます。
U-net(LTRCLobes_R231): これにより、R231 および LTRCLobes モデルが実行され、結果が融合されます。 LTRCLobes からの偽陰性は R231 予測によって埋められ、隣接ラベルにマッピングされます。 LTRCLobes からの誤検知は削除されます。融合プロセスは大量の計算を必要とするため、データと結果によっては、ボリュームごとに最大で数分かかる場合があります。
U-net(R231CovidWeb)
2 つのモデルを適用した例。左: U-net(R231) は、左右の肺を区別し、胸水 (3 行目)、腫瘍または重度の線維症 (4 行目) などの非常に密度の高い領域を含みます。右: U-net(LTRLobes)、肺葉を区別しますが、非常に密度の高い領域は含まれません。 LTRCLobes_R231 は、LTRCLobes と R231 の結果を融合します。 R231CovidWeb は、追加の COVID-19 データを使用してトレーニングされています。
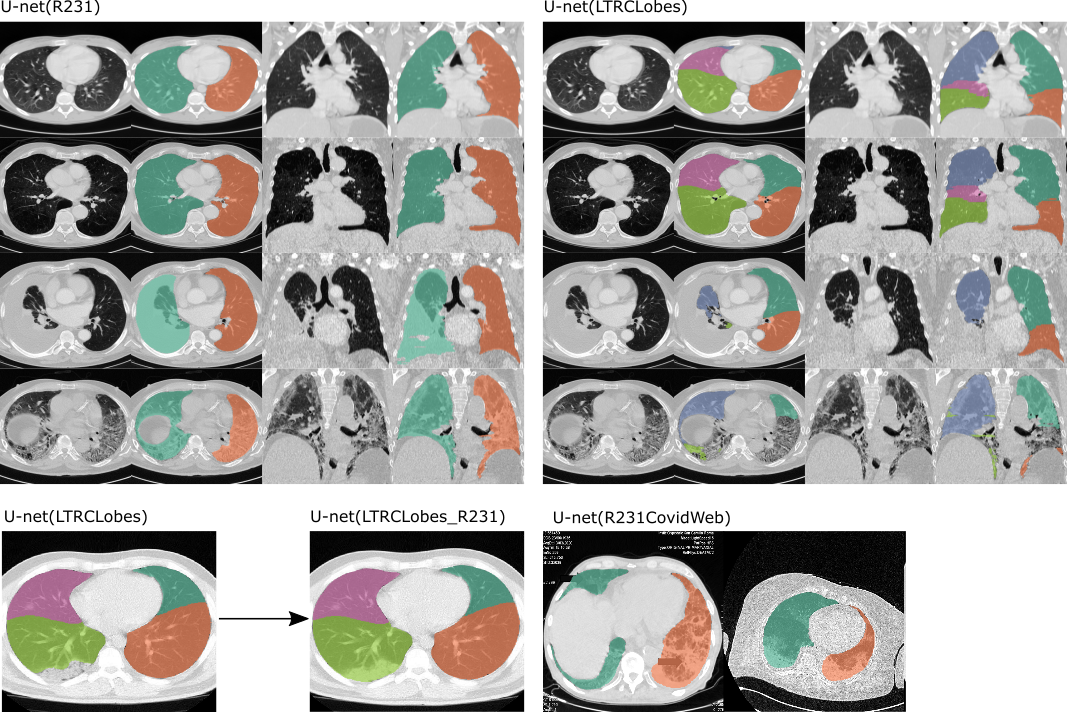
出力のセマンティクス:
2 つのラベル モデル (左-右):
1 = 右肺
2 = 左肺
5 つのラベル モデル (肺葉):
1 = 左上葉
2 = 左下葉
3 = 右上葉
4 = 右中葉
5 = 右下葉
肺 CT データに関するさらに刺激的な研究については、私たちの研究グループの Web サイトをチェックしてください: https://www.cir.meduniwien.ac.at/research/lung/
このコードまたはトレーニング済みモデルのいずれかを作業で使用する場合は、以下を参照してください。
Hofmanninger, J.、Prayer, F.、Pan, J. 他日常的な画像処理における肺の自動セグメンテーションは、主にデータの多様性の問題であり、方法論の問題ではありません。ユーロラジオルExp 4、50(2020)。 https://doi.org/10.1186/s41747-020-00173-2
このペーパーには、使用したデータセットの詳細な説明、U-net(R231) モデルの徹底的な評価、および参照手法との比較が含まれています。
pip install lungmask
または
pip install git+https://github.com/JoHof/lungmask
Windows では、設定によっては、事前に torch をインストールする必要がある場合があります: https://pytorch.org
CPU のみの推論と GPU でサポートされる推論の間の実行時間は大きく異なります。 GPU を使用するとボリュームの処理にかかる時間はわずか数秒ですが、CPU のみを使用すると数分かかります。 GPU を利用するには、トーチのインストールが CUDA をサポートしていることを確認してください。 cuda のメモリ不足エラーが発生した場合は、オプションの引数--batchsize 1を使用してバッチサイズを 1 に減らします。
lungmask INPUT OUTPUT
INPUT がファイルを指している場合、そのファイルが処理されます。 INPUT がディレクトリを指している場合、そのディレクトリで DICOM シリーズが検索されます。見つかった最大ボリューム (ボクセル数に関して) が肺マスクの計算に使用されます。 OUTPUT は出力ファイル名です。すべての ITK 形式がサポートされています。
モデルを選択してください:
デフォルトではU-net(R231)が使用されます。ただし、LTRCLobes などの代替モデルを指定することもできます。
lungmask INPUT OUTPUT --modelname LTRCLobes
追加のオプションについては、次のように入力します。
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image は SimpleITK オブジェクトである必要があります。
次のように代替モデルをロードします。
inferer = LMInferer(modelname="R231CovidWeb")
(例: LTRCLobes_R231) のモデル融合機能を使用するには、以下を使用します。
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
バージョン 0.2.9 では、numpy 配列が入力ボリュームとしてサポートされています。このモードは、入力 numpy 配列が各軸に対して次の形式であることを前提としています。
モデルは完全なスライスでのみ機能します。処理するスライスは肺全体を示す必要があり、セグメント化するには肺が組織で囲まれている必要があります。ただし、肺が組織に囲まれている限り、このモデルは視野が切り取られた場合でも非常に安定しています。
lungmask INPUT OUTPUT --modelname R231CovidWeb
通常の U-net(R231) モデルは、新型コロナウイルス感染症 (COVID-19) の CT スキャンに非常に適しています。ただし、Web からのスライスのコレクションや症例レポートは、通常の画像形式で切り取られたり、注釈が付けられたり、エンコードされたりすることが多いため、元のハウンズフィールド単位 (HU) 値を推定することしかできません。 U-net(R231CovidWeb) モデルのトレーニング データは、通常の画像形式から HU にマップバックされた COVID-19 スライスで強化されました。データは MedSeg (http://medicalsegmentation.com/covid19/) によって収集および作成されました。通常の U-net(R231) はこれらの画像に対して非常に良好な結果を示しましたが、このモデルではセグメンテーションがわずかに改善される場合があります。 Web からの画像を使用する場合は、画像を HU にマップし直す必要があることに注意してください。このブログ投稿では、その方法について説明します。あるいは、 --noHUタグを設定することもできます。 
この機能は、0.2.5 ~ 0.2.14 のバージョンでのみ使用できます。バージョン 0.2.5 では、これらのイメージがサポートされています。 HU でエンコードされていない画像を処理する場合は、 --noHUタグを使用します。モデルは HU でエンコードされた適切な CT スキャンでトレーニングされたことに留意してください。トリミングされ、注釈が付けられ、非常に高い、または非常に低い強度にシフトされた画像の結果は、あまり信頼できない可能性があります。 --noHUタグを使用する場合、単一のスライスのみを処理できます。