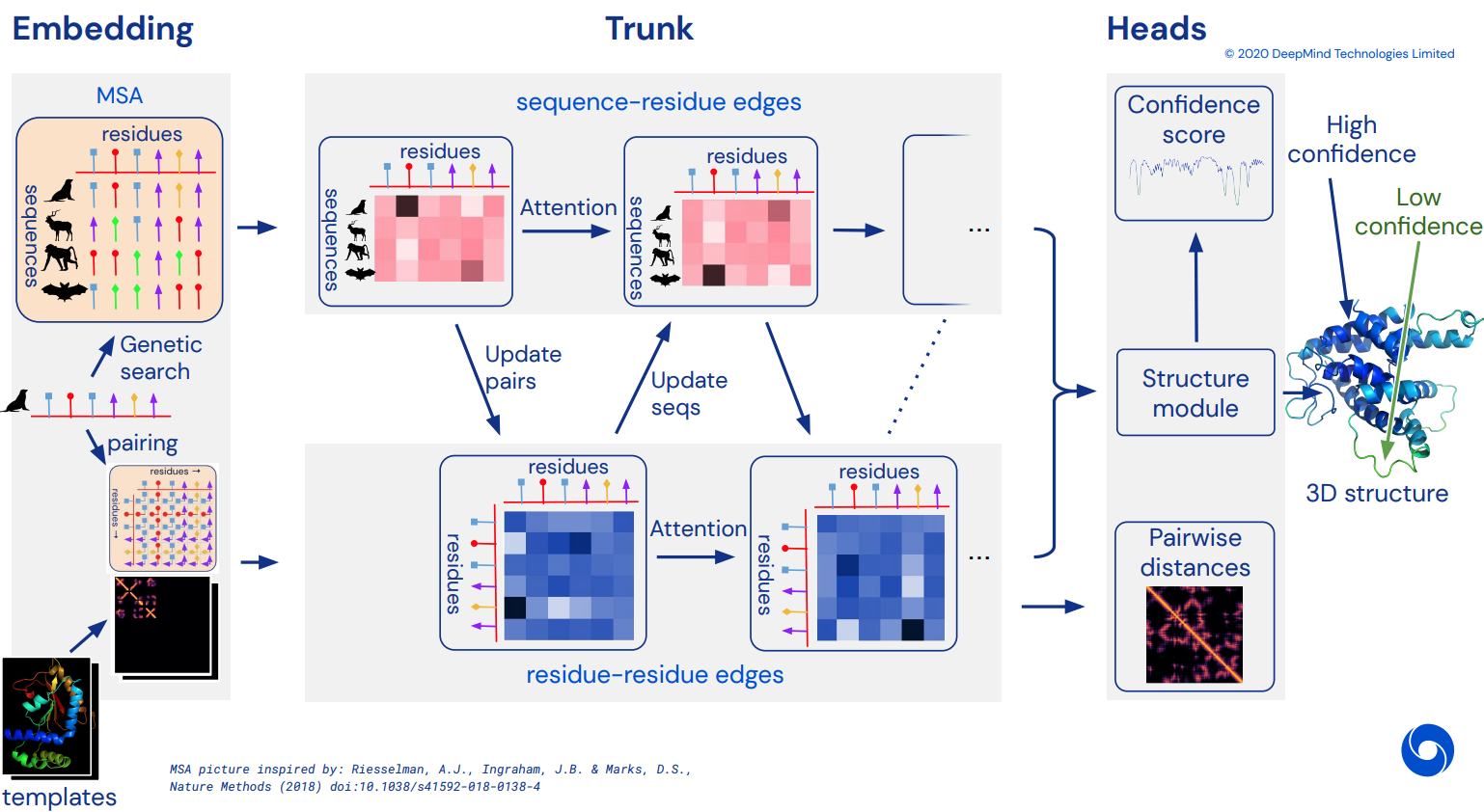
最終的には、CASP14 を解決した驚異的な注目ネットワークである Alphafold2 の非公式に動作する Pytorch 実装になります。アーキテクチャの詳細がリリースされるにつれて、徐々に実装されます。
これが再現されたら、利用可能なすべてのアミノ酸配列をインシリコで折り畳んで、学術の急流として公開し、科学をさらに発展させたいと考えています。複製の取り組みに興味がある場合は、この Discord チャンネルの #alphafold にお立ち寄りください。
更新: Deepmind は、Jax の公式コードと重みをオープンソース化しました。このリポジトリは、位置エンコーディングがいくつか改善された、ストレートな pytorch 翻訳を対象としています。
ArxivInsights ビデオ
$ pip install alphafold2-pytorchlhatsk は、trRosetta と同じセットアップを使用して、このリポジトリの変更されたトランクをトレーニングし、競合する結果を得たと報告しました。
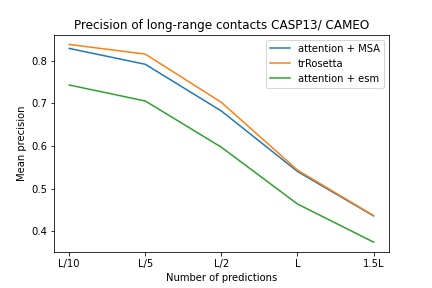
blue used the the trRosetta input (MSA -> potts -> axial attention), green used the ESM embedding (only sequence) -> tiling -> axial attention 。
Alphafold-1 のようなディストグラムを予測しますが、注意が必要です
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
reversible = False # set this to True for fully reversible self / cross attention for the trunk
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda () # AA length of 128
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda () # MSA doesn't have to be the same length as primary sequence
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) init でpredict_angles = Trueを渡すことで、角度の予測をオンにすることもできます。以下の例は trRosetta と同等ですが、セルフ/クロスアテンションが付いています。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_angles = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram , theta , phi , omega = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
)
# distogram - (1, 128, 128, 37),
# theta - (1, 128, 128, 25),
# phi - (1, 128, 128, 13),
# omega - (1, 128, 128, 25) Fabian の最近の論文は、重みを共有して座標を SE3 Transformer に繰り返しフィードバックすることが機能する可能性があることを示唆しています。実際にどうなるかはまだ未定ですが、私はこのアイデアに基づいて実行することにしました。
E(n)-Transformer または EGNN を使用して構造を改善することもできます。
更新: Baker の研究室は、シーケンスおよび MSA 埋め込みから SE3 Transformer までのエンドツーエンド アーキテクチャが trRosetta を上回り、Alphafold2 との差を縮めることができることを示しました。トランクの埋め込みに作用するグラフ トランスフォーマーを使用して、等変ネットワークに送信する初期の座標セットを生成します。 (これは、Baker 研究所以前の論文で MSA Transformer の埋め込みから 3D 座標を取り出した Costa らの研究によってさらに裏付けられています)
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
structure_module_type = 'se3' , # use SE3 Transformer - if set to False, will use E(n)-Transformer, Victor and Max Welling's new paper
structure_module_dim = 4 , # se3 transformer dimension
structure_module_depth = 1 , # depth
structure_module_heads = 1 , # heads
structure_module_dim_head = 16 , # dimension of heads
structure_module_refinement_iters = 2 , # number of equivariant coordinate refinement iterations
structure_num_global_nodes = 1 # number of global nodes for the structure module, only works with SE3 transformer
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue 基礎となる仮定は、トランクが残基レベルで動作し、その後、精製を行う SE3 Transformer、E(n)-Transformer、または EGNN のいずれであっても、構造モジュールの原子レベルを構成するということです。このライブラリのデフォルトは 3 つのバックボーン原子 (C、Ca、N) ですが、Cb やサイドチェーンなど、他の任意の原子を含めるように構成できます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
atoms = 'backbone-with-cbeta'
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 4, 3) <-- 4 atoms per residue (C, Ca, N, Cb) atomsの有効な選択肢は次のとおりです。
backbone - 3 つのバックボーン原子 (C、Ca、N) [デフォルト]backbone-with-cbeta - 3 つのバックボーン原子と C ベータbackbone-with-oxygen - 3 つの主鎖原子とカルボキシルからの酸素backbone-with-cbeta-and-oxygen - C ベータと酸素を含む 3 つの骨格原子all - バックボーンとサイドチェーンの他のすべての原子どの原子を含めたいかを定義する形状 (14,) のテンソルを渡すこともできます。
元。
atoms = torch . tensor ([ 1 , 1 , 1 , 1 , 1 , 1 , 0 , 1 , 0 , 0 , 0 , 0 , 0 , 1 ])このリポジトリは、Facebook AI からの事前トレーニングされた埋め込みを使用してネットワークを簡単に補完できます。これには、事前トレーニングされた ESM、MSA Transformer、または Protein Transformer のラッパーが含まれています。
いくつかの前提条件があります。事前トレーニングされたトランスフォーマーはいくつかの融合操作を利用するため、Nvidia の apex ライブラリがインストールされていることを確認する必要があります。
または、以下のスクリプトを実行してみることもできます
git clone https://github.com/NVIDIA/apex
cd apex
pip install -v --disable-pip-version-check --no-cache-dir --global-option= " --cpp_ext " --global-option= " --cuda_ext " ./次に、 Alphafold2インスタンスをESMEmbedWrapper 、 MSAEmbedWrapper 、またはProtTranEmbedWrapperでインポートしてラップするだけで、シーケンスと複数シーケンスのアライメントの両方の埋め込みが自動的に行われます (そして、それを、モデル)。ラッパーを追加する以外は何も変更する必要はありません。
import torch
from alphafold2_pytorch import Alphafold2
from alphafold2_pytorch . embeds import MSAEmbedWrapper
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64
)
model = MSAEmbedWrapper (
alphafold2 = alphafold2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
)デフォルトでは、ラッパーがトランクにシーケンスと MSA 埋め込みを提供したとしても、それらは通常のトークン埋め込みと合計されます。トークン埋め込みなしで Alphafold2 をトレーニングする場合 (事前トレーニングされた埋め込みのみに依存する)、 Alphafold2初期化でdisable_token_embed Trueに設定する必要があります。
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
disable_token_embed = True
)Jinbo Xu の論文は、距離をビン化する必要がなく、代わりに平均と標準偏差を直接予測できることを示唆しています。これは、 predict_real_value_distancesフラグをオンにすることで使用できます。その場合、返される距離予測の平均と標準偏差の次元はそれぞれ2になります。
predict_coordsもオンになっている場合、MDS は、ディストグラム ビンから平均値と標準偏差の予測を計算することなく、平均値と標準偏差の予測を直接受け入れます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
predict_real_value_distances = True , # set this to True
structure_module_type = 'se3' ,
structure_module_dim = 4 ,
structure_module_depth = 1 ,
structure_module_heads = 1 ,
structure_module_dim_head = 16 ,
structure_module_refinement_iters = 2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue 追加のキーワード引数use_conv = Trueを 1 つ設定するだけで、プライマリ シーケンスと MSA の両方に畳み込みブロックを追加できます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)畳み込みカーネルはこの論文の先導に従い、1 次元カーネルと 2 次元カーネルを 1 つの resnet のようなブロックに結合します。カーネル自体を完全にカスタマイズできます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
conv_seq_kernels = (( 9 , 1 ), ( 1 , 9 ), ( 3 , 3 )), # kernels for N x N primary sequence
conv_msa_kernels = (( 1 , 9 ), ( 3 , 3 )), # kernels for {num MSAs} x N MSAs
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)追加のキーワード引数を 1 つ使用してサイクル拡張を行うこともできます。デフォルトの拡張はすべてのレイヤーで1です。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
dilations = ( 1 , 3 , 5 ) # cycle between dilations of 1, 3, 5
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)最後に、畳み込み、セルフ アテンション、深度ごとのクロス アテンションの繰り返しのパターンに従う代わりに、 custom_block_typesキーワードを使用して任意の順序をカスタマイズできます。
元。最初に主に畳み込みを実行し、次にセルフ アテンション + クロス アテンション ブロックを実行するネットワーク
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
heads = 8 ,
dim_head = 64 ,
custom_block_types = (
* (( 'conv' ,) * 6 ),
* (( 'self' , 'cross' ) * 6 )
)
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Microsoft Deepspeed の Sparse Attend を使用してトレーニングすることはできますが、インストール プロセスに耐える必要があります。それは2段階です。
まず、Sparse Attendant を使用して Deepspeed をインストールする必要があります
$ sh install_deepspeed.sh次に、pip パッケージtritonインストールする必要があります
$ pip install triton上記の両方が成功した場合は、Sparse tention でトレーニングできるようになります。
悲しいことに、まばらな注意は自己注意に対してのみサポートされており、交差注意にはサポートされていません。クロスアテンションのパフォーマンスを向上させるための別のソリューションを導入します。
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
max_seq_len = 2048 , # the maximum sequence length, this is required for sparse attention. the input cannot exceed what is set here
sparse_self_attn = ( True , False ) * 6 # interleave sparse and full attention for all 12 layers
). cuda ()また、相互参加の負担を軽減することを期待して、最も優れたリニア アテンション バリアントの 1 つを追加しました。私個人としては、Performer がそれほどうまく機能するとは思いませんでしたが、論文の中でタンパク質のベンチマークとして問題のない数値が報告されていたため、これを含めて他の人が実験できるようにしようと思いました。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = True # simply set this to True to use Performer for all cross attention
). cuda ()深さと同じ長さのタプルを渡すことで、線形アテンションを使用したい正確なレイヤーを指定することもできます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = ( True , False ) * 3 # interleave linear and full attention
). cuda ()この論文では、軸 (画像など) を定義したクエリまたはコンテキストがある場合、それらの軸 (高さと幅) を平均し、平均した軸を 1 つのシーケンスに連結することで、必要な注意の量を減らすことができることを提案しています。これは、クロス アテンション、特にプライマリ シーケンスのメモリ節約手法としてオンにできます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_kron_primary = True # make sure primary sequence undergoes the kronecker operator during cross attention
). cuda () MSA が整列され、同じ幅であれば、 cross_attn_kron_msaフラグを使用してクロス アテンション中に同じ演算子を MSA に適用することもできます。
藤堂
クロスアテンションのためにメモリを節約するには、このペーパーで説明されているスキームに従って、キー/値の圧縮率を設定できます。通常、2 ~ 4 の圧縮率が許容されます。
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_compress_ratio = 3
). cuda ()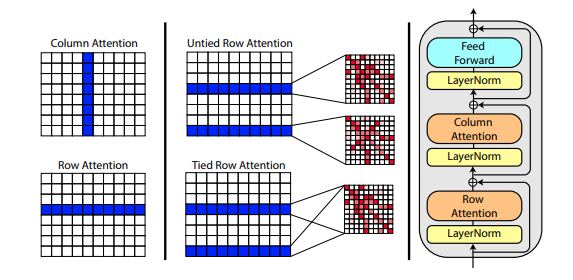
Roshan Rao による新しい論文では、MSA の事前トレーニングに軸方向の注意を使用することを提案しています。強力な結果を考慮して、このリポジトリは、特に MSA セルフ アテンションのために、トランク内で同じスキームを使用します。
Alphafold2の初期化時にmsa_tie_row_attn = True設定を使用して MSA の行アテンションを結び付けることもできます。ただし、これを使用するには、プライマリ シーケンスごとの MSA の数が奇数である場合、使用されていない行に対して MSA マスクが適切にFalseに設定されていることを確認する必要があります。
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
msa_tie_row_attn = True # just set this to true
)テンプレートの処理も主に軸方向の注意で行われ、テンプレートの数の次元に沿って横方向の注意が行われます。これは、ここで示したビデオ分類に対する最近の全注意アプローチとほぼ同じスキームに従います。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randint ( 0 , 37 , ( 1 , 2 , 16 , 3 )). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_coors = templates_coors ,
templates_mask = templates_mask
)各残基の C 座標と C-α 座標の間の単位ベクトルの形式でサイドチェーン情報も存在する場合は、次のように渡すこともできます。
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randn ( 1 , 2 , 16 , 3 ). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
templates_sidechains = torch . randn ( 1 , 2 , 16 , 3 ). cuda () # unit vectors of difference of C and C-alpha coordinates
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_mask = templates_mask ,
templates_coors = templates_coors ,
templates_sidechains = templates_sidechains
)Fabian Fuchs が推測的なブログ投稿で説明したように、私は SE3 Transformer の再実装を準備しました。
さらに、Victor と Welling の新しい論文では、E(n) 等分散に不変特徴を使用しており、SOTA に到達し、多くのベンチマークで SE3 Transformer を上回るパフォーマンスを示しながら、はるかに高速です。私はこの論文から主なアイデアを取り入れ、それをトランスフォーマーになるように修正しました (機能と座標の更新の両方に注意を追加しました)。
上記の 3 つの等変ネットワークはすべて統合されており、1 つのハイパーパラメーターstructure_module_type設定するだけで、原子座標改良用のリポジトリで使用できます。
se3 SE3 トランスフォーマー
egnnえぐん
en E(n)-トランスフォーマー
読者にとって興味深いのは、3 つのフレームワークのそれぞれが、関連する問題に関して研究者によって検証されていることです。
$ python setup.py test このライブラリは、このリポジトリにある Jonathan King による素晴らしい作品を使用します。ジョナサン、ありがとう!
また、The-Eye プロジェクトを所有するアーキビストによってダウンロードおよびホストされている、すべて約 3.5 TB 相当の MSA データもあります。 (Eleuther AI のデータとモデルもホストしています) 役に立ったと思われる場合は、寄付をご検討ください。
$ curl -s https://the-eye.eu/eleuther_staging/globus_stuffs/tree.txthttps://xukui.cn/alphafold2.html
https://moalqraishi.wordpress.com/2020/12/08/alphafold2-casp14-it-feels-like-ones-child-has-left-home/

https://www.biorxiv.org/content/10.1101/2020.12.10.419994v1.full.pdf
https://pubmed.ncbi.nlm.nih.gov/33637700/
Tencent AI labs による tFold プレゼンテーション
cd downloads_folder > pip install pyrosetta_wheel_filename.whlOpenMM アンバー
@misc { unpublished2021alphafold2 ,
title = { Alphafold2 } ,
author = { John Jumper } ,
year = { 2020 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @article { Rao2021.02.12.430858 ,
author = { Rao, Roshan and Liu, Jason and Verkuil, Robert and Meier, Joshua and Canny, John F. and Abbeel, Pieter and Sercu, Tom and Rives, Alexander } ,
title = { MSA Transformer } ,
year = { 2021 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/02/13/2021.02.12.430858 } ,
journal = { bioRxiv }
} @article { Rives622803 ,
author = { Rives, Alexander and Goyal, Siddharth and Meier, Joshua and Guo, Demi and Ott, Myle and Zitnick, C. Lawrence and Ma, Jerry and Fergus, Rob } ,
title = { Biological Structure and Function Emerge from Scaling Unsupervised Learning to 250 Million Protein Sequences } ,
year = { 2019 } ,
doi = { 10.1101/622803 } ,
publisher = { Cold Spring Harbor Laboratory } ,
journal = { bioRxiv }
} @article { Elnaggar2020.07.12.199554 ,
author = { Elnaggar, Ahmed and Heinzinger, Michael and Dallago, Christian and Rehawi, Ghalia and Wang, Yu and Jones, Llion and Gibbs, Tom and Feher, Tamas and Angerer, Christoph and Steinegger, Martin and BHOWMIK, DEBSINDHU and Rost, Burkhard } ,
title = { ProtTrans: Towards Cracking the Language of Life{textquoteright}s Code Through Self-Supervised Deep Learning and High Performance Computing } ,
elocation-id = { 2020.07.12.199554 } ,
year = { 2021 } ,
doi = { 10.1101/2020.07.12.199554 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554.full.pdf } ,
journal = { bioRxiv }
} @misc { king2020sidechainnet ,
title = { SidechainNet: An All-Atom Protein Structure Dataset for Machine Learning } ,
author = { Jonathan E. King and David Ryan Koes } ,
year = { 2020 } ,
eprint = { 2010.08162 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { alquraishi2019proteinnet ,
title = { ProteinNet: a standardized data set for machine learning of protein structure } ,
author = { Mohammed AlQuraishi } ,
year = { 2019 } ,
eprint = { 1902.00249 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { gomez2017reversible ,
title = { The Reversible Residual Network: Backpropagation Without Storing Activations } ,
author = { Aidan N. Gomez and Mengye Ren and Raquel Urtasun and Roger B. Grosse } ,
year = { 2017 } ,
eprint = { 1707.04585 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CV }
} @misc { fuchs2021iterative ,
title = { Iterative SE(3)-Transformers } ,
author = { Fabian B. Fuchs and Edward Wagstaff and Justas Dauparas and Ingmar Posner } ,
year = { 2021 } ,
eprint = { 2102.13419 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { satorras2021en ,
title = { E(n) Equivariant Graph Neural Networks } ,
author = { Victor Garcia Satorras and Emiel Hoogeboom and Max Welling } ,
year = { 2021 } ,
eprint = { 2102.09844 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { su2021roformer ,
title = { RoFormer: Enhanced Transformer with Rotary Position Embedding } ,
author = { Jianlin Su and Yu Lu and Shengfeng Pan and Bo Wen and Yunfeng Liu } ,
year = { 2021 } ,
eprint = { 2104.09864 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CL }
} @article { Gao_2020 ,
title = { Kronecker Attention Networks } ,
ISBN = { 9781450379984 } ,
url = { http://dx.doi.org/10.1145/3394486.3403065 } ,
DOI = { 10.1145/3394486.3403065 } ,
journal = { Proceedings of the 26th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining } ,
publisher = { ACM } ,
author = { Gao, Hongyang and Wang, Zhengyang and Ji, Shuiwang } ,
year = { 2020 } ,
month = { Jul }
} @article { Si2021.05.10.443415 ,
author = { Si, Yunda and Yan, Chengfei } ,
title = { Improved protein contact prediction using dimensional hybrid residual networks and singularity enhanced loss function } ,
elocation-id = { 2021.05.10.443415 } ,
year = { 2021 } ,
doi = { 10.1101/2021.05.10.443415 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415.full.pdf } ,
journal = { bioRxiv }
} @article { Costa2021.06.02.446809 ,
author = { Costa, Allan and Ponnapati, Manvitha and Jacobson, Joseph M. and Chatterjee, Pranam } ,
title = { Distillation of MSA Embeddings to Folded Protein Structures with Graph Transformers } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.02.446809 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809.full.pdf } ,
journal = { bioRxiv }
} @article { Baek2021.06.14.448402 ,
author = { Baek, Minkyung and DiMaio, Frank and Anishchenko, Ivan and Dauparas, Justas and Ovchinnikov, Sergey and Lee, Gyu Rie and Wang, Jue and Cong, Qian and Kinch, Lisa N. and Schaeffer, R. Dustin and Mill{'a}n, Claudia and Park, Hahnbeom and Adams, Carson and Glassman, Caleb R. and DeGiovanni, Andy and Pereira, Jose H. and Rodrigues, Andria V. and van Dijk, Alberdina A. and Ebrecht, Ana C. and Opperman, Diederik J. and Sagmeister, Theo and Buhlheller, Christoph and Pavkov-Keller, Tea and Rathinaswamy, Manoj K and Dalwadi, Udit and Yip, Calvin K and Burke, John E and Garcia, K. Christopher and Grishin, Nick V. and Adams, Paul D. and Read, Randy J. and Baker, David } ,
title = { Accurate prediction of protein structures and interactions using a 3-track network } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.14.448402 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402.full.pdf } ,
journal = { bioRxiv }
}