MedSegDiff は、医療画像セグメンテーション用の拡散確率モデル (DPM) ベースのフレームワークです。このアルゴリズムは、論文「MedSegDiff: 拡散確率モデルを使用した医療画像セグメンテーション」および「MedSegDiff-V2: Transformer を使用した拡散ベースの医療画像セグメンテーション」で詳しく説明されています。
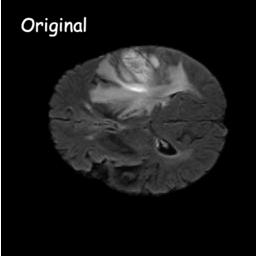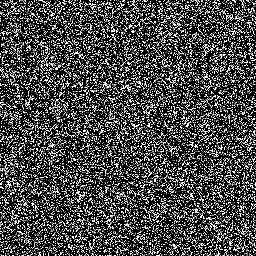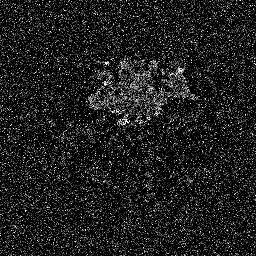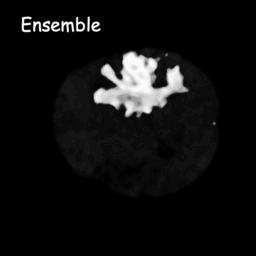 拡散モデルは、ガウス ノイズを連続的に追加することでトレーニング データを破壊し、このノイズ プロセスを逆にしてデータを回復することを学習することで機能します。トレーニング後は、拡散モデルを使用して、ランダムにサンプリングされたノイズを学習されたノイズ除去プロセスに渡すだけでデータを生成できます。このプロジェクトでは、このアイデアを医療画像のセグメンテーションに拡張します。元の画像を条件として利用し、ランダムなノイズから複数のセグメンテーション マップを生成し、それらに対してアンサンブルを実行して最終結果を取得します。このアプローチは医療画像の不確実性を捉え、いくつかのベンチマークで以前の方法よりも優れたパフォーマンスを発揮します。
拡散モデルは、ガウス ノイズを連続的に追加することでトレーニング データを破壊し、このノイズ プロセスを逆にしてデータを回復することを学習することで機能します。トレーニング後は、拡散モデルを使用して、ランダムにサンプリングされたノイズを学習されたノイズ除去プロセスに渡すだけでデータを生成できます。このプロジェクトでは、このアイデアを医療画像のセグメンテーションに拡張します。元の画像を条件として利用し、ランダムなノイズから複数のセグメンテーション マップを生成し、それらに対してアンサンブルを実行して最終結果を取得します。このアプローチは医療画像の不確実性を捉え、いくつかのベンチマークで以前の方法よりも優れたパフォーマンスを発揮します。
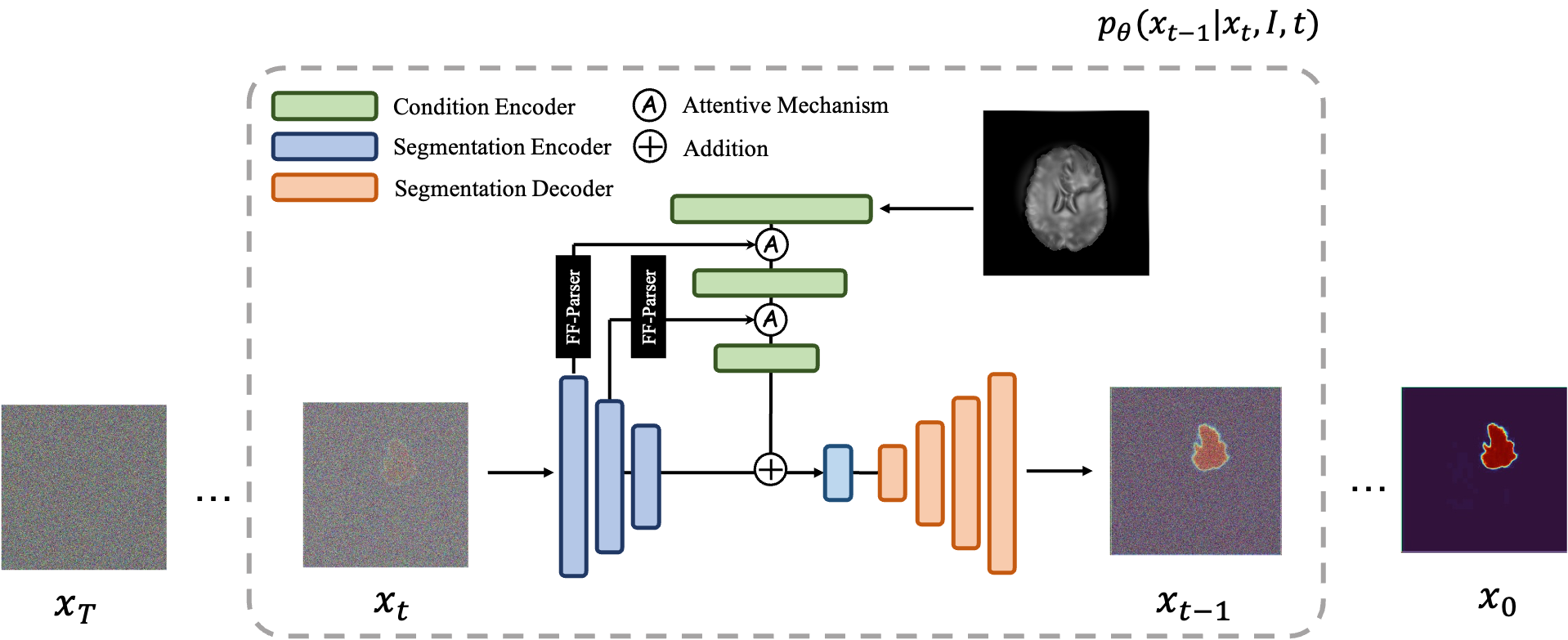 | 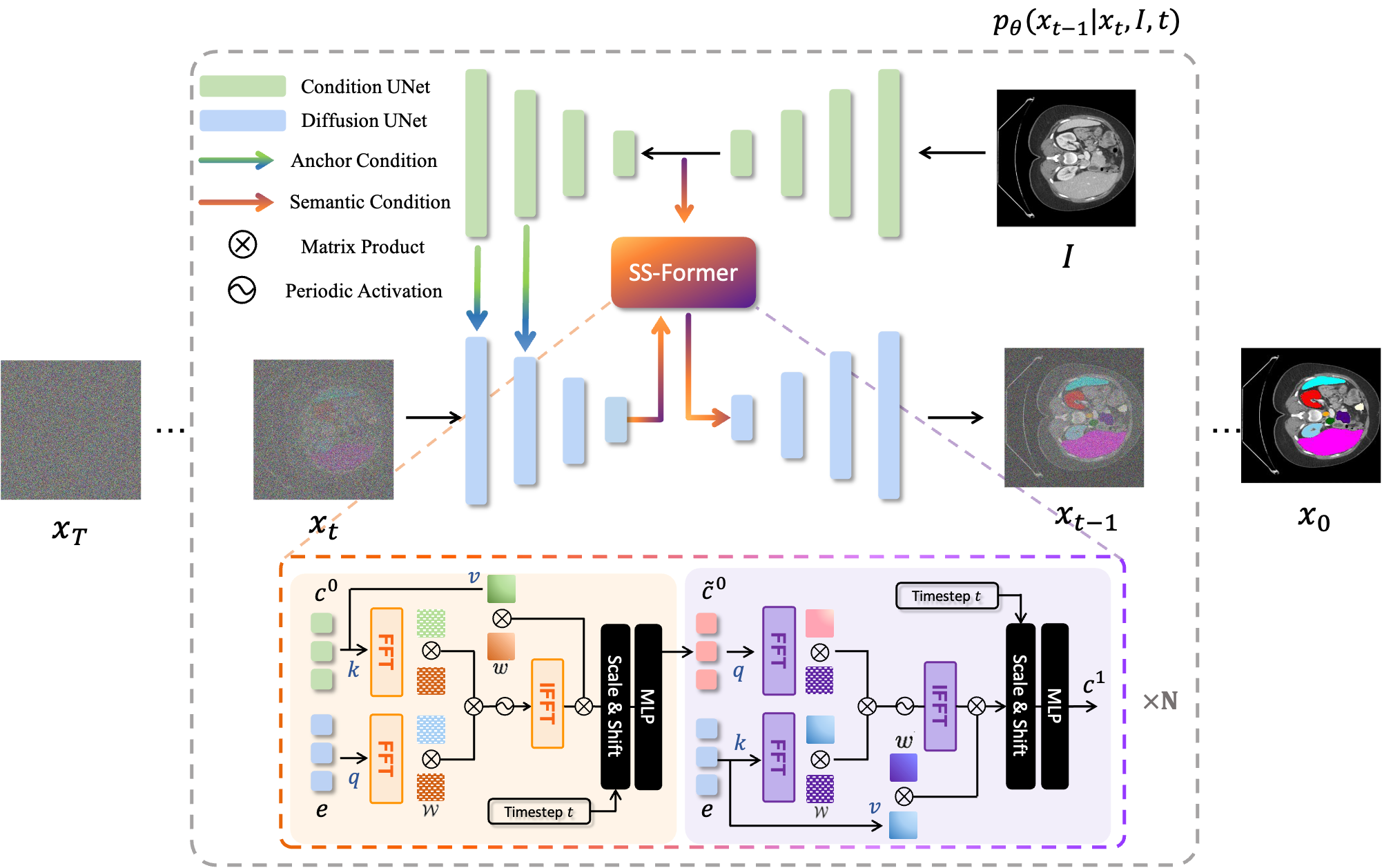 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver Trueを設定して、超高速サンプリング (1000 ステップ 20 ステップ ⭕️) をお楽しみください。python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
トレーニングするには、次を実行します: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
サンプリングの場合は、次を実行します: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
評価するには、 python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
デフォルトでは、サンプルは./results/に保存されます。
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
トレーニングの場合は、次を実行します: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
サンプリングの場合は、次を実行します: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
他のデータセットに対して MedSegDiff を実行するのは簡単です。 ./guided_diffusion/isicloader.pyまたは./guided_diffusion/bratsloader.pyの後に別のデータ ローダー ファイルを作成するだけです。問題が発生した場合は、問題をオープンしてください。データセット拡張機能を提供していただければ幸いです。自然画像とは異なり、医療画像はさまざまなタスクに応じて大きく異なります。メソッドの一般化を拡大するには、全員の努力が必要です。
細かいモデル、つまり論文の MedSegDiff-B をトレーニングするには、モデルのハイパーパラメーターを次のように設定します。
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
拡散ハイパーパラメータは次のとおりです。
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
サンプリングを高速化するには:
--diffusion_steps 50 --dpm_solver True
複数の GPU で実行します。
--multi-gpu 0,1,2 (for example)
ハイパーパラメータを次のようにトレーニングします。
--lr 5e-5 --batch_size 8
そしてサンプリングに--num_ensemble 5設定します。
トレーニングで約 100,000 ステップを実行すると、ほとんどのデータセットに収束します。後のステップのほとんどでは損失は減少しませんが、結果の品質は依然として向上していることに注意してください。このようなプロセスは、イメージ生成などの他の DPM アプリケーションでも観察されます。誰か賢い人が理由を教えてくれるといいのですが?
比較の必要がある場合に備えて、より小さいバッチ サイズ (24GB GPU での実行に適した) でのパフォーマンスを近々公開します。
その可能性を最大限に引き出す設定は (MedSegDiff++) です。
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
次に、バッチ サイズ--batch_size 64でトレーニングし、アンサンブル番号--num_ensemble 25でサンプリングします。
MedSegDiff への貢献を歓迎します。パフォーマンスを向上させたり、アルゴリズムを高速化したりできるあらゆるテクニックは歓迎されます。 Nature ジャーナル/CVPR のような出版を目指して、MedSegDiff V2 を書いています。寄稿者を共著者として記載してもよろしいでしょうか?
コードは openai/improved-diffusion、WuJunde/MrPrism、WuJunde/DiagnosisFirst、LuChengTHU/dpm-solver、JuliaWolleb/Diffusion-based-Segmentation、hojonathanho/diffusion、guided-diffusion、bigmb/Unet-Segmentation-Pytorch-Nest から多くコピーされました。 -of-Unets、nnUnet、 lucidrains/vit-pytorch
引用してください
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu