최종 업데이트 날짜: 2024년 9월 30일
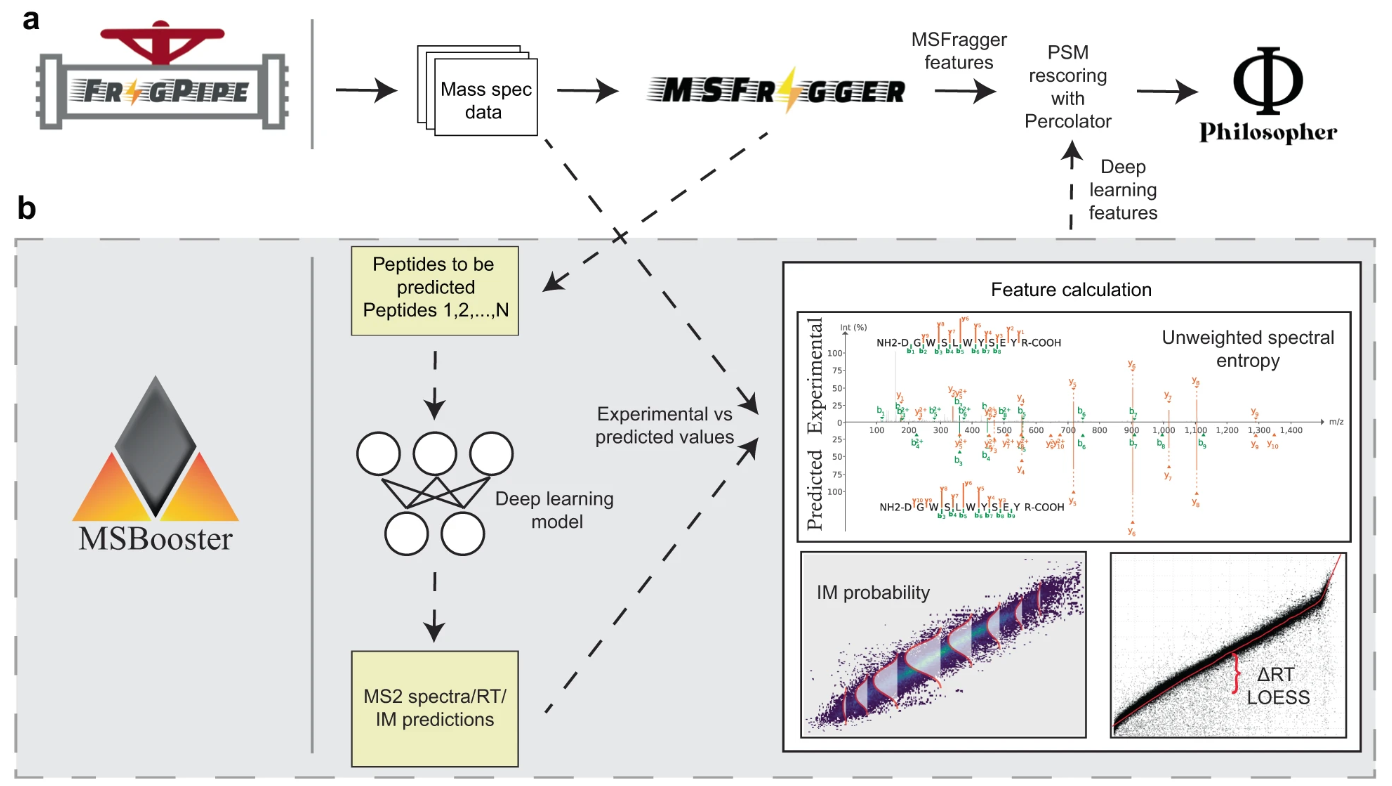
MSBooster는 상향식 탠덤 액체 크로마토그래피 질량 분석법 프로테오믹스 데이터의 점수를 다시 매기는 펩타이드-스펙트럼 일치(PSM)에 스펙트럼 라이브러리 예측을 통합하기 위한 도구입니다. 대략 4단계로 나누어집니다.
검색 결과의 PSM에서 펩타이드 추출 및 머신/딥 러닝(ML/DL) 예측 변수의 입력 파일 형식 지정
예측 모델 호출 및 출력 저장
특징 계산
검색 결과 파일에 새로운 기능 추가
MSBooster는 HLA 면역펩티도믹스, DDA 및 DIA, 단일 세포 단백질체학을 포함한 다양한 유형의 데이터베이스 검색과 호환됩니다. 이는 FragPipe에 통합되어 있으며 많은 워크플로에 포함되어 있습니다. MSBooster는 FragPipe-PDV와 같은 다른 FragPipe 도구를 염두에 두고 개발되었습니다.
MSBooster는 여러 입력 파일 형식 및 모델을 처리할 수 있는 기능을 갖추고 있습니다.
| 질량 분석기 출력 |
|---|
| .mzML |
| .mgf |
| PSM 파일 |
|---|
| .핀 |
| .pepXML(진행 중) |
| 예측 모델 |
|---|
| DIA-NN |
| 코이나 모델 |
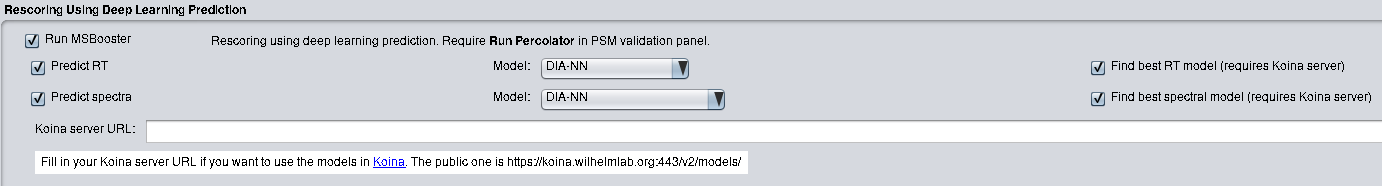
MSBooster는 Windows 및 Linux 시스템에서 실행할 수 있습니다. FragPipe를 사용하는 경우 FragPipe 설치 외에 다른 설치 단계가 필요하지 않습니다. MSBooster는 "검증" 탭에 있습니다. "RT 예측"으로 머무름 시간 기능을 활성화하고 "스펙트럼 예측"으로 MS/MS 스펙트럼 기능을 활성화하도록 선택합니다. 분석 실행 방법은 FragPipe 설명서를 참조하세요.
독립 실행형 MSBooster를 사용하여 명령줄에서 실행하는 경우 릴리스에서 최신 jar 파일을 다운로드하세요. MSBooster에는 MS/MS 및 RT 예측을 위한 DIA-NN도 필요합니다. DIA-NN을 설치하고 DIA-NN 실행 파일 경로를 기록해 두십시오(예: Windows의 경우 DiaNN.exe, Linux의 경우 diann-1.8.1.8).
다음과 유사한 명령을 사용하여 MSBooster를 실행할 수 있습니다.
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
전달해야 하는 최소 매개변수는 다음과 같습니다.
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
이러한 매개변수를 개별적으로 전달할 수도 있지만 paramsList 파일의 각 줄에 매개변수를 배치하는 것이 더 쉽습니다. 템플릿은 msbooster_params.txt를 참조하세요.
아래 매개변수는 일반적인 사용을 위한 것입니다. Koina 관련 매개변수는 Koina 문서에 있습니다.
paramsList (String) : 이 실행에 대한 매개변수가 포함된 텍스트 파일의 위치
fragger (String) : MSFragger 실행에서 fragger.params 파일의 파일 경로입니다. MSBooster는 여러 매개변수를 읽고 이를 기반으로 조각 질량 오류 허용 및 질량 오프셋과 같은 내부 매개변수를 조정합니다.
outputDirectory (String) : 새 파일을 출력할 위치
editedPin (String) : MSBooster는 제공된 파일을 기반으로 새 파일의 이름을 지정합니다. 예를 들어, A.pin에는 A_edited.pin이라는 대응 항목이 있습니다. 기본값인 "편집됨"을 변경하려면 여기에 새 문자열을 제공하세요.
renamePin (int) : 새 핀 파일을 생성할지 아니면 이전 핀 파일을 다시 쓸지 여부입니다. 여기서 기본값은 1이며 덮어쓰지 않습니다. 0으로 설정하면 이전 핀 파일을 덮어쓰게 됩니다.
deletePreds (boolean) : 성공적인 실행을 마친 후 모델 예측을 저장하는 파일을 삭제할지 여부입니다. 기본적으로 false로 설정됩니다. 이를 삭제하려면 true로 설정하세요.
loadingPercent (int) : 진행률 보고를 사용하여 작업 진행률을 보고하는 빈도입니다. 기본적으로 10으로 설정됩니다. 즉, 업데이트가 10%마다 인쇄됩니다.
numThreads (int) : 사용할 스레드 수입니다. 기본적으로 사용 가능한 모든 스레드에서 1을 뺀 값을 사용하는 0으로 설정됩니다.
splitPredInputFile (int) : 메모리 부족 오류(137)로 인해 DIA-NN 예측이 실패한 경우에만 사용됩니다. 기본적으로 1로 설정되지만 이 값을 늘려 DIA-NN 입력 파일을 분할해야 하는 더 작은 파일 수를 지정할 수 있습니다. 그러면 각 파일이 순차적으로 예측되므로 메모리 부담이 줄어듭니다.
plotExtension (String) : 플롯에 포함되어야 하는 파일 형식입니다. 기본적으로 png이며 pdf도 허용됩니다.
features (String) : 계산할 기능 목록입니다. 대소문자를 구분하며, 사이에 공백 없이 com으로 구분됩니다. 기본값은 "predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS"입니다.
spectraPredFile (String) : 이전 스펙트럼 예측(예: DIA-NN 또는 Koina)을 재사용하는 경우 여기에서 파일 위치를 지정할 수 있습니다.
RTPredFile (String) : spectraPredFile과 동일하지만 RT 예측용
IMPredFile (String) : spectraPredFile과 동일하지만 IM 예측용
spectraModel (String) : 사용할 스펙트럼 예측 모델
rtModel (String) : spectraModel과 동일하지만 RT용
imModel (String) : spectraModel과 동일하지만 IM용
useSpectra (boolean) : 스펙트럼 예측 기반 기능을 사용할지 여부입니다. 기본적으로 true로 설정
useRT (boolean) : RT 예측 기반 기능을 사용할지 여부입니다. 기본적으로 true로 설정
useIM (boolean) : IM 예측 기반 기능을 사용할지 여부입니다. 기본적으로 false로 설정
ppmTolerance (float) : 조각 오류ppm 허용 오차(기본값 20ppm)
matchWithDaltons (boolean) : Daltons에서 예측 및 관찰된 조각을 일치시킬지 여부(기본값 false)
DaTolerance (float) : 실험적 피크를 찾기 위해 예측된 피크 주변의 달톤 수(기본값 0.05)
useTopFragments (boolean) : 스펙트럼 예측을 N개의 최고 강도 피크로 필터링할지 여부(기본값은 true)
topFragments (int) : 기능 계산에 사용해야 하는 예측 조각 수까지입니다(기본값 20). useTopFragments가 true인 경우에만 적용됩니다.
removeRankPeaks (boolean) : 기본적으로 true로 설정되어 일치된 실험 스펙트럼에서 조각을 필터링합니다. false인 경우 실험 조각은 동일한 스캔의 여러 PSM과 일치할 수 있습니다.
useBasePeak (boolean) : 더 높은 강도의 조각만 사용하기 위해 MS2 예측에 하한을 적용해야 하는지 여부(기본값은 true)
percentBasePeak (float) : 기본 피크 강도의 몇 퍼센트 강도를 갖는 조각이 유사성 계산에 포함되는 백분율입니다. useBasePeak가 true인 경우에만 적용됩니다(기본값 1).
loessEscoreCutoff (float) : RT/IM 교정을 위한 PSM 수집 시 첫 번째 통과에 사용되는 기대값 컷오프입니다. 기본값은 10^-3.5 또는 약 0.000316입니다.
rtLoessRegressionSize (int) : RT LOESS 교정에 사용되는 최대 PSM 수(기본값 5000)
imLoessRegressionSize (int) : rtLoessRegressionSize와 동일하지만 IM의 경우(기본값 1000)
minLoessRegressionSize (int) : LOESS RT/IM 교정을 시도하는 데 필요한 최소 PSM 수(기본값 100). 이 PSM 수보다 적은 수를 사용할 수 있는 경우 대신 선형 회귀가 사용됩니다.
minLinearRegressionSize (int) : 선형 회귀 RT/IM 교정을 시도하는 데 필요한 최소 PSM 수(기본값 10). 사용 가능한 PSM 수가 이 수보다 적으면 교정이 시도되지 않습니다.
loessBandwidth (String) : RT/IM LOESS 교정을 위해 시도할 대역폭 목록 (기본값 0.01,0.05,0.1,0.2). 사이에 공백 없이 쉼표로 구분해야 합니다.
regressionSplits (int) : RT/IM LOESS 보정에 사용되는 교차 검증 수(기본값 5)
massesForLoessCalibration (String) : 자체 교정 곡선에 맞아야 하는 질량 이동의 질량입니다. 목록은 사이에 공백 없이 쉼표로 구분됩니다. 질량은 PIN 파일과 동일한 자릿수로 작성되어야 합니다.
loessScatterOpacity (float) : LOESS 교정 수치의 산점도 불투명도, 0에서 1까지(기본값 0.35)
새로운 기능이 포함된 .pin 파일. 기본적으로 "_edited.pin"으로 끝나는 새 핀 파일이 생성됩니다. 사용되는 기본 기능은 "unweighted_spectral_entropy", "delta_RT_loess" 및 "pred_RT_real_units"입니다. Ion Mobility 기능이 활성화된 경우 "delta_IM_loess" 및 "ion_mobility"도 포함됩니다.
spectraRT.tsv 및 spectraRT_full.tsv: DIA-NN 예측 모델을 위한 입력 파일
spectraRT.predicted.bin: 기능 계산을 위해 MSBooster에서 사용할 DIA-NN의 예측이 포함된 이진 파일입니다. FragPipe-PDV를 사용하는 경우 이러한 파일은 실험 및 예측 스펙트럼의 미러 플롯을 생성하는 데 사용됩니다.
MSBooster는 데이터가 모델 예측과 어떻게 비교되는지 자세히 조사하는 데 사용할 수 있는 여러 그래프를 생성합니다.
MSBooster_plots 폴더:
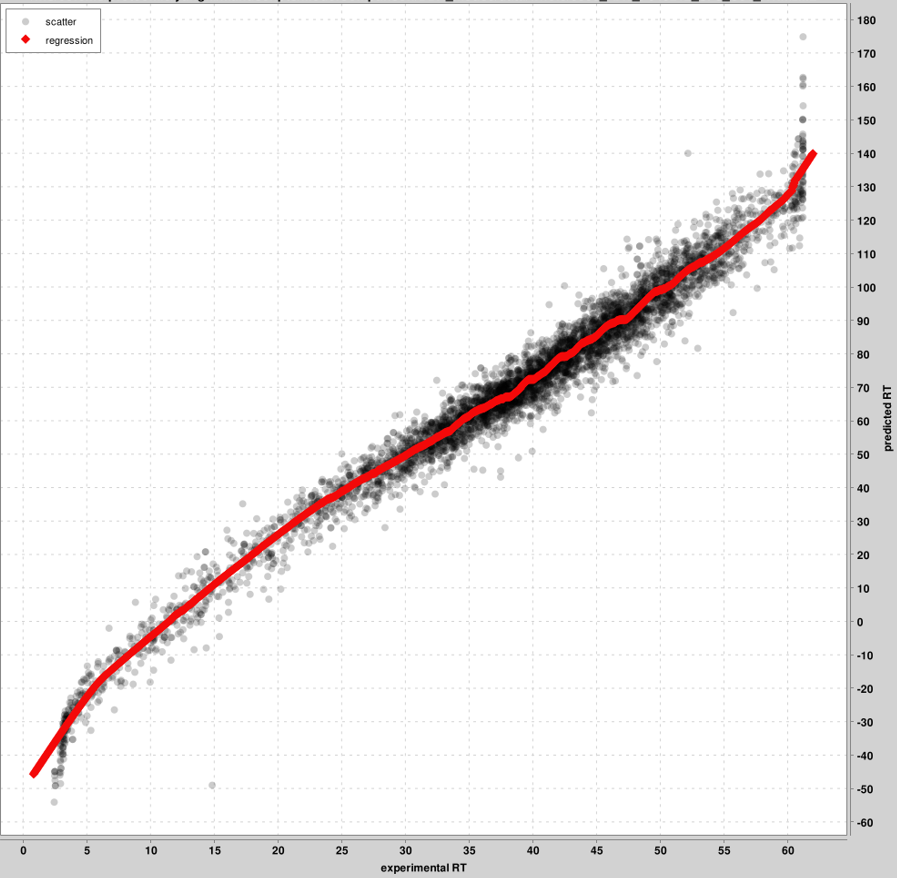
RT_calibration_curves: 최대 5000개의 PSM이 실험 및 예측 RT 척도 간의 교정에 사용됩니다. 모든 PSM이 아닌 이러한 상위 PSM이 그래프에 표시됩니다. 핀 파일당 하나의 그래프가 생성됩니다.
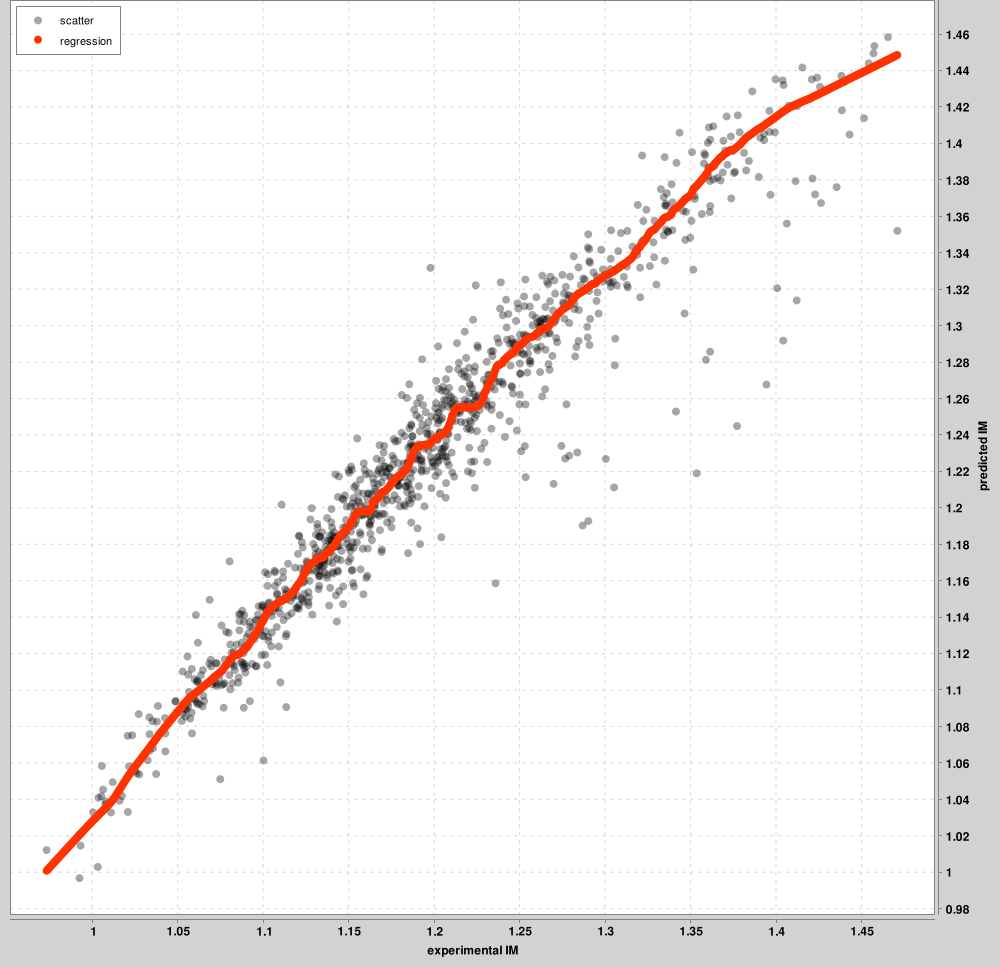
IM_calibration_curves: 최대 1000개의 PSM이 실험 및 예측 IM 척도 간의 교정에 사용됩니다. 모든 PSM이 아닌 이러한 상위 PSM이 그래프에 표시됩니다. 각 충전 상태에 대해 별도의 곡선이 학습됩니다. 아래 그림은 Charge 2 전구체의 예입니다.
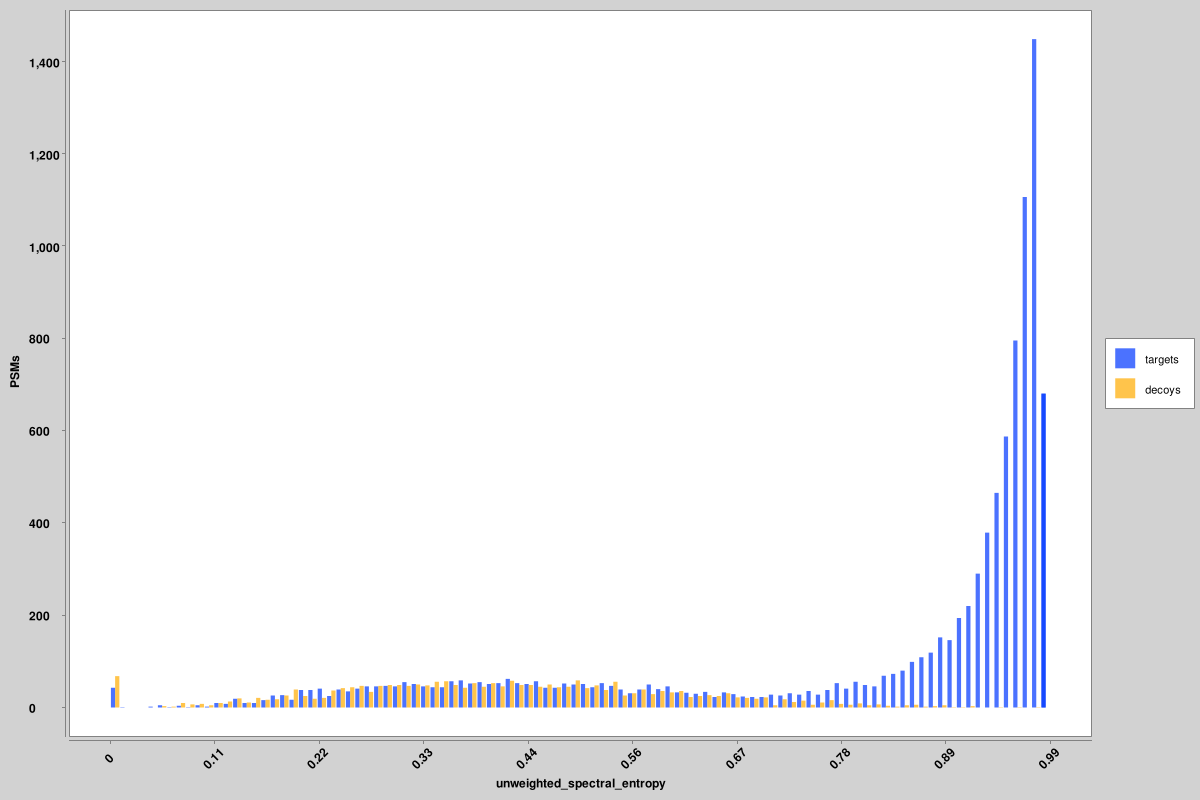
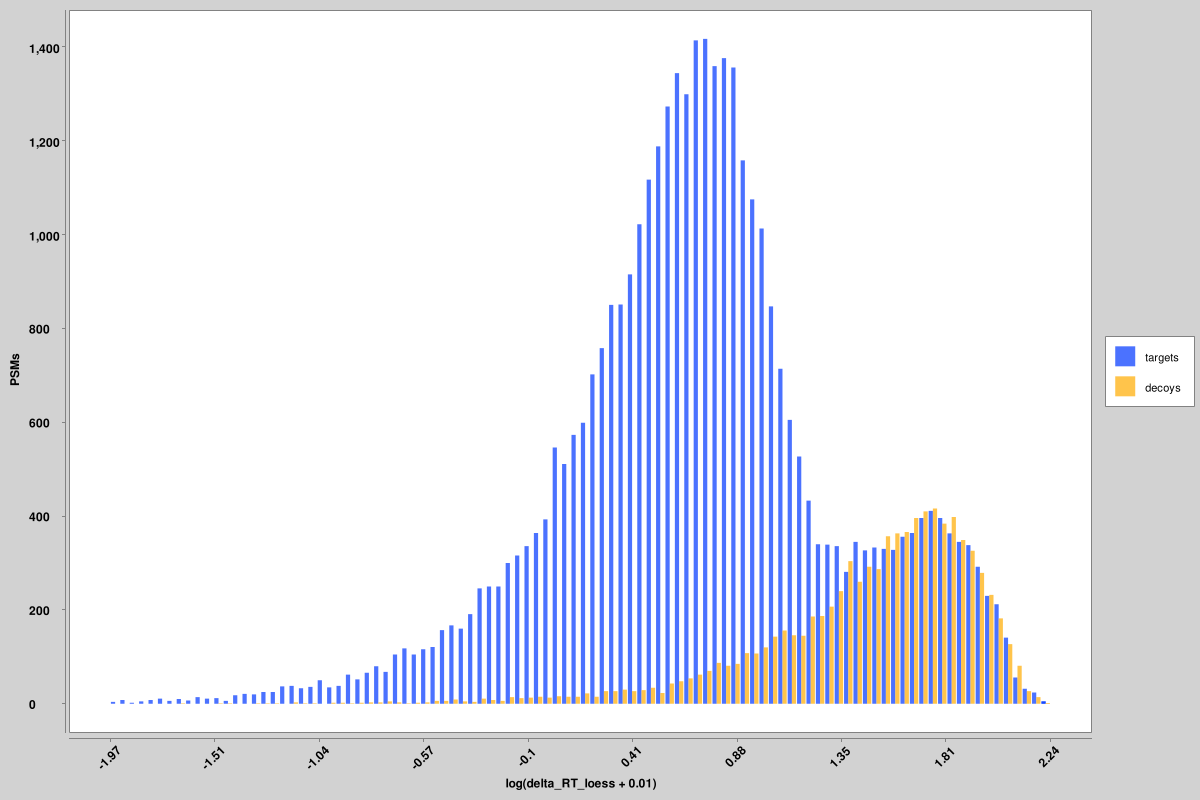
Score_histograms: 각 핀 파일에 대한 모든 대상 및 미끼 PSM의 오버레이된 히스토그램입니다. 일부 기능은 참 긍정과 거짓 긍정의 이중 모드 분포를 더 잘 시각화하기 위해 여기에 로그 스케일로 표시되어 있지만 원래 값은 로그 스케일 버전이 아니라 핀 파일에 사용된 값입니다. 여기에는 비가중 스펙트럼 엔트로피 및 델타 RT 점수에 대한 히스토그램이 표시되어 있지만 모든 기능에 대해 유사한 히스토그램이 생성됩니다.
MSBooster 기능 생성을 위해 Koina의 펩타이드 예측 모델 사용: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
MGF 파일을 통해 모든 모델의 예측 읽기
허용되는 모든 기능에 대한 문서와 그래픽 출력으로 이를 QC하는 방법
MSBooster를 사용할 때 다음을 인용하십시오: https://www.nature.com/articles/s41467-023-40129-9