이것은 "모든 것을 지배하는 하나의 모델: 텍스트 프롬프트를 사용한 의료 이미지의 보편적인 분할을 향하여"의 공식 저장소입니다.
이는 전례 없는 데이터 수집(72개의 공개 3D 의료 세분화 데이터 세트)을 기반으로 구축된 지식 강화 범용 세분화 모델로, 텍스트(해부학 술어).
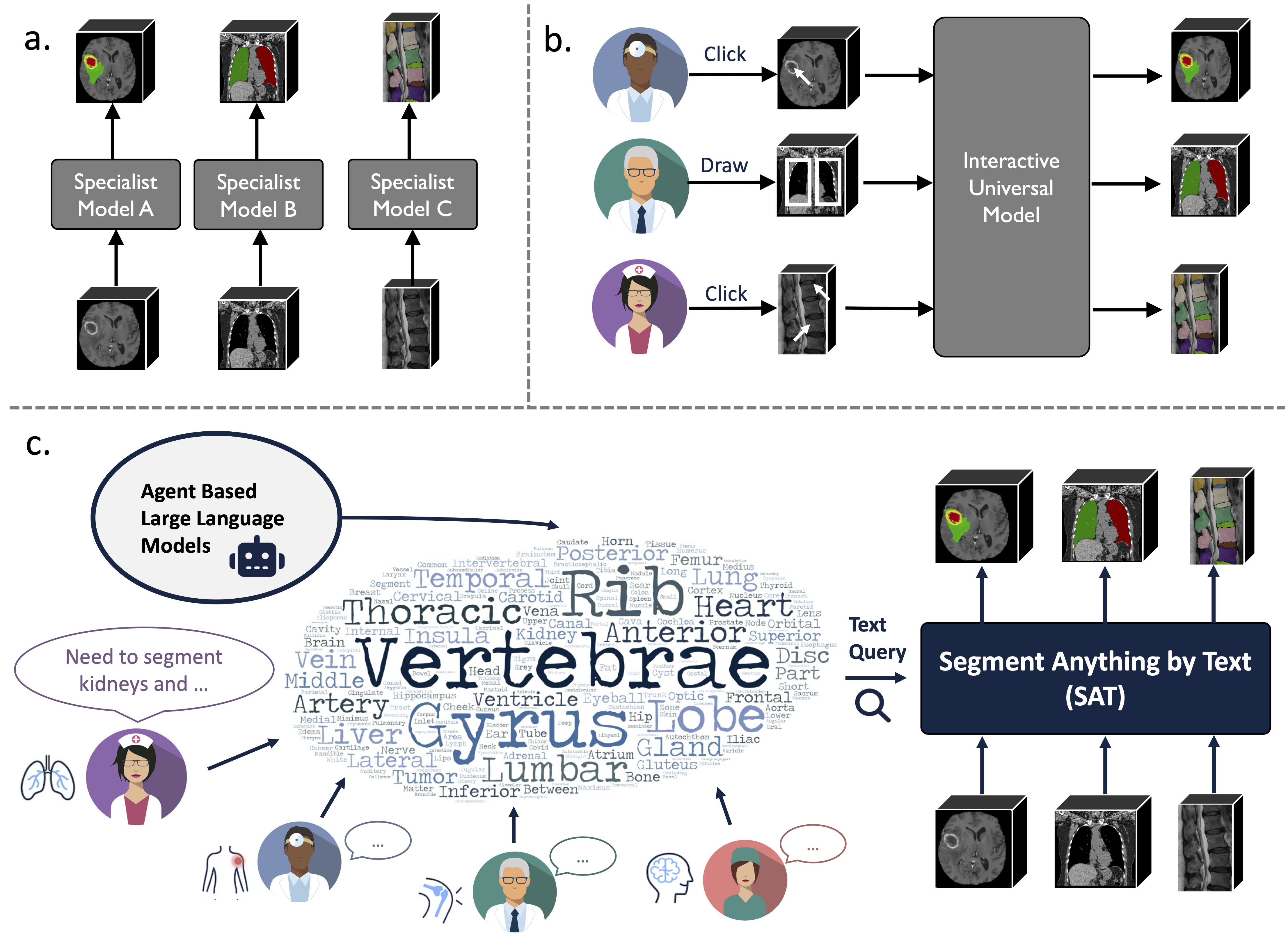
일련의 전문가 모델을 교육하고 배포하는 것보다 강력하고 효율적일 수 있습니다. 자세한 내용은 웹사이트나 자료를 참조하세요.
2024.08 ? SAT 및 대규모 언어 모델을 기반으로 포괄적이고 대규모이며 지역 기반의 3D 흉부 CT 해석 데이터 세트를 구축합니다. 여기에는 196개 카테고리에 대한 기관 수준 세분화와 각 문장이 해당 세분화에 기반을 둔 다중 세분화 보고서가 포함되어 있습니다. 허깅페이스에서 확인해보세요.
2024.06? 우리는 72개의 공개 분할 데이터 세트 모음인 SAT-DS를 구축하기 위한 코드를 출시했습니다. 여기에는 22K 이상의 3D 이미지, 302K 분할 마스크, 3가지 다른 양식(MRI, CT, PET) 및 8가지 인체 영역의 497개 클래스가 포함되어 있습니다. 우리는 SAT를 구축합니다. 또한 42/72 데이터세트에 대한 바로가기 다운로드 링크를 제공합니다. 이 링크는 귀하의 편의를 위해 사전 처리 및 패키징되어 다운로드 및 추출 시 즉시 사용할 수 있습니다. 자세한 내용은 이 저장소를 확인하세요.
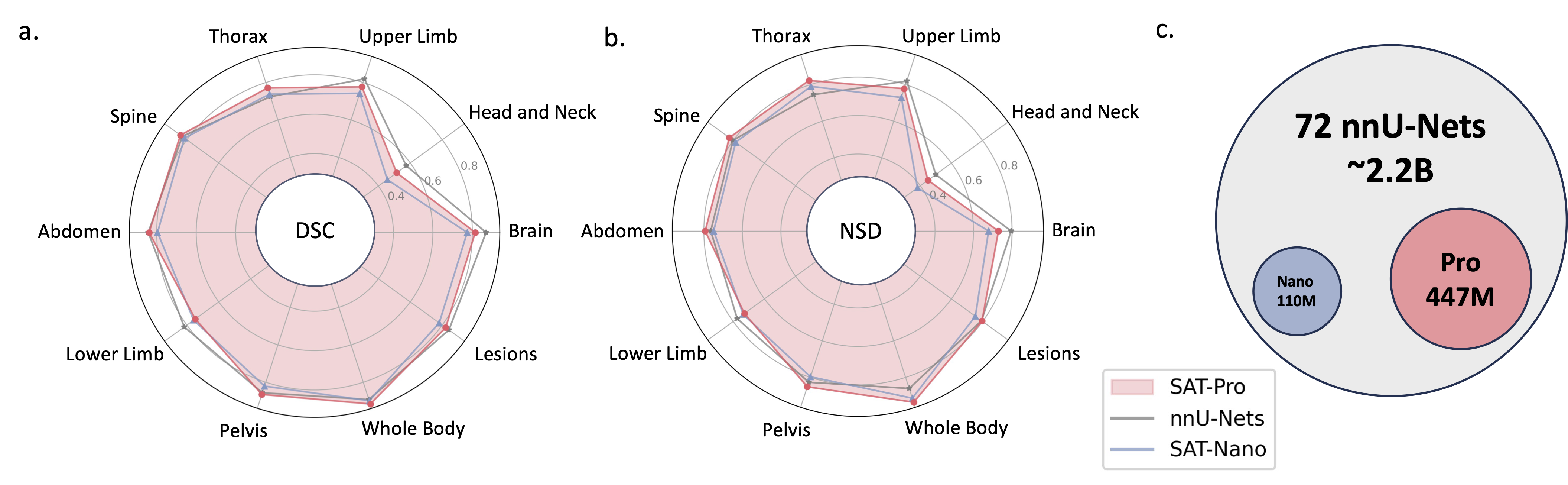
2024.05 ? 더 큰 모델 크기( SAT-Pro )와 더 많은 데이터 세트( 72 )를 갖춘 새 버전의 SAT를 교육하며 현재 497개의 클래스를 지원합니다! 또한 SAT-Nano를 갱신하고 다양한 시각적 백본(U-Mamba 및 SwinUNETR) 및 텍스트 인코더(MedCPT 및 BERT-Base)를 기반으로 SAT-Nano의 일부 변형을 출시합니다. 이 업데이트에 대한 자세한 내용은 새 문서를 참조하세요.
U-Net 구현은 동적 네트워크 아키텍처의 사용자 정의 버전을 사용하여 설치합니다.
cd model
pip install -e dynamic-network-architectures-main
기타 주요 요구사항:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
SAT-Nano의 U-Mamba 변형을 원한다면 mamba_ssm 도 설치해야 합니다.
S1. requirements.txt 에 따라 환경을 구축합니다.
S2. Huggingface에서 SAT 및 Text Encoder의 체크포인트를 다운로드합니다.
S3. jsonl 파일에 데이터를 준비합니다. data/inference_demo/demo.jsonl 에서 데모를 확인하세요.
분할할 각 샘플에는 image (이미지 경로), labe (세그먼트 대상 이름), dataset (샘플이 속한 데이터세트) 및 modality (ct, mri 또는 pet)이 필요합니다. SAT가 지원하는 양식과 클래스는 논문의 표 12에서 확인할 수 있습니다.
orientation_code (orientation)는 기본적으로 RAS 이며 축 평면의 대부분의 이미지에 적합합니다. 시상면의 이미지(예: 척추 검사)의 경우 이를 ASR 로 설정합니다. 입력 이미지는 H,W,D 모양이어야 합니다. 우리의 데이터 처리 코드는 방향, 강도, 간격 등의 측면에서 입력 이미지를 정규화합니다. 성공적으로 처리된 두 개의 이미지는 demoprocessed_data 에서 찾을 수 있습니다. SAT 성능을 보장하려면 정규화가 올바르게 수행되었는지 확인하세요.
S4. SAT-Pro로 추론을 시작하시겠습니까?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d 입력 이미지 패치의 배치 크기이며 GPU 메모리를 기반으로 조정해야 합니다(아래 표 확인). --max_queries GPU 메모리가 매우 제한되지 않는 한 추론 데이터 세트의 클래스보다 크게 설정하는 것이 좋습니다.
| 모델 | 배치 크기_3d | GPU 메모리 |
|---|---|---|
| SAT-프로 | 1 | ~ 34GB |
| SAT-Pro | 2 | ~ 62GB |
| SAT-나노 | 1 | ~ 24GB |
| SAT-나노 | 2 | ~ 36GB |
S5. 출력을 보려면 --rcd_dir 확인하세요. 결과는 데이터 세트별로 구성됩니다. 각 경우에 대해 입력 이미지, 집계된 분할 결과 및 각 클래스의 분할이 포함된 폴더가 검색됩니다. 모든 출력은 nifiti 파일로 저장됩니다. ITK-SNAP을 사용하여 시각화할 수 있습니다.
72개의 데이터 세트에 대해 훈련된 SAT-Nano를 사용하려면 --vision_backbone 'UNET'으로 수정하고 --checkpoint 및 --text_encoder_checkpoint 그에 따라 변경하면 됩니다.
다른 SAT-Nano 변형의 경우(49개 데이터 세트로 훈련됨):
UNET-우리: set --vision_backbone 'UNET' 및 --text_encoder 'ours' ;
UNET-CPT: --vision_backbone 'UNET' 및 --text_encoder 'medcpt' 설정;
UNET-BB: --vision_backbone 'UNET' 및 --text_encoder 'basebert' 설정;
UMamba-CPT: --vision_backbone 'UMamba' 및 --text_encoder 'medcpt' 설정;
SwinUNETR-CPT: --vision_backbone 'SwinUNETR' 및 --text_encoder 'medcpt' 설정;
훈련을 시작하기 전 몇 가지 준비사항:
sh/ 의 slurm 스크립트를 사용하세요. SAT-Pro를 예로 들어보겠습니다. sbatch sh/train_sat_pro.sh
또한 이 리포지토리에 따라 테스트 데이터를 구축해야 합니다. 평가 프로세스를 시작하려면 slurm 스크립트 sh/evaluate_sat_pro.sh 를 참조하세요.
sbatch sh/evaluate_sat_pro.sh
연구나 프로젝트에 이 코드를 사용하는 경우 다음을 인용해 주세요.
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}