STRS(Spatial Total RNA-Sequencing)를 위한 분자 및 계산 워크플로우 제품군
이 저장소에는 거의 모든 샘플에서 STRS를 실행하고 분석하는 데 필요한 모든 프로토콜, 파이프라인 및 스크립트가 있습니다!
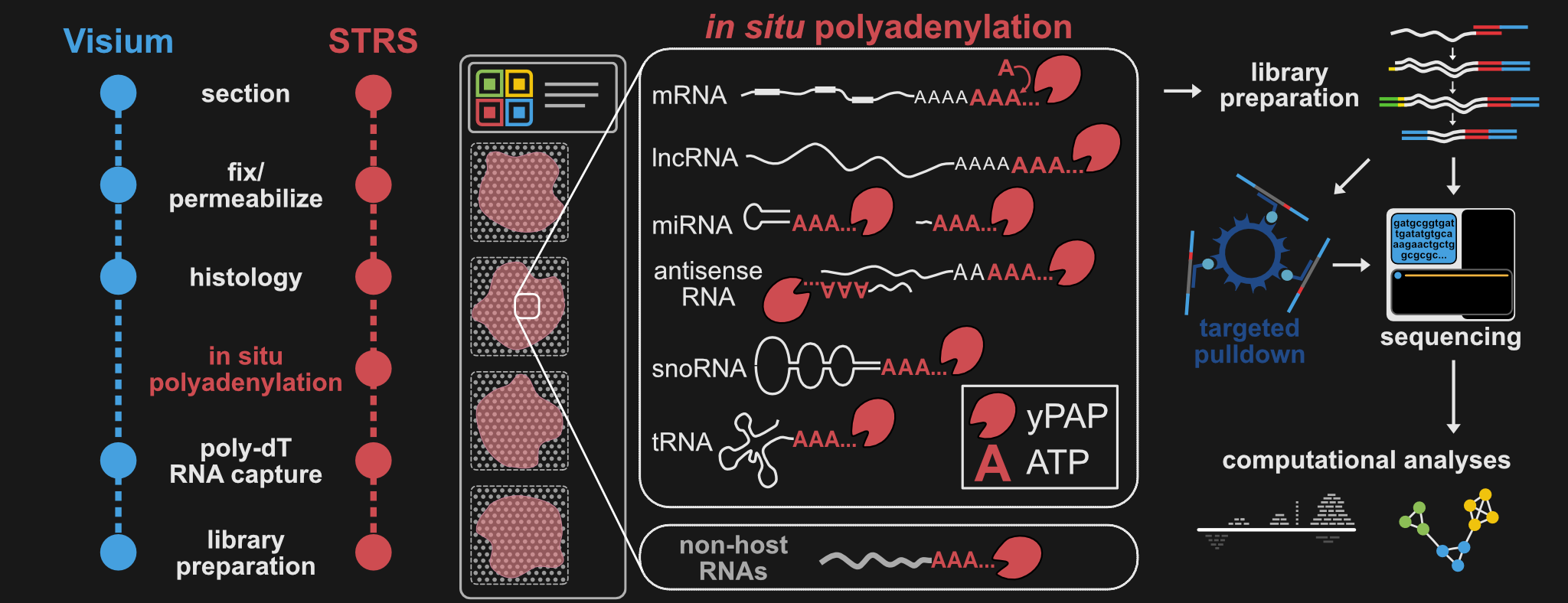
txg_snake 확인하세요!McKellar 외, Nature Biotechnology , 2022
bioRxiv 사전 인쇄 링크
manuscripts : 사전 인쇄본 및 후속 원고의 .pdf 파일(희망합니다!)
protocols : 여기에 사용된 프로토콜에 대한 Microsoft Word 및 .pdf 문서
pipelines : 우리 연구에 사용된 Snakemake 워크플로입니다. 여기에는 STRS 데이터(kallisto, STARsolo 및 miRge3.0), 소형 RNAseq 데이터(STAR 및 miRge3.0), SmartSeqTotal(kallisto) 및 VASAdrop(kalisto)에 대한 정렬 파이프라인이 포함됩니다.
scripts : 우리가 사용한 다른 모든 코드! 대부분 공간 분석에 사용되는 R 스크립트와 유틸리티 함수가 포함되어 있습니다.
references : 이 연구에 사용된 참조 게놈 및 주석에 대한 정보 및 스크립트
resources : 데이터를 분석하는 데 사용된 다양한 메타데이터, 유전자 목록 및 기타 정보 자세한 내용은 각 하위 디렉터리의 README 파일을 참조하세요.
STRS 데이터 세트: GSE200481
SkM/심장 작은 RNAseq 데이터: GSE200480
단일핵 총 RNAseq 데이터(C2C12 세포주): GSE209780
Small RNA Atlas 데이터(Isakova et al, PNAS , 2020): GSE119661
SmartSeqTotal 데이터(Isakova et al, PNAS , 2021): GSE151334
VASAdrop 데이터(Salmen et al, Nature Biotechnology , 2022): GSE176588
이윈 드 블라민크([email protected])
벤자민 D. 코스그로브([email protected])
데이비드 W. 맥켈러([email protected])