
이 저장소는 CELL-E 2: 양방향 텍스트-이미지 변환기를 사용하여 단백질을 그림으로 변환하고 다시 변환하는 공식 구현입니다.
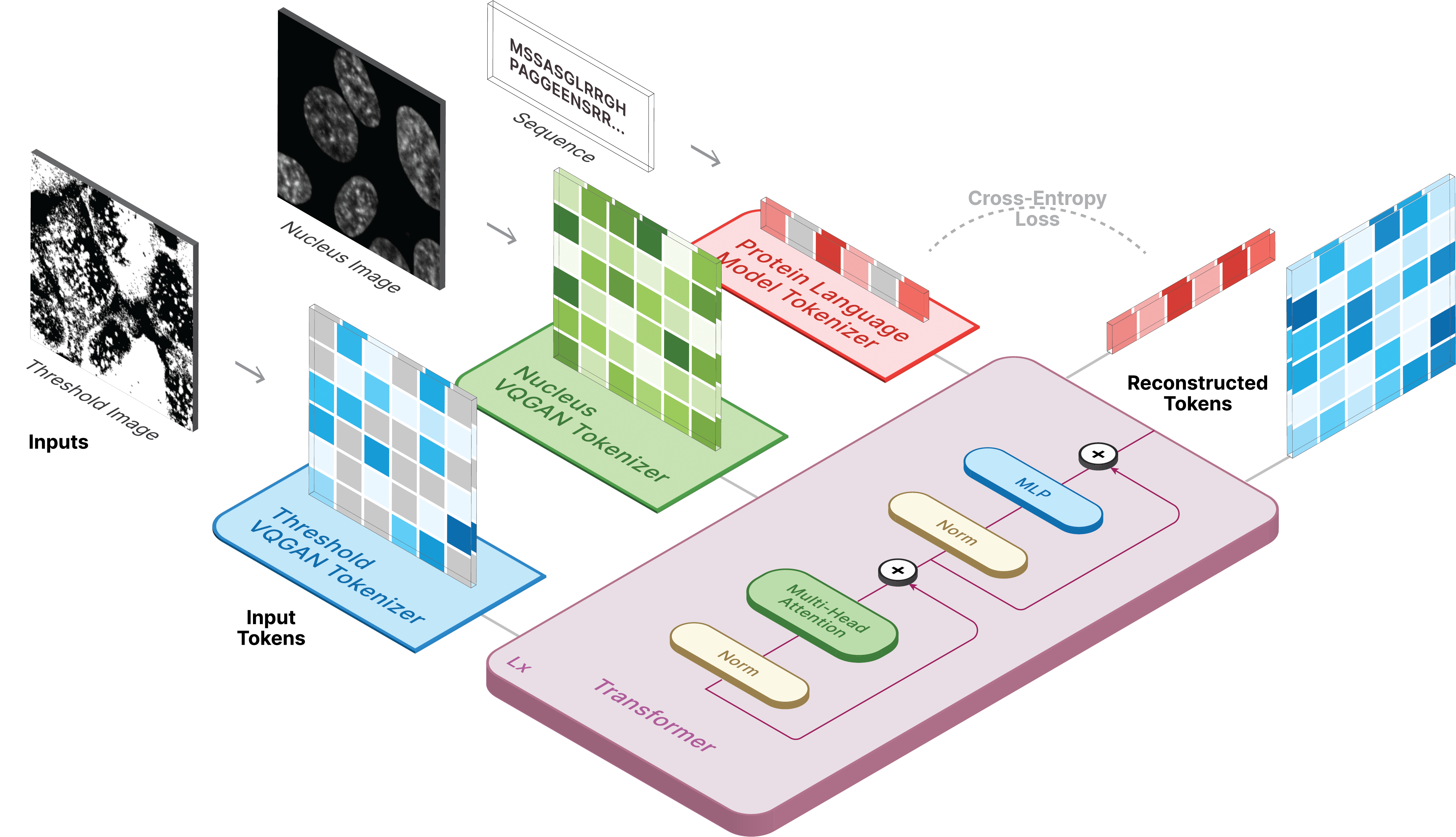
다음을 통해 가상 환경을 만들고 필요한 패키지를 설치합니다.
pip install -r requirements.txt
다음으로, 적절한 CUDA 버전과 함께 torch = 2.0.0 설치합니다.
Hugging Face에서 모델을 만나실 수 있습니다.
또한 자신의 데이터에 대해 예측을 실행할 수 있는 두 개의 공간도 있습니다!
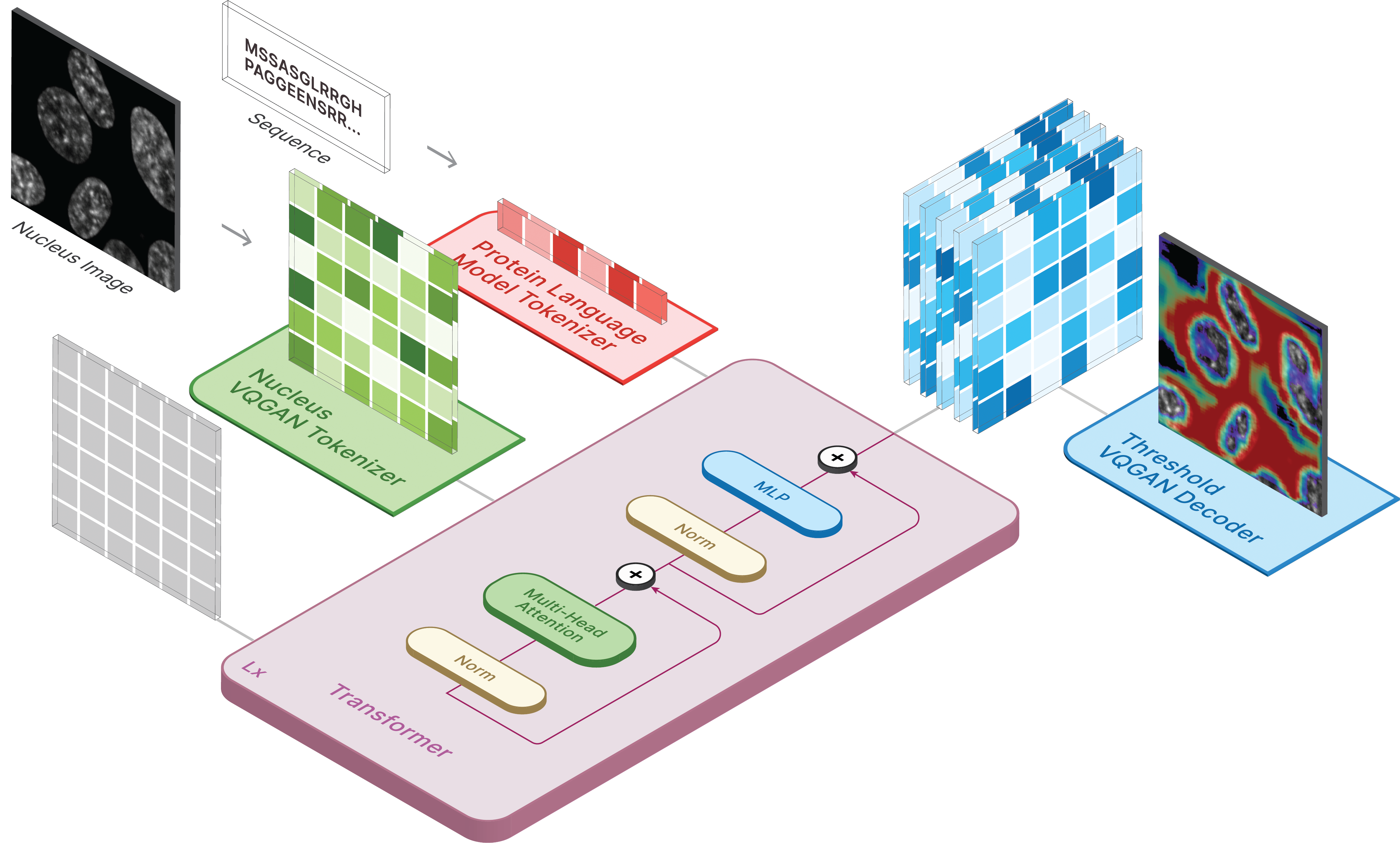
이미지를 생성하려면 저장된 모델을 ckpt_path로 설정하세요. 이 방법은 불안정할 수 있으므로 Demo.ipynb 참조하여 다른 로딩 방법을 확인하세요.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )CELL-E 교육은 3단계로 진행됩니다.
단백질 임계값 이미지를 사용하는 경우 데이터 세트에 대해 threshold: True 설정합니다.
우리는 taming-transformers 코드의 약간 수정된 버전을 사용합니다.
훈련하려면 다음 스크립트를 실행하십시오.
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
--gpus 와 같은 추가 플래그는 원본 저장소를 참조하세요.
scripts 폴더에는 Human Protein Atlas 및 OpenCell 이미지를 다운로드하기 위한 스크립트가 제공됩니다. 데이터로더에는 data_csv 필요합니다. nucleus_image_path , protein_image_path , metadata_path , split (트레인 또는 Val) 및 sequence (선택 사항) 열을 포함하는 csv 파일을 생성해야 합니다. 이 파일은 이미지 및 메타데이터 파일과 동일한 일반 data 폴더 내에 존재한다고 가정합니다.
메타데이터는 모든 단백질 서열과 함께 제공되는 JSON입니다. 시퀀스가 data_csv 에 표시되지 않으면 protein_sequence 라는 키와 함께 metadata.json 에 표시되어야 합니다.
여기에 더 많은 정보를 추가하면 개별 단백질을 쿼리하는 데 유용할 수 있습니다. 데이터세트 객체 내에 self.metadata 변수를 생성하는 retrieve_metadata 통해 검색할 수 있습니다.
훈련하려면 다음 스크립트를 실행하십시오.
python celle_main.py --base configs/celle.yaml -t True
VQGAN과 동일한 형식으로 --gpus 지정합니다.
CELL-E에는 다음과 같은 옵션이 포함되어 있습니다.
ckpt_path : 이전 CELL-E 2 훈련을 재개합니다. state_dict로 저장된 모델vqgan_model_path : 단백질 이미지 인코더를 위해 저장된 단백질 이미지 모델(state_dict 포함)vqgan_config_path : 저장된 단백질 이미지 모델 yamlcondition_model_path : 단백질 이미지 인코더에 대해 저장된 조건(핵) 모델(state_dict 포함)condition_config_path : 저장된 조건(nucleus) 모델 yamlnum_images : 단백질 이미지 인코더만 사용하는 경우 1, 조건 이미지 인코더를 포함하는 경우 2image_key : nucleus , target 또는 thresholddim : 언어 모델 임베딩의 차원num_text_tokens : 언어 모델의 총 토큰 수(ESM-2의 경우 33)text_seq_len : 고려되는 총 아미노산 수depth : 트랜스포머 모델 깊이, 일반적으로 VRAM을 희생하면 깊을수록 좋습니다.heads : 다중 헤드 어텐션에 사용되는 헤드 수dim_head : 주의 머리의 크기attn_dropout : 훈련 중 주의 이탈률ff_dropout : 훈련 중 피드포워드 드롭아웃 비율loss_img_weight : 이미지 재구성에 적용되는 가중치입니다. 텍스트 가중치 = 1loss_text_weight : 조건 이미지 재구성에 적용되는 가중치입니다.stable : Norms 가중치(그라디언트 폭발이 발생할 때)learning_rate : Adam 최적화 프로그램의 학습률monitor : 모델을 저장하는 데 사용되는 매개변수 연구의 어떤 부분에든 우리 코드를 사용하기로 결정했다면 우리를 인용해 주세요.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}