이 패키지는 폐 분할을 위해 훈련된 U-net 모델을 제공합니다. 현재 4가지 모델을 사용할 수 있습니다.
U-net(R231): 이 모델은 광범위한 시각적 변수를 포괄하는 크고 다양한 데이터 세트에서 훈련되었습니다. 이 모델은 개별 조각에 대해 분할을 수행하고 공기 주머니, 종양 및 삼출물을 포함하는 오른쪽-왼쪽 폐를 별도로 추출합니다. 기관은 폐 분할에 포함되지 않습니다. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): 이 모델은 LTRC 데이터세트의 하위 집합에 대해 학습되었습니다. 이 모델은 개별 폐엽의 분할을 수행하지만 치밀한 병리가 있거나 모든 조각에서 균열이 보이지 않는 경우 성능이 제한됩니다.
U-net(LTRCLobes_R231): R231 및 LTRCLobes 모델을 실행하고 결과를 융합합니다. LTRCLobe의 거짓 부정은 R231 예측으로 채워지고 이웃 레이블에 매핑됩니다. LTRCLobe의 거짓양성은 제거됩니다. 융합 프로세스는 계산 집약적이며 데이터 및 결과에 따라 볼륨당 최대 몇 분이 걸릴 수 있습니다.
U-net(R231CovidWeb)
두 가지 모델이 적용된 예 . 왼쪽: U-net(R231)은 왼쪽 폐와 오른쪽 폐를 구별하며 삼출액(세 번째 줄), 종양 또는 심각한 섬유증(네 번째 줄)과 같은 매우 밀집된 영역을 포함합니다. 오른쪽: U-net(LTRLobes)은 폐엽을 구별하지만 밀도가 매우 높은 영역은 포함하지 않습니다. LTRCLobes_R231은 LTRCLobe와 R231 결과를 융합합니다. R231CovidWeb은 추가적인 코로나19 데이터로 훈련되었습니다.
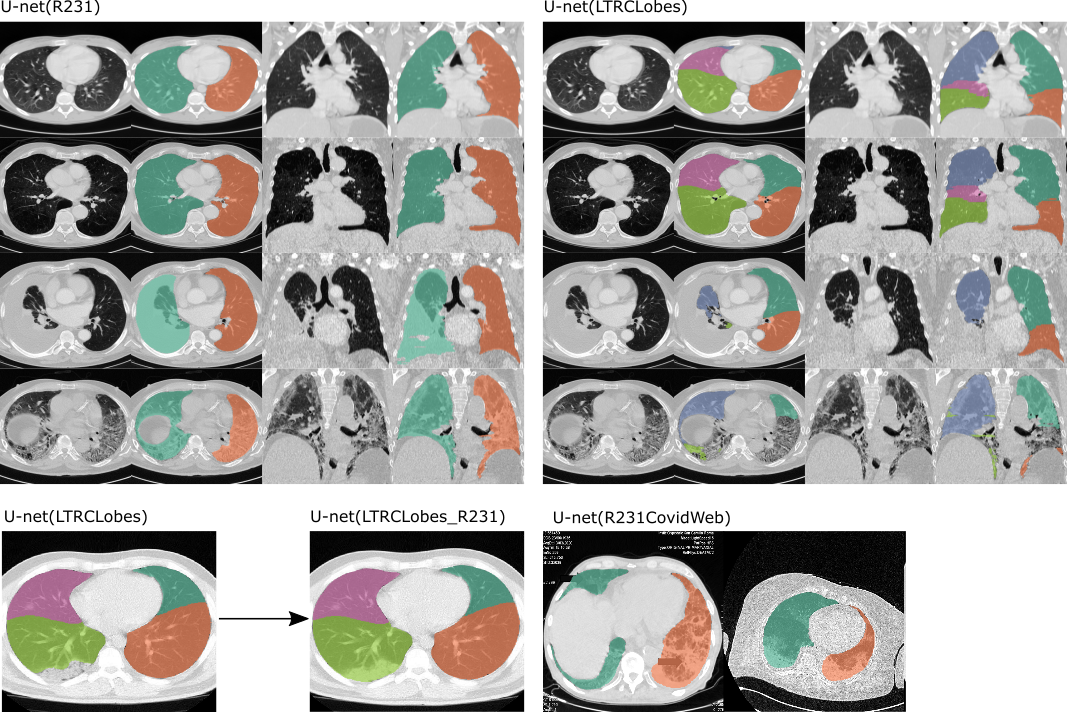
출력의 의미 :
두 가지 라벨 모델(왼쪽-오른쪽):
1 = 오른쪽 폐
2 = 왼쪽 폐
5가지 라벨 모델(폐엽):
1 = 왼쪽 상엽
2 = 왼쪽 하엽
3 = 오른쪽 상엽
4 = 오른쪽 중간엽
5 = 우하엽
폐 CT 데이터에 대한 더 흥미로운 연구를 보려면 당사 연구 그룹 웹사이트(https://www.cir.meduniwien.ac.at/research/lung/)를 확인하세요.
작업에 이 코드나 훈련된 모델 중 하나를 사용하는 경우 다음을 참조하세요.
Hofmanninger, J., 기도, F., Pan, J. 외. 일상적인 영상 촬영에서 자동 폐 분할은 주로 방법론 문제가 아니라 데이터 다양성 문제입니다. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
본 논문에는 사용된 데이터 세트에 대한 자세한 설명, U-net(R231) 모델에 대한 철저한 평가 및 참조 방법과의 비교가 포함되어 있습니다.
pip install lungmask
또는
pip install git+https://github.com/JoHof/lungmask
Windows에서는 설정에 따라 미리 토치를 설치해야 할 수도 있습니다: https://pytorch.org
CPU 전용 추론과 GPU 지원 추론 간의 런타임은 크게 다릅니다. GPU를 사용하면 볼륨을 처리하는 데 몇 초밖에 걸리지 않으며, CPU만 사용하면 몇 분 정도 걸립니다. GPU를 사용하려면 토치 설치에 CUDA가 지원되는지 확인하십시오. cuda 메모리 부족 오류가 발생하는 경우 선택적 인수 --batchsize 1 사용하여 배치 크기를 1로 줄입니다.
lungmask INPUT OUTPUT
INPUT이 파일을 가리키면 파일이 처리됩니다. INPUT이 디렉터리를 가리키는 경우 해당 디렉터리에서 DICOM 시리즈를 검색합니다. 발견된 가장 큰 볼륨(복셀 수 기준)이 폐마스크를 계산하는 데 사용됩니다. OUTPUT은 출력 파일 이름입니다. 모든 ITK 형식이 지원됩니다.
모델을 선택하세요:
기본적으로 U-net(R231)이 사용됩니다. 그러나 LTRCLobes와 같은 대체 모델을 지정할 수 있습니다.
lungmask INPUT OUTPUT --modelname LTRCLobes
추가 옵션을 보려면 다음을 입력하세요.
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image는 SimpleITK 객체여야 합니다.
다음과 같이 대체 모델을 로드합니다.
inferer = LMInferer(modelname="R231CovidWeb")
모델 융합 기능(예: LTRCLobes_R231)을 사용하려면 다음을 사용하세요.
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
버전 0.2.9부터 numpy 배열이 입력 볼륨으로 지원됩니다. 이 모드에서는 입력 numpy 배열이 각 축에 대해 다음 형식을 갖는다고 가정합니다.
모델은 전체 조각에서만 작동합니다. 처리할 슬라이스는 전체 폐를 보여야 하며 분할되기 위해서는 폐가 조직으로 둘러싸여 있어야 합니다. 그러나 폐가 조직으로 둘러싸여 있는 한 이 모델은 시야가 잘린 경우에 매우 안정적입니다.
lungmask INPUT OUTPUT --modelname R231CovidWeb
일반 U-net(R231) 모델은 코로나19 CT 스캔에 매우 효과적입니다. 그러나 웹의 슬라이스 모음 및 사례 보고서는 원래의 하운스필드 단위(HU) 값만 추정할 수 있도록 일반 이미지 형식으로 잘리거나 주석이 추가되거나 인코딩되는 경우가 많습니다. U-net(R231CovidWeb) 모델의 훈련 데이터는 일반 영상 형식에서 HU로 다시 매핑된 코로나19 슬라이스로 보강되었습니다. 데이터는 MedSeg(http://medicalsegmentation.com/covid19/)에 의해 수집되고 준비되었습니다. 일반 U-net(R231)은 이러한 이미지에 대해 매우 좋은 결과를 보여 주었지만 이 모델이 약간 향상된 분할을 제공하는 경우가 있을 수 있습니다. 웹의 이미지를 사용할 때는 이미지를 HU에 다시 매핑해야 합니다. 이 블로그 게시물에서는 이를 수행하는 방법을 설명합니다. 또는 --noHU 태그를 설정할 수 있습니다. 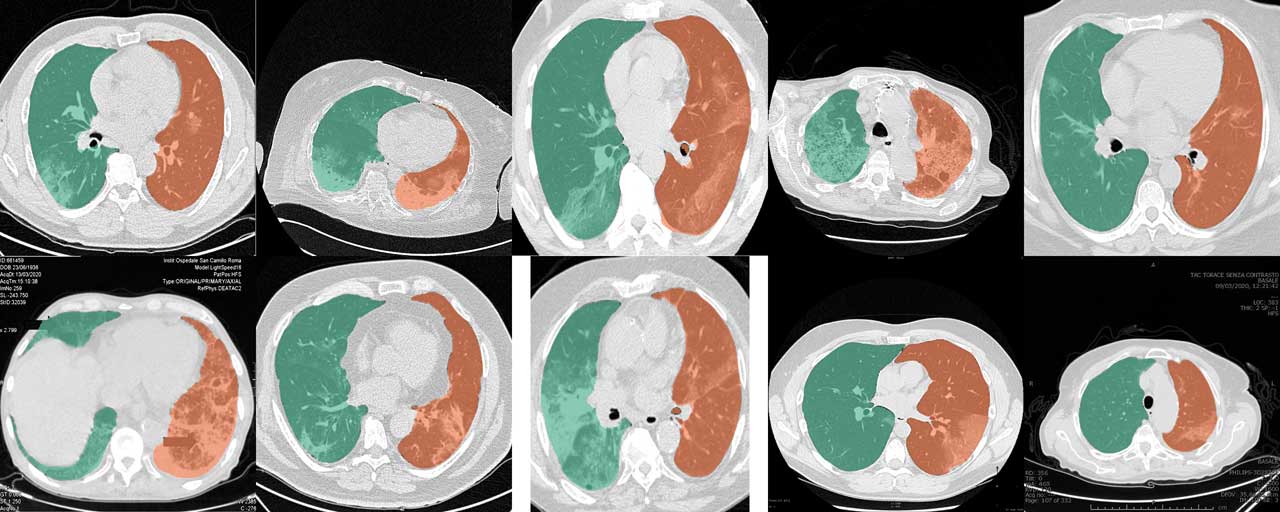
이 기능은 0.2.5와 0.2.14 사이의 버전에서만 사용할 수 있습니다. 버전 0.2.5부터 이러한 이미지가 지원됩니다. HU로 인코딩되지 않은 이미지를 처리하는 경우 --noHU 태그를 사용하세요. 모델은 HU로 인코딩된 적절한 CT 스캔에 대해 훈련되었다는 점을 명심하세요. 자르고, 주석을 달고, 매우 높고 매우 낮은 강도로 이동된 이미지에 대한 결과는 그다지 신뢰할 수 없을 수 있습니다. --noHU 태그를 사용하면 단일 슬라이스만 처리할 수 있습니다.