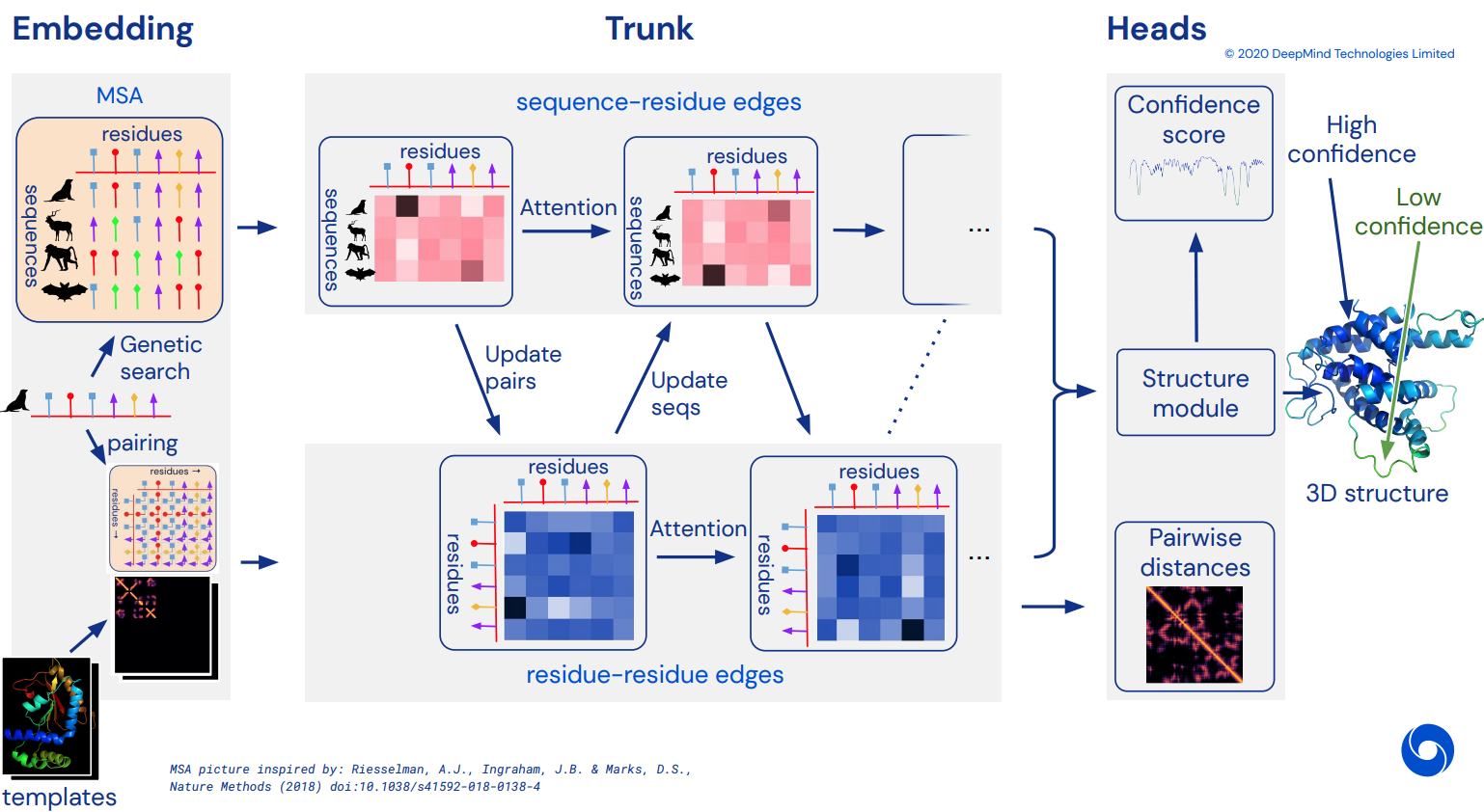
결국 CASP14를 해결한 놀라운 관심 네트워크인 Alphafold2의 비공식 작동 Pytorch 구현이 됩니다. 아키텍처에 대한 자세한 내용이 공개되면 점차적으로 구현될 예정입니다.
이것이 복제되면 사용 가능한 모든 아미노산 서열을 인실리코로 접어 학문적 급류로 공개하여 과학을 발전시킬 계획입니다. 복제 노력에 관심이 있으시면 이 Discord 채널에 #alphafold를 들러주세요.
업데이트: Deepmind는 가중치와 함께 Jax의 공식 코드를 오픈 소스로 공개했습니다! 이 저장소는 이제 위치 인코딩이 일부 개선되어 직선 파이토치 변환에 맞춰져 있습니다.
ArxivInsights 비디오
$ pip install alphafold2-pytorchlhatsk는 trRosetta와 동일한 설정을 사용하여 이 저장소의 수정된 트렁크를 교육하고 경쟁력 있는 결과를 보고했습니다.
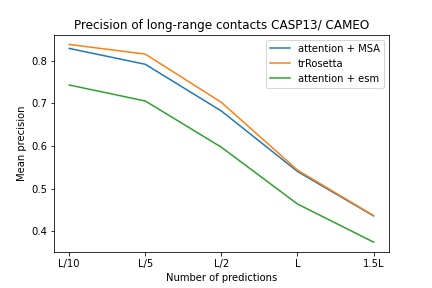
blue used the the trRosetta input (MSA -> potts -> axial attention), green used the ESM embedding (only sequence) -> tiling -> axial attention - lhatsk
Alphafold-1과 유사하지만 주의를 기울여 디스토그램을 예측합니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
reversible = False # set this to True for fully reversible self / cross attention for the trunk
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda () # AA length of 128
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda () # MSA doesn't have to be the same length as primary sequence
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) 또한 초기화 시 predict_angles = True 전달하여 각도에 대한 예측을 켤 수도 있습니다. 아래 예는 trRosetta와 동일하지만 self/cross attention을 사용합니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_angles = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram , theta , phi , omega = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
)
# distogram - (1, 128, 128, 37),
# theta - (1, 128, 128, 25),
# phi - (1, 128, 128, 13),
# omega - (1, 128, 128, 25) Fabian의 최근 논문에서는 가중치를 공유하여 좌표를 SE3 Transformer에 반복적으로 다시 공급하는 것이 효과가 있을 수 있다고 제안합니다. 나는 이 아이디어를 바탕으로 실행하기로 결정했습니다. 비록 그것이 실제로 어떻게 작동할지는 아직 미정이지만요.
구조적 개선을 위해 E(n)-Transformer 또는 EGNN을 사용할 수도 있습니다.
업데이트: Baker의 연구실에서는 시퀀스 및 MSA 임베딩부터 SE3 Transformer까지의 엔드투엔드 아키텍처가 trRosetta에 가장 적합하고 Alphafold2와의 격차를 줄일 수 있음을 보여주었습니다. 우리는 등변 네트워크로 전송될 초기 좌표 세트를 생성하기 위해 트렁크 임베딩에 작용하는 그래프 변환기를 사용할 것입니다. (이는 Baker 연구실 이전의 논문에서 MSA Transformer 임베딩으로부터 3D 좌표를 알아내는 작업에서 Costa 등이 추가로 확증했습니다.)
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
structure_module_type = 'se3' , # use SE3 Transformer - if set to False, will use E(n)-Transformer, Victor and Max Welling's new paper
structure_module_dim = 4 , # se3 transformer dimension
structure_module_depth = 1 , # depth
structure_module_heads = 1 , # heads
structure_module_dim_head = 16 , # dimension of heads
structure_module_refinement_iters = 2 , # number of equivariant coordinate refinement iterations
structure_num_global_nodes = 1 # number of global nodes for the structure module, only works with SE3 transformer
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue 기본 가정은 트렁크가 잔여물 수준에서 작동한 다음 SE3 Transformers, E(n)-Transformer 또는 EGNN이 개선을 수행하는지 여부에 관계없이 구조 모듈에 대한 원자 수준으로 구성된다는 것입니다. 이 라이브러리는 기본적으로 3개의 백본 원자(C, Ca, N)로 설정되어 있지만 Cb 및 사이드체인을 포함하여 원하는 다른 원자를 포함하도록 구성할 수 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
atoms = 'backbone-with-cbeta'
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 4, 3) <-- 4 atoms per residue (C, Ca, N, Cb) atoms 에 대한 유효한 선택은 다음과 같습니다.
backbone - 3개의 백본 원자(C, Ca, N) [기본값]backbone-with-cbeta - 3개의 백본 원자와 C 베타backbone-with-oxygen - 카르복실기의 백본 원자 3개와 산소backbone-with-cbeta-and-oxygen - C 베타와 산소가 있는 3개의 백본 원자all - 사이드체인의 백본 및 기타 모든 원자또한 포함하려는 원자를 정의하는 모양(14)의 텐서를 전달할 수도 있습니다.
전.
atoms = torch . tensor ([ 1 , 1 , 1 , 1 , 1 , 1 , 0 , 1 , 0 , 0 , 0 , 0 , 0 , 1 ])이 저장소는 Facebook AI의 사전 훈련된 임베딩을 통해 네트워크를 쉽게 보완할 수 있는 기능을 제공합니다. 여기에는 사전 훈련된 ESM, MSA 변환기 또는 단백질 변환기에 대한 래퍼가 포함되어 있습니다.
몇 가지 전제 조건이 있습니다. 사전 학습된 변환기는 일부 융합 작업을 사용하므로 Nvidia의 apex 라이브러리가 설치되어 있는지 확인해야 합니다.
또는 아래 스크립트를 실행해 볼 수도 있습니다.
git clone https://github.com/NVIDIA/apex
cd apex
pip install -v --disable-pip-version-check --no-cache-dir --global-option= " --cpp_ext " --global-option= " --cuda_ext " ./ 다음으로 Alphafold2 인스턴스를 가져와서 ESMEmbedWrapper , MSAEmbedWrapper 또는 ProtTranEmbedWrapper 로 래핑하기만 하면 시퀀스와 다중 시퀀스 정렬을 모두 포함하고 프로젝트에 지정된 치수로 투영하는 작업을 처리합니다. 모델). 래퍼 추가 외에는 아무것도 변경할 필요가 없습니다.
import torch
from alphafold2_pytorch import Alphafold2
from alphafold2_pytorch . embeds import MSAEmbedWrapper
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64
)
model = MSAEmbedWrapper (
alphafold2 = alphafold2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) 기본적으로 래퍼가 트렁크에 시퀀스 및 MSA 임베딩을 제공하더라도 일반적인 토큰 임베딩으로 합산됩니다. 토큰 임베딩 없이 Alphafold2를 학습시키려면(사전 학습된 임베딩에만 의존) Alphafold2 초기화에서 disable_token_embed True 로 설정해야 합니다.
alphafold2 = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
disable_token_embed = True
) Jinbo Xu의 논문에서는 거리를 구간화할 필요가 없으며 대신 평균과 표준 편차를 직접 예측할 수 있다고 제안합니다. predict_real_value_distances 거리 플래그 하나를 켜서 이를 사용할 수 있습니다. 이 경우 반환된 거리 예측은 평균 및 표준 편차에 대해 각각 2 차원을 갖게 됩니다.
predict_coords 도 켜져 있으면 MDS는 디스토그램 저장소에서 계산할 필요 없이 평균 및 표준 편차 예측을 직접 받아들입니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
predict_coords = True ,
predict_real_value_distances = True , # set this to True
structure_module_type = 'se3' ,
structure_module_dim = 4 ,
structure_module_depth = 1 ,
structure_module_heads = 1 ,
structure_module_dim_head = 16 ,
structure_module_refinement_iters = 2
). cuda ()
seq = torch . randint ( 0 , 21 , ( 2 , 64 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 2 , 5 , 60 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
coords = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (2, 64 * 3, 3) <-- 3 atoms per residue 하나의 추가 키워드 인수 use_conv = True 설정하여 기본 시퀀스와 MSA 모두에 대해 컨벌루션 블록을 추가할 수 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True # set this to True
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37)컨벌루션 커널은 이 논문의 지침을 따르며 1d 및 2d 커널을 하나의 resnet 유사 블록에 결합합니다. 커널을 완전히 사용자 정의할 수 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
conv_seq_kernels = (( 9 , 1 ), ( 1 , 9 ), ( 3 , 3 )), # kernels for N x N primary sequence
conv_msa_kernels = (( 1 , 9 ), ( 3 , 3 )), # kernels for {num MSAs} x N MSAs
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) 하나의 추가 키워드 인수를 사용하여 순환 확장을 수행할 수도 있습니다. 모든 레이어의 기본 팽창은 1 입니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
use_conv = True , # set this to True
dilations = ( 1 , 3 , 5 ) # cycle between dilations of 1, 3, 5
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) 마지막으로 컨볼루션, 셀프 어텐션, 심도별 크로스 어텐션 반복 패턴을 따르는 대신 custom_block_types 키워드를 사용하여 원하는 순서를 맞춤 설정할 수 있습니다.
전. 주로 컨볼루션을 먼저 수행하고 그다음에 self-attention + cross-attention 블록을 수행하는 네트워크
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
heads = 8 ,
dim_head = 64 ,
custom_block_types = (
* (( 'conv' ,) * 6 ),
* (( 'self' , 'cross' ) * 6 )
)
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 128 )). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 5 , 120 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask
) # (1, 128, 128, 37) Microsoft Deepspeed의 Sparse Attention으로 훈련할 수 있지만 설치 과정을 견뎌야 합니다. 2단계입니다.
먼저 Sparse Attention이 포함된 Deepspeed를 설치해야 합니다.
$ sh install_deepspeed.sh 다음으로 pip 패키지 triton 설치해야 합니다.
$ pip install triton위의 두 가지 모두 성공했다면 이제 Sparse Attention으로 훈련할 수 있습니다!
안타깝게도 희소 주의는 자기 주의에만 지원되며 교차 주의에는 지원되지 않습니다. 저는 교차 주의를 효과적으로 수행하기 위한 다른 솔루션을 가져올 것입니다.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
max_seq_len = 2048 , # the maximum sequence length, this is required for sparse attention. the input cannot exceed what is set here
sparse_self_attn = ( True , False ) * 6 # interleave sparse and full attention for all 12 layers
). cuda ()또한 교차 참석의 부담을 줄이기 위해 최고의 선형 주의 변형 중 하나를 추가했습니다. 나는 개인적으로 Performer가 그렇게 잘 작동하는 것을 찾지 못했지만 논문에서 단백질 벤치마크에 대한 괜찮은 수치를 보고했기 때문에 이를 포함하고 다른 사람들이 실험할 수 있도록 허용해야 한다고 생각했습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = True # simply set this to True to use Performer for all cross attention
). cuda ()깊이와 동일한 길이의 튜플을 전달하여 선형 주의를 사용하려는 정확한 레이어를 지정할 수도 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_linear = ( True , False ) * 3 # interleave linear and full attention
). cuda ()이 문서에서는 축(예: 이미지)을 정의한 쿼리나 컨텍스트가 있는 경우 해당 축(높이 및 너비)에 걸쳐 평균을 계산하고 평균된 축을 하나의 시퀀스로 연결하여 필요한 주의를 줄일 수 있다고 제안합니다. 특히 기본 시퀀스에 대해 교차 주의를 위한 메모리 절약 기술로 이 기능을 켤 수 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 6 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_kron_primary = True # make sure primary sequence undergoes the kronecker operator during cross attention
). cuda () MSA가 정렬되어 있고 너비가 동일한 경우 cross_attn_kron_msa 플래그를 사용하여 교차 주의 중에 MSA에 동일한 연산자를 적용할 수도 있습니다.
토도
교차 주의를 위해 메모리를 절약하려면 이 백서에 설명된 구성표에 따라 키/값에 대한 압축 비율을 설정할 수 있습니다. 일반적으로 2-4의 압축 비율이 허용됩니다.
model = Alphafold2 (
dim = 256 ,
depth = 12 ,
heads = 8 ,
dim_head = 64 ,
cross_attn_compress_ratio = 3
). cuda ()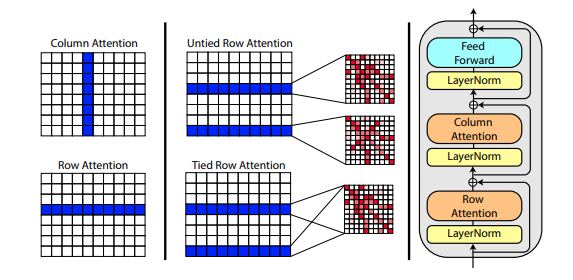
Roshan Rao의 새 논문에서는 MSA 사전 교육에 축 주의를 사용할 것을 제안합니다. 강력한 결과를 바탕으로 이 저장소는 특히 MSA self-attention을 위해 트렁크에서 동일한 체계를 사용합니다.
Alphafold2 초기화 시 msa_tie_row_attn = True 설정을 사용하여 MSA의 행 주의를 연결할 수도 있습니다. 그러나 이를 사용하려면 기본 시퀀스당 MSA 수가 홀수인 경우 사용하지 않는 행에 대해 MSA 마스크가 False 로 올바르게 설정되어 있는지 확인해야 합니다.
model = Alphafold2 (
dim = 256 ,
depth = 2 ,
heads = 8 ,
dim_head = 64 ,
msa_tie_row_attn = True # just set this to true
)템플릿 처리는 또한 템플릿 차원의 수를 따라 수행되는 교차 주의와 함께 주로 축 주의로 수행됩니다. 이는 여기에 표시된 비디오 분류에 대한 최근의 all-attention 접근 방식과 동일한 방식을 크게 따릅니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randint ( 0 , 37 , ( 1 , 2 , 16 , 3 )). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_coors = templates_coors ,
templates_mask = templates_mask
)사이드체인 정보도 각 잔기의 C와 C-알파 좌표 사이의 단위 벡터 형태로 존재하는 경우 다음과 같이 전달할 수도 있습니다.
import torch
from alphafold2_pytorch import Alphafold2
model = Alphafold2 (
dim = 256 ,
depth = 5 ,
heads = 8 ,
dim_head = 64 ,
reversible = True ,
sparse_self_attn = False ,
max_seq_len = 256 ,
cross_attn_compress_ratio = 3
). cuda ()
seq = torch . randint ( 0 , 21 , ( 1 , 16 )). cuda ()
mask = torch . ones_like ( seq ). bool (). cuda ()
msa = torch . randint ( 0 , 21 , ( 1 , 10 , 16 )). cuda ()
msa_mask = torch . ones_like ( msa ). bool (). cuda ()
templates_seq = torch . randint ( 0 , 21 , ( 1 , 2 , 16 )). cuda ()
templates_coors = torch . randn ( 1 , 2 , 16 , 3 ). cuda ()
templates_mask = torch . ones_like ( templates_seq ). bool (). cuda ()
templates_sidechains = torch . randn ( 1 , 2 , 16 , 3 ). cuda () # unit vectors of difference of C and C-alpha coordinates
distogram = model (
seq ,
msa ,
mask = mask ,
msa_mask = msa_mask ,
templates_seq = templates_seq ,
templates_mask = templates_mask ,
templates_coors = templates_coors ,
templates_sidechains = templates_sidechains
)나는 추측성 블로그 게시물에서 Fabian Fuchs가 설명한 대로 SE3 Transformer의 재구현을 준비했습니다.
또한 Victor와 Welling의 새 논문에서는 E(n) 등분산에 대한 불변 기능을 사용하여 SOTA에 도달하고 여러 벤치마크에서 SE3 Transformer보다 성능이 뛰어나면서도 훨씬 빠릅니다. 나는 이 백서의 주요 아이디어를 취하여 이를 변환기로 수정했습니다(기능과 조정 업데이트 모두에 주의를 기울였습니다).
위의 세 가지 등변 네트워크가 모두 통합되었으며, 하나의 하이퍼 매개변수인 structure_module_type 설정하기만 하면 원자 좌표 개선을 위해 저장소에서 사용할 수 있습니다.
se3 SE3 트랜스포머
egnn EGNN
en E(n)-변압기
독자들의 관심을 끄는 점은 세 가지 프레임워크 각각이 관련 문제에 대해 연구자들에 의해 검증되었다는 것입니다.
$ python setup.py test 이 라이브러리는 이 저장소에 있는 Jonathan King의 멋진 작업을 사용할 것입니다. 고마워요 조나단!
또한 The-Eye 프로젝트를 소유한 Archivist가 다운로드하고 호스팅하는 약 3.5TB 상당의 MSA 데이터도 있습니다. (Eleuther AI용 데이터와 모델도 호스팅합니다.) 도움이 되셨다면 기부를 고려해 보세요.
$ curl -s https://the-eye.eu/eleuther_staging/globus_stuffs/tree.txthttps://xukui.cn/alphafold2.html
https://moalquraishi.wordpress.com/2020/12/08/alphafold2-casp14-it-feels-like-ones-child-has-left-home/

https://www.biorxiv.org/content/10.1101/2020.12.10.419994v1.full.pdf
https://pubmed.ncbi.nlm.nih.gov/33637700/
Tencent AI 연구소의 tFold 프레젠테이션
cd downloads_folder > pip install pyrosetta_wheel_filename.whlOpenMM 앰버
@misc { unpublished2021alphafold2 ,
title = { Alphafold2 } ,
author = { John Jumper } ,
year = { 2020 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @article { Rao2021.02.12.430858 ,
author = { Rao, Roshan and Liu, Jason and Verkuil, Robert and Meier, Joshua and Canny, John F. and Abbeel, Pieter and Sercu, Tom and Rives, Alexander } ,
title = { MSA Transformer } ,
year = { 2021 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/02/13/2021.02.12.430858 } ,
journal = { bioRxiv }
} @article { Rives622803 ,
author = { Rives, Alexander and Goyal, Siddharth and Meier, Joshua and Guo, Demi and Ott, Myle and Zitnick, C. Lawrence and Ma, Jerry and Fergus, Rob } ,
title = { Biological Structure and Function Emerge from Scaling Unsupervised Learning to 250 Million Protein Sequences } ,
year = { 2019 } ,
doi = { 10.1101/622803 } ,
publisher = { Cold Spring Harbor Laboratory } ,
journal = { bioRxiv }
} @article { Elnaggar2020.07.12.199554 ,
author = { Elnaggar, Ahmed and Heinzinger, Michael and Dallago, Christian and Rehawi, Ghalia and Wang, Yu and Jones, Llion and Gibbs, Tom and Feher, Tamas and Angerer, Christoph and Steinegger, Martin and BHOWMIK, DEBSINDHU and Rost, Burkhard } ,
title = { ProtTrans: Towards Cracking the Language of Life{textquoteright}s Code Through Self-Supervised Deep Learning and High Performance Computing } ,
elocation-id = { 2020.07.12.199554 } ,
year = { 2021 } ,
doi = { 10.1101/2020.07.12.199554 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/04/2020.07.12.199554.full.pdf } ,
journal = { bioRxiv }
} @misc { king2020sidechainnet ,
title = { SidechainNet: An All-Atom Protein Structure Dataset for Machine Learning } ,
author = { Jonathan E. King and David Ryan Koes } ,
year = { 2020 } ,
eprint = { 2010.08162 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { alquraishi2019proteinnet ,
title = { ProteinNet: a standardized data set for machine learning of protein structure } ,
author = { Mohammed AlQuraishi } ,
year = { 2019 } ,
eprint = { 1902.00249 } ,
archivePrefix = { arXiv } ,
primaryClass = { q-bio.BM }
} @misc { gomez2017reversible ,
title = { The Reversible Residual Network: Backpropagation Without Storing Activations } ,
author = { Aidan N. Gomez and Mengye Ren and Raquel Urtasun and Roger B. Grosse } ,
year = { 2017 } ,
eprint = { 1707.04585 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CV }
} @misc { fuchs2021iterative ,
title = { Iterative SE(3)-Transformers } ,
author = { Fabian B. Fuchs and Edward Wagstaff and Justas Dauparas and Ingmar Posner } ,
year = { 2021 } ,
eprint = { 2102.13419 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { satorras2021en ,
title = { E(n) Equivariant Graph Neural Networks } ,
author = { Victor Garcia Satorras and Emiel Hoogeboom and Max Welling } ,
year = { 2021 } ,
eprint = { 2102.09844 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.LG }
} @misc { su2021roformer ,
title = { RoFormer: Enhanced Transformer with Rotary Position Embedding } ,
author = { Jianlin Su and Yu Lu and Shengfeng Pan and Bo Wen and Yunfeng Liu } ,
year = { 2021 } ,
eprint = { 2104.09864 } ,
archivePrefix = { arXiv } ,
primaryClass = { cs.CL }
} @article { Gao_2020 ,
title = { Kronecker Attention Networks } ,
ISBN = { 9781450379984 } ,
url = { http://dx.doi.org/10.1145/3394486.3403065 } ,
DOI = { 10.1145/3394486.3403065 } ,
journal = { Proceedings of the 26th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining } ,
publisher = { ACM } ,
author = { Gao, Hongyang and Wang, Zhengyang and Ji, Shuiwang } ,
year = { 2020 } ,
month = { Jul }
} @article { Si2021.05.10.443415 ,
author = { Si, Yunda and Yan, Chengfei } ,
title = { Improved protein contact prediction using dimensional hybrid residual networks and singularity enhanced loss function } ,
elocation-id = { 2021.05.10.443415 } ,
year = { 2021 } ,
doi = { 10.1101/2021.05.10.443415 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415 } ,
eprint = { https://www.biorxiv.org/content/early/2021/05/11/2021.05.10.443415.full.pdf } ,
journal = { bioRxiv }
} @article { Costa2021.06.02.446809 ,
author = { Costa, Allan and Ponnapati, Manvitha and Jacobson, Joseph M. and Chatterjee, Pranam } ,
title = { Distillation of MSA Embeddings to Folded Protein Structures with Graph Transformers } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.02.446809 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/02/2021.06.02.446809.full.pdf } ,
journal = { bioRxiv }
} @article { Baek2021.06.14.448402 ,
author = { Baek, Minkyung and DiMaio, Frank and Anishchenko, Ivan and Dauparas, Justas and Ovchinnikov, Sergey and Lee, Gyu Rie and Wang, Jue and Cong, Qian and Kinch, Lisa N. and Schaeffer, R. Dustin and Mill{'a}n, Claudia and Park, Hahnbeom and Adams, Carson and Glassman, Caleb R. and DeGiovanni, Andy and Pereira, Jose H. and Rodrigues, Andria V. and van Dijk, Alberdina A. and Ebrecht, Ana C. and Opperman, Diederik J. and Sagmeister, Theo and Buhlheller, Christoph and Pavkov-Keller, Tea and Rathinaswamy, Manoj K and Dalwadi, Udit and Yip, Calvin K and Burke, John E and Garcia, K. Christopher and Grishin, Nick V. and Adams, Paul D. and Read, Randy J. and Baker, David } ,
title = { Accurate prediction of protein structures and interactions using a 3-track network } ,
year = { 2021 } ,
doi = { 10.1101/2021.06.14.448402 } ,
publisher = { Cold Spring Harbor Laboratory } ,
URL = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402 } ,
eprint = { https://www.biorxiv.org/content/early/2021/06/15/2021.06.14.448402.full.pdf } ,
journal = { bioRxiv }
}