MedSegDiff는 의료 영상 분할을 위한 DPM(확산 확률 모델) 기반 프레임워크입니다. 알고리즘은 우리 논문 MedSegDiff: 확산 확률 모델을 사용한 의료 영상 분할 및 MedSegDiff-V2: Transformer를 사용한 확산 기반 의료 영상 분할에 자세히 설명되어 있습니다.
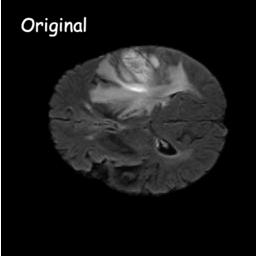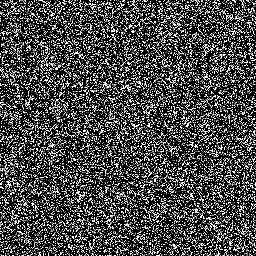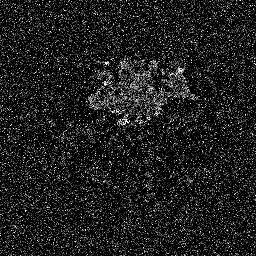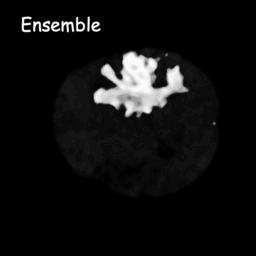 확산 모델은 가우스 잡음을 연속적으로 추가하여 훈련 데이터를 파괴한 다음 이 잡음 과정을 역전시켜 데이터를 복구하는 방법을 학습하는 방식으로 작동합니다. 훈련 후에는 확산 모델을 사용하여 학습된 노이즈 제거 프로세스를 통해 무작위로 샘플링된 노이즈를 전달함으로써 데이터를 생성할 수 있습니다. 이 프로젝트에서는 이 아이디어를 의료 영상 분할로 확장합니다. 원본 이미지를 조건으로 활용하고 무작위 노이즈로부터 여러 개의 분할 맵을 생성한 다음 이를 앙상블링하여 최종 결과를 얻습니다. 이 접근 방식은 의료 이미지의 불확실성을 포착하고 여러 벤치마크에서 이전 방법보다 성능이 뛰어납니다.
확산 모델은 가우스 잡음을 연속적으로 추가하여 훈련 데이터를 파괴한 다음 이 잡음 과정을 역전시켜 데이터를 복구하는 방법을 학습하는 방식으로 작동합니다. 훈련 후에는 확산 모델을 사용하여 학습된 노이즈 제거 프로세스를 통해 무작위로 샘플링된 노이즈를 전달함으로써 데이터를 생성할 수 있습니다. 이 프로젝트에서는 이 아이디어를 의료 영상 분할로 확장합니다. 원본 이미지를 조건으로 활용하고 무작위 노이즈로부터 여러 개의 분할 맵을 생성한 다음 이를 앙상블링하여 최종 결과를 얻습니다. 이 접근 방식은 의료 이미지의 불확실성을 포착하고 여러 벤치마크에서 이전 방법보다 성능이 뛰어납니다.
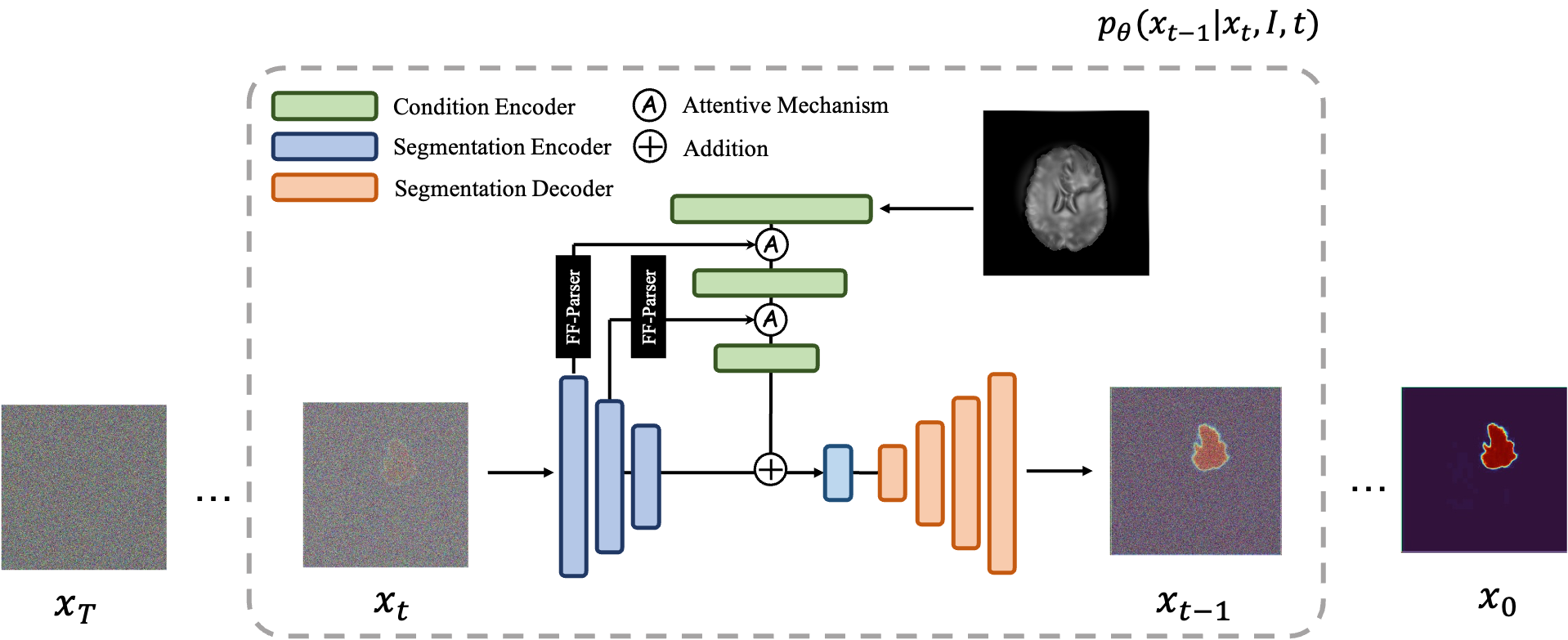 | 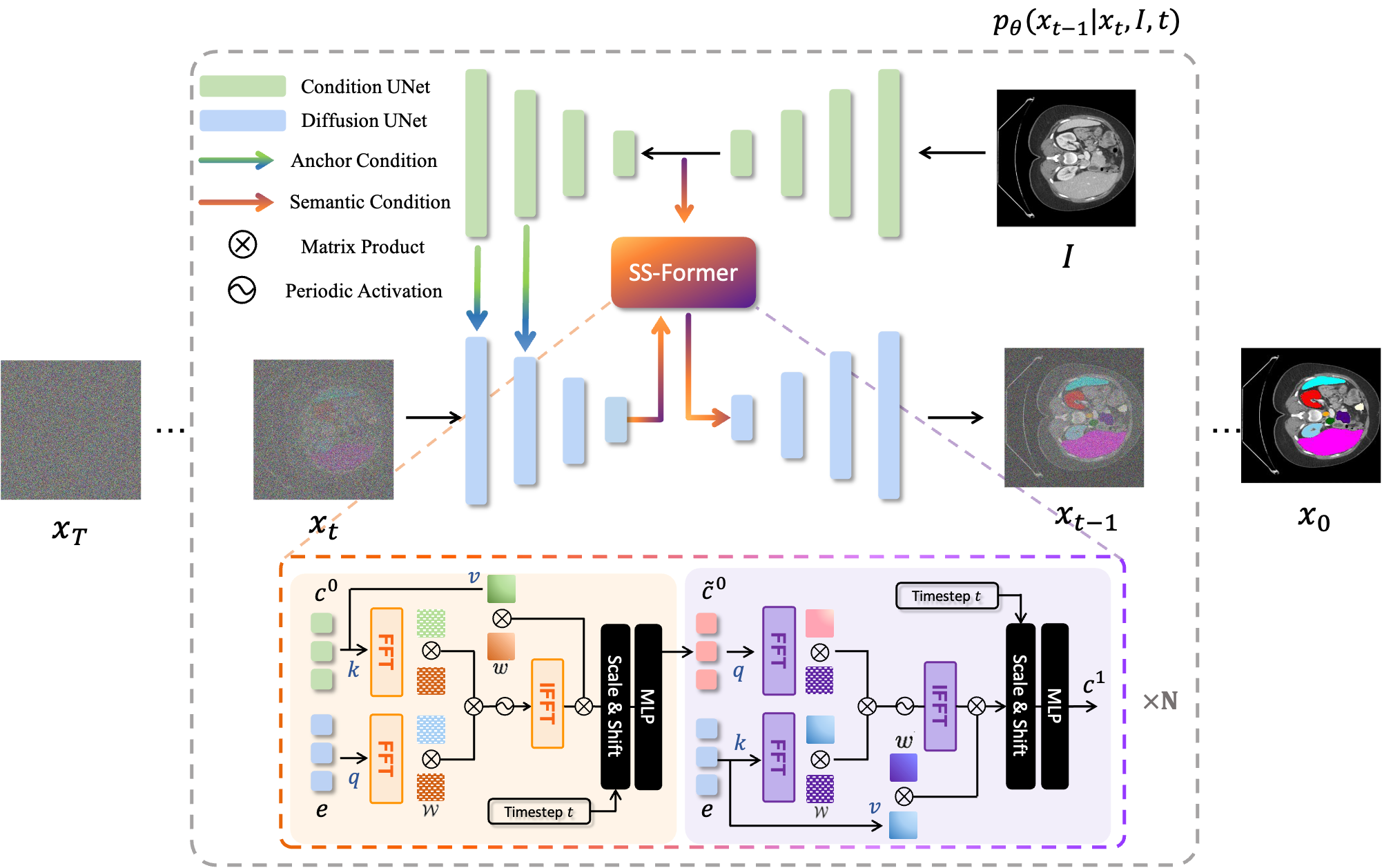 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True 설정하여 매우 빠른 샘플링(1000단계 20단계 ⭕️)을 즐겨보세요.python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
훈련을 위해 다음을 실행하십시오: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
샘플링을 위해 다음을 실행합니다: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
평가를 위해 python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* 실행합니다.
기본적으로 샘플은 ./results/ 에 저장됩니다.
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
훈련을 위해 다음을 실행합니다: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
샘플링을 위해 다음을 실행합니다. python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
다른 데이터 세트에서 MedSegDiff를 실행하는 것은 간단합니다. ./guided_diffusion/isicloader.py 또는 ./guided_diffusion/bratsloader.py 다음에 다른 데이터 로더 파일을 작성하면 됩니다. 문제가 발생하면 오픈 이슈에 오신 것을 환영합니다. 데이터 세트 확장에 기여해 주시면 감사하겠습니다. 의료영상은 자연스러운 영상과 달리 업무에 따라 많이 달라집니다. 방법의 일반화를 확장하려면 모든 사람의 노력이 필요합니다.
논문의 MedSegDiff-B와 같은 정밀한 모델을 훈련하려면 모델 하이퍼파라미터를 다음과 같이 설정하십시오.
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
확산 하이퍼파라미터는 다음과 같습니다:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
샘플링 속도를 높이려면:
--diffusion_steps 50 --dpm_solver True
여러 GPU에서 실행:
--multi-gpu 0,1,2 (for example)
하이퍼파라미터를 다음과 같이 훈련합니다:
--lr 5e-5 --batch_size 8
샘플링에 --num_ensemble 5 설정합니다.
훈련에서 약 100,000단계를 실행하면 대부분의 데이터 세트에 수렴됩니다. 대부분의 이후 단계에서 손실이 줄어들지는 않지만 결과의 품질은 여전히 향상되고 있습니다. 이러한 프로세스는 이미지 생성과 같은 다른 DPM 응용 프로그램에서도 관찰됩니다. 똑똑한 사람이 이유를 말해 줄 수 있기를 바랍니다.
비교가 필요하기 때문에 더 작은 배치 크기(24GB GPU에서 실행하기에 적합)로 성능을 곧 게시할 예정인가요?
모든 잠재력을 발휘하기 위한 설정은 (MedSegDiff++)입니다:
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
그런 다음 배치 크기 --batch_size 64 로 훈련하고 앙상블 번호 --num_ensemble 25 로 샘플링합니다.
MedSegDiff에 참여하신 것을 환영합니다. 어떤 기술이라도 성능을 향상시키거나 알고리즘 속도를 높일 수 있다는 점은 높이 평가됩니다. 저는 Nature 저널/CVPR 출판을 목표로 MedSegDiff V2를 작성하고 있습니다. 기여자들을 공동저자로 기재하게 되어 기쁩니다.
코드는 openai/improved-diffusion, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion,guide-diffusion, bigmb/Unet-Segmentation-Pytorch-Nest에서 많이 복사되었습니다. -Unets, nnUnet, 루시드레인/vit-pytorch
인용해주세요
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu