
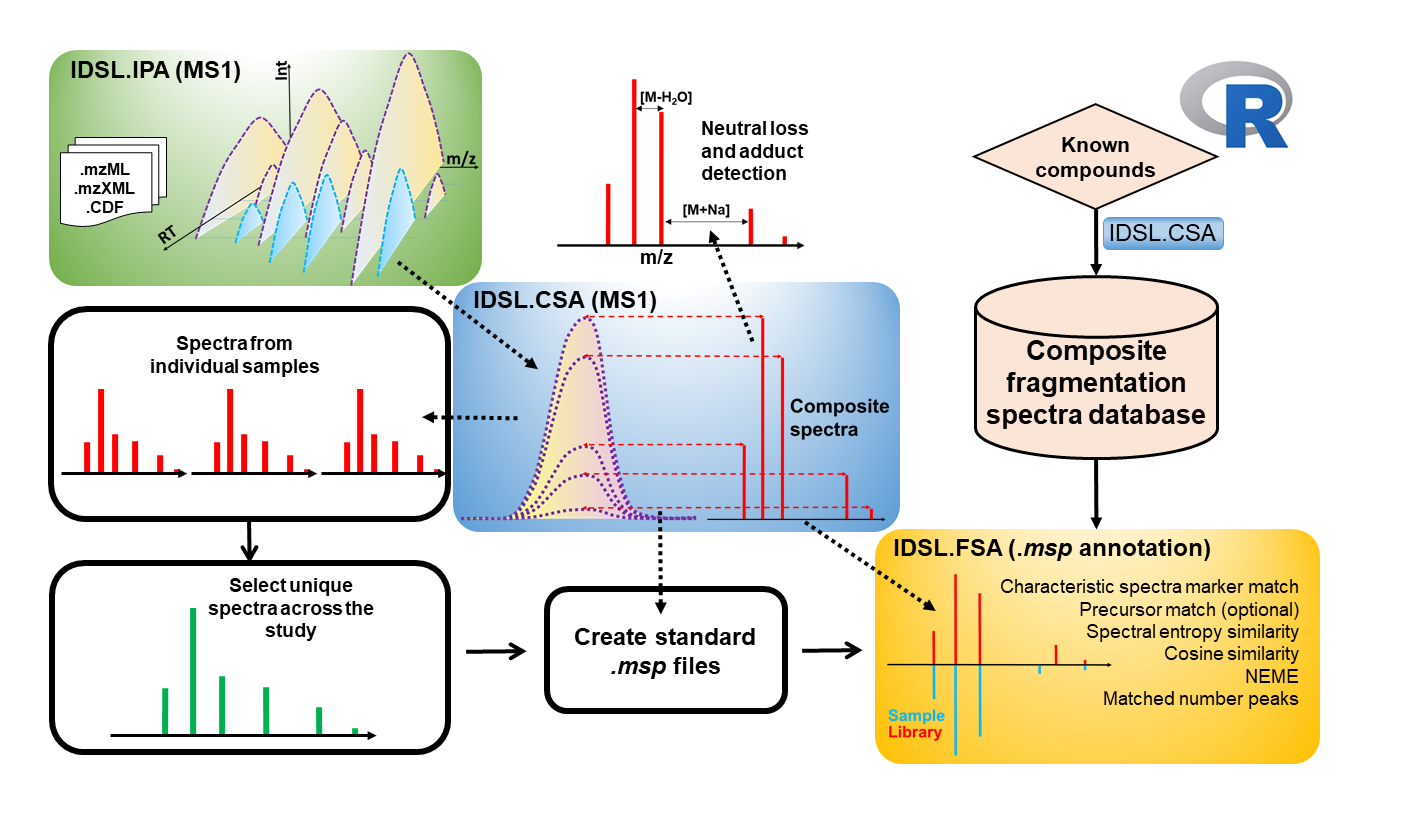
질량 분석법 데이터 분석을위한 복합 스펙트럼 분석 (IDSL.CSA) R 패키지는 대사체 및 Exposomics (IDSL.ME)를위한 Integrated Data Science Laboratory 에 의해 개발되었습니다. 이 패키지는 MS1 전용 복합 스펙트럼 디컨 볼 루션 분석 ( CSA ), 데이터 종속 획득 ( DDA ) 및 MS E를 포함한 다양한 데이터 독립적 획득 ( DIA ) 방법과 같은 다양한 분석 방법을 통해 얻은 단편화 스펙트럼의 디컨 볼 루션에 사용될 수 있습니다. , ALL-EON 단편화 (AIF) 및 SWATH-MS 분석. IDSL.CSA 패키지의 목적은 데이터 분석 프로세스를 간소화하고 대사체 및 설명 분야의 전체 화학 구조 주석을 개선하는 데 도움이됩니다.
Start 탭에서 PARAM0006을 사용해보십시오. install.packages("IDSL.CSA")
IDSL.CSA 워크 플로우를 사용하여 질량 분석 데이터 ( MZXML , MZML , NETCDF )를 처리하기 전에 질량 분석법 데이터는 IDSL.IPA 워크 플로를 사용하여 처리되어 피크 ( M/Z-RT )의 크로마토 그래피 정보를 얻습니다. IDSL.IPA 워크 플로를 사용하여 개별 및 집계 된 정렬 된 피크리스트의 크로마토 그래피 정보가 생성되면 IDSL.CSA 매개 변수 스프레드 시트를 다운로드 한 다음 그에 따라 매개 변수를 선택한 다음이 스프레드 시트를 IDSL.CSA 워크 플로우의 입력으로 사용하십시오.
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
다음 단계를 따르십시오. 빠른 사례 연구 (n = 33) ST002263은 HILIC-ESI-POS/NEG 모드에서 수집 된 Thermo Q Exactive HF 하이브리드 궤도 랩 데이터를 갖습니다.
idsl.ipa에 기술 된 방법을 사용하여 원시 질량 분석 데이터 및 크로마토 그래피 정보를 처리합니다.
복합 스펙트럼 분석은 풀 스케일 분석을 위해 5 개의 개별 섹션으로 분배 된 39 개의 매개 변수가 필요합니다. 이 연구의 경우 idsl.csa 매개 변수 스프레드 시트에 제시된 기본 매개 변수 값을 사용하십시오. 다음으로 정보를 제공하십시오
2.1. CSA 워크 플로를 처리하려면 Start 탭에서 Param0001 의 예를 선택하십시오.
2.2. HRMS 데이터 위치 주소 의 CSA0005 (MS1 레벨 HRMS 데이터)
2.3. idsl.ipa 워크 플로에서 생성 된 peaklists 디렉토리의 주소 용 CSA0008
2.4. idsl.ipa 워크 플로에서 생성 된 peak_alignment 디렉토리 주소의 CSA0009
2.5. 출력 위치 에 대한 CSA0011 (.MSP 파일 및 EIC)
2.6. 계산 능력에 따라 CSA0004를 사용하여 처리 스레드 수를 늘릴 수도 있습니다.
R/rstudio 콘솔 또는 터미널 에서이 명령을 실행하십시오.
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
CSA0011 에 제공 한 주소에서 결과를 구문 분석 할 수 있습니다.
4.1. CSA_MSP에는 .msp 파일이 포함됩니다
4.2. CSA_ADDUCT_ANNOTATION 에는 잠재적 부가물 정보가 포함 된 피크리스트가 포함됩니다
4.3. peak_alignment_subset 각 CSA 클러스터의 주요 이온에 대한 정렬 된 피크 테이블의 서브 세트
4.4. Aligned_spectra_table은 정렬 된 테이블의 CSA 집계에 대한 정보가 포함됩니다.
[1] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL.CSA : 표적화되지 않은 대사체 데이터 세트의 화학적 주석을위한 복합 스펙트럼 분석. 분석 화학 , 2023 , 95 (25) , 9480–9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, DK Idsl. IPA는 표적이지 않은 LC/HRMS 데이터 세트의 유기 화학 공간을 특징으로합니다. Proteome Research , 2022 , 21 (6) , 1485-1494.