Este pacote fornece modelos U-net treinados para segmentação pulmonar. Por enquanto, quatro modelos estão disponíveis:
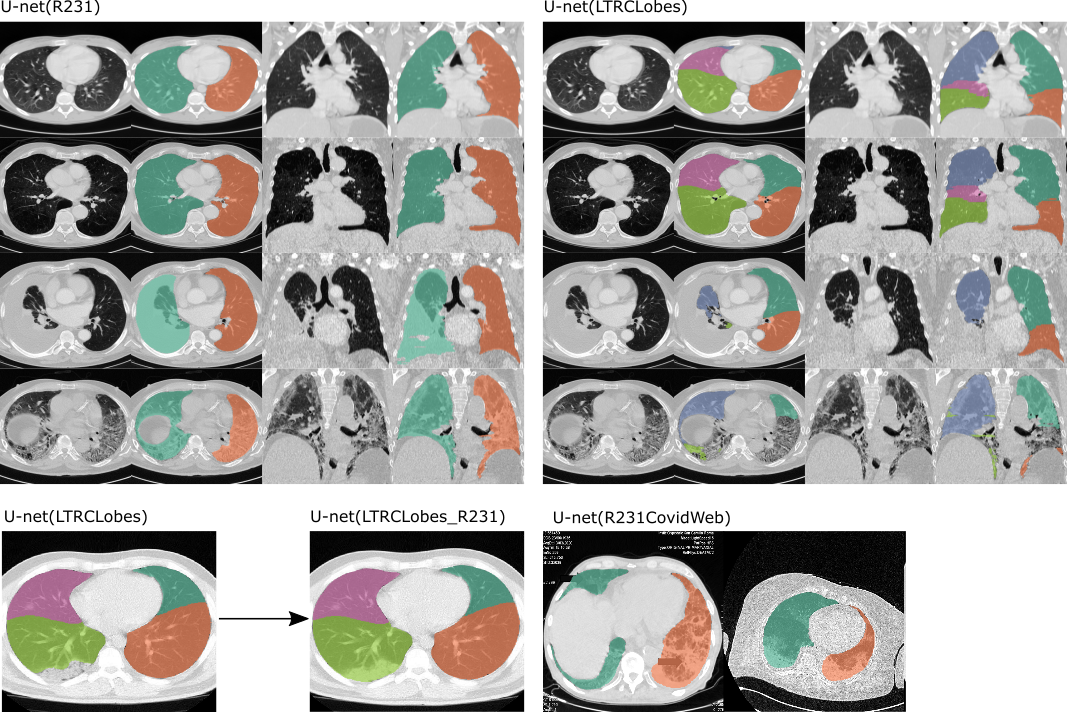
U-net (R231): Este modelo foi treinado em um conjunto de dados grande e diversificado que cobre uma ampla gama de variabilidade visual. O modelo realiza a segmentação em fatias individuais, extrai o pulmão direito-esquerdo separadamente, incluindo bolsas de ar, tumores e efusões. A traqueia não será incluída na segmentação pulmonar. https://doi.org/10.1186/s41747-020-00173-2
U-net (LTRCLobes): Este modelo foi treinado em um subconjunto do conjunto de dados LTRC. O modelo realiza a segmentação de lobos pulmonares individuais, mas apresenta desempenho limitado quando patologias densas estão presentes ou quando fissuras não são visíveis em todos os cortes.
U-net (LTRCLobes_R231): Isso executará o modelo R231 e LTRCLobes e fundirá os resultados. Os falsos negativos de LTRCLobes serão preenchidos pelas previsões R231 e mapeados para um rótulo vizinho. Os falsos positivos de LTRCLobes serão removidos. O processo de fusão é computacionalmente intensivo e pode, dependendo dos dados e resultados, levar vários minutos por volume.
Rede U(R231CovidWeb)
Exemplos dos dois modelos aplicados . Esquerda: U-net (R231), distinguirá entre o pulmão esquerdo e direito e incluirá áreas muito densas, como derrames (terceira linha), tumor ou fibrose grave (quarta linha). À direita: U-net (LTRLobes), distinguirá entre lobos pulmonares, mas não incluirá áreas muito densas. LTRCLobes_R231 fundirá os resultados LTRCLobes e R231. R231CovidWeb é treinado com dados adicionais do COVID-19.
Semântica de saída :
Dois modelos de etiquetas (esquerda-direita):
1 = Pulmão direito
2 = Pulmão esquerdo
Cinco modelos de rótulos (lóbulos pulmonares):
1 = Lobo superior esquerdo
2 = Lobo inferior esquerdo
3 = Lobo superior direito
4 = Lobo médio direito
5 = Lobo inferior direito
Para pesquisas mais interessantes sobre dados de tomografia computadorizada de pulmão, confira o site do nosso grupo de pesquisa: https://www.cir.meduniwien.ac.at/research/lung/
Se você usar este código ou um dos modelos treinados em seu trabalho, consulte:
Hofmanninger, J., Prayer, F., Pan, J. et al. A segmentação pulmonar automática em imagens de rotina é principalmente um problema de diversidade de dados, não um problema de metodologia. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Este artigo contém uma descrição detalhada do conjunto de dados utilizado, uma avaliação completa do modelo U-net (R231) e uma comparação com métodos de referência.
pip install lungmask
ou
pip install git+https://github.com/JoHof/lungmask
No Windows, dependendo da sua configuração, pode ser necessário instalar o torch previamente: https://pytorch.org
O tempo de execução entre a inferência suportada apenas pela CPU e pela GPU varia muito. Usando a GPU, o processamento de um volume leva apenas alguns segundos, usando apenas a CPU levará vários minutos. Para usar a GPU, certifique-se de que a instalação da tocha tenha suporte CUDA. Em caso de erros de cuda sem memória, reduza o tamanho do lote para 1 com o argumento opcional --batchsize 1
lungmask INPUT OUTPUT
Se INPUT apontar para um arquivo, o arquivo será processado. Se INPUT apontar para um diretório, o diretório será pesquisado pela série DICOM. O maior volume encontrado (em termos de número de voxels) será utilizado para calcular a máscara pulmonar. OUTPUT é o nome do arquivo de saída. Todos os formatos ITK são suportados.
Escolha um modelo:
O U-net(R231) será usado como padrão. No entanto, você pode especificar um modelo alternativo como LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Para opções adicionais digite:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image deve ser um objeto SimpleITK.
Carregue um modelo alternativo assim:
inferer = LMInferer(modelname="R231CovidWeb")
Para usar a capacidade de fusão do modelo (por exemplo, LTRCLobes_R231), use:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
A partir da versão 0.2.9, matrizes numpy são suportadas como volumes de entrada. Este modo assume que a matriz numpy de entrada tem o seguinte formato para cada eixo:
O modelo funciona apenas em fatias completas. O corte a ser processado deve mostrar o pulmão cheio e o pulmão deve ser circundado por tecido para ser segmentado. No entanto, o modelo é bastante estável para casos com campo de visão recortado, desde que o pulmão esteja rodeado por tecido.
lungmask INPUT OUTPUT --modelname R231CovidWeb
O modelo U-net regular (R231) funciona muito bem para tomografias computadorizadas COVID-19. No entanto, coleções de cortes e relatos de casos da web são frequentemente cortados, anotados ou codificados em formatos de imagem regulares, de modo que os valores originais da unidade hounsfield (HU) só possam ser estimados. Os dados de treinamento do modelo U-net (R231CovidWeb) foram aumentados com fatias COVID-19 que foram mapeadas de formatos de imagem regulares para HU. Os dados foram coletados e preparados pela MedSeg (//medicalsegmentation.com/covid19/). Embora a rede U regular (R231) tenha mostrado resultados muito bons para essas imagens, pode haver casos em que este modelo produzirá segmentações ligeiramente melhoradas. Observe que você deve mapear imagens de volta para HU ao usar imagens da web. Esta postagem do blog descreve como você pode fazer isso. Alternativamente, você pode definir a tag --noHU . 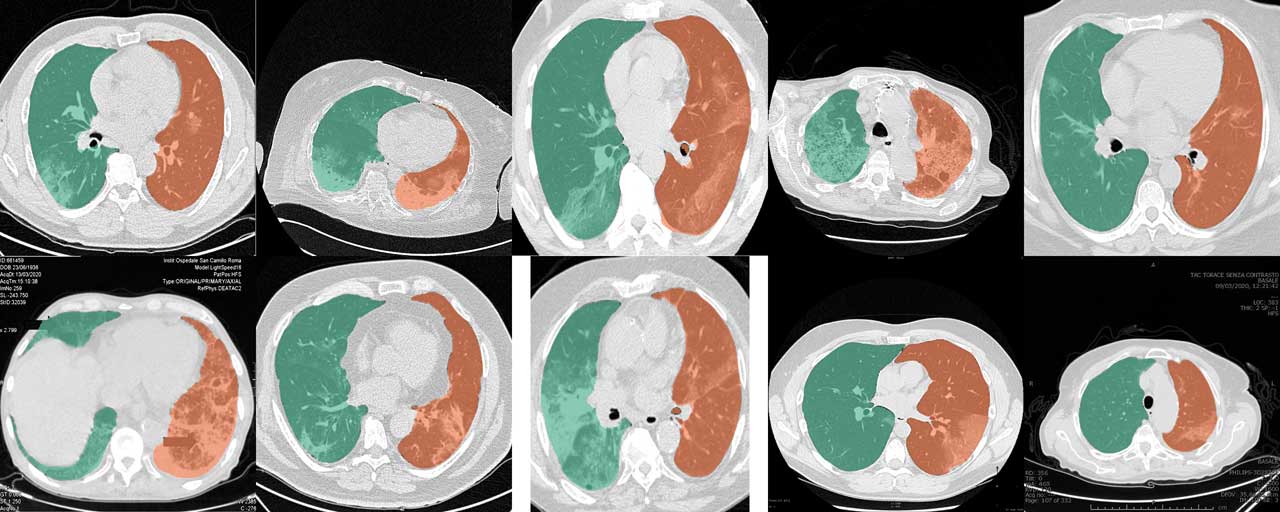
Este recurso está disponível apenas nas versões entre 0.2.5 e 0.2.14. A partir da versão 0.2.5 essas imagens são suportadas. Use a tag --noHU se você processar imagens que não estão codificadas em HU. Tenha em mente que os modelos foram treinados em tomografias computadorizadas adequadas codificadas em HU. Os resultados em imagens recortadas, anotadas, com intensidade muito alta e muito baixa podem não ser muito confiáveis. Ao usar a tag --noHU , apenas fatias únicas podem ser processadas.