В этом репозитории представлен наш сценарий для воспроизведения корреляционного corrplot генетической корреляции (рис. 2) на основе результатов двумерной регрессии оценки LD (Kanai, M. et al ., Nat. Genet. 2018).
install.packages("dplyr")
devtools::install_github("mkanai/corrplot")
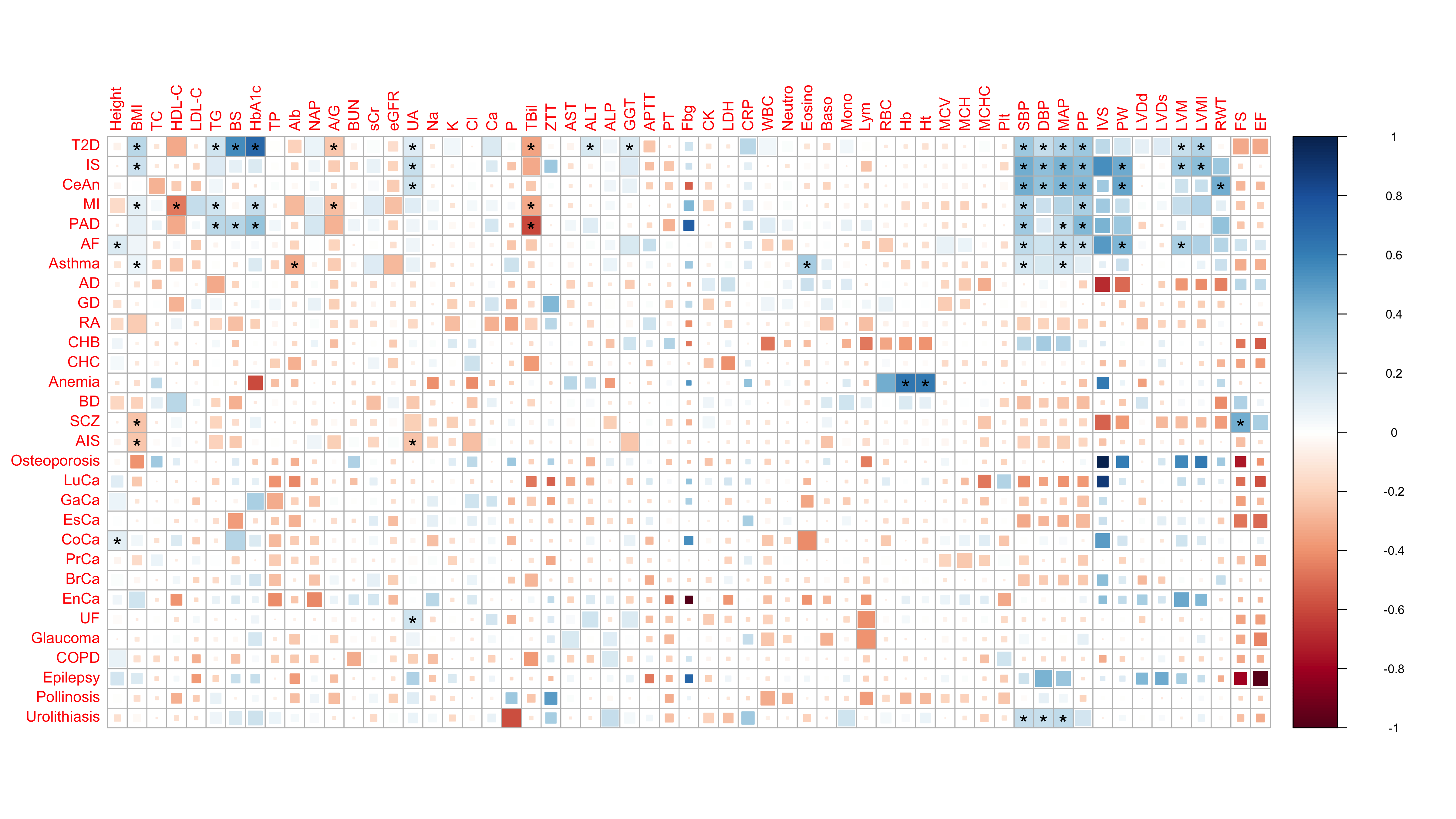
Мы модифицировали график для визуализации парных генетических корреляций, оцененных с помощью двумерной регрессии оценки LD. Более крупные квадраты соответствуют более значимым FDR corrplot(method = 'psquare', p.mat = p.mat, ...) ). Значимые корреляции (FDR <0,05) отмечены звездочками ( corrplot(sig = 'pch', sig.level = 0.05, pch = '*') ).
Rscript plot_corrplot_rg.R input_example/input_rg.txt input_example/traitlist.txt
input_rg.txt ).В этом файле представлен список всех парных генетических корреляций, оцененных с помощью программного обеспечения ldsc. Скрипт ожидает, что все строки уникальны ( т. е. по одной строке на каждую пару признаков). Обязательные поля следующие:
p1_category : Категория признака признака 1.p1 : Черта 1p2_category : Категория признака признака 2.p2 : Черта 2rg : генетическая корреляцияp : P-значениеq : q-значение FDRtraitlist.txt )Этот файл содержит список характеристик и их категорий. Он определяет цвет каждой категории на рисунке. Обязательные поля следующие:
CATEGORY : Категория признакаTRAIT : название черты.COLOR : Цвет категорииПример вывода показан ниже. Чтобы получить опубликованный рисунок, мы отредактировали PDF-файл с помощью Adobe Illustrator.
Оригинальный пакет corrplot :
Пример данных и опубликованный рисунок:
Масахиро Канаи ([email protected])
http://mkanai.github.io/