Имейте в виду, что этот репозиторий предназначен только для архивных целей. Автономные функции ANCOM в этом хранилище больше не поддерживаются. Пользователю настоятельно рекомендуется использовать функции ancom или ancombc в нашем пакете ANCOMBC R. Отчеты об ошибках можно найти в репозитории ANCOMBC.
Текущий код реализует ANCOM в наборах поперечных и продольных данных, позволяя при этом использовать ковариаты. Для запуска кода R необходимо включить следующие библиотеки:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Мы приняли методологию ANCOM-II в качестве этапа предварительной обработки для работы с различными типами нулей перед выполнением дифференциального анализа численности.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : Кадр данных или матрица, представляющая наблюдаемую таблицу OTU/SV с таксонами в строках ( rownames ) и образцами в столбцах ( colnames ). Обратите внимание, что это таблица абсолютной численности, не преобразуйте ее в таблицу относительной численности (где суммы столбцов равны 1).meta_data : кадр данных или матрица всех переменных и ковариат, представляющих интерес.sample_var : Персонаж. Имя столбца, в котором хранятся идентификаторы образцов.group_var : Персонаж. Название группового индикатора. group_var требуется для обнаружения структурных нулей и выбросов. Определения различных нулей (структурный ноль, ноль выброса и ноль выборки) см. в ANCOM-II.out_cut Числовая дробь от 0 до 1. Для каждого таксона наблюдения с долей распределения смеси меньше, чем out_cut , будут обнаружены как выбросы с нулями; в то время как наблюдения с долей распределения смеси больше 1 - out_cut будут обнаружены как значения выброса.zero_cut : числовая дробь от 0 до 1. Таксоны с долей нулей больше, чем zero_cut не включаются в анализ.lib_cut : Числовой. Образцы с размером библиотеки меньше lib_cut в анализ не включаются.neg_lb : Логично. TRUE указывает, что таксон будет классифицирован как структурный ноль в соответствующей экспериментальной группе с использованием его асимптотической нижней границы. Более конкретно, neg_lb = TRUE указывает, что вы используете оба критерия, указанные в разделе 3.2 ANCOM-II, для обнаружения структурных нулей; В противном случае neg_lb = FALSE будет использовать только уравнение 1 из раздела 3.2 ANCOM-II для объявления структурных нулей. feature_table : кадр данных предварительно обработанной таблицы OTU.meta_data : кадр данных предварительно обработанных метаданных.structure_zeros : Матрица состоит из 0 и 1, где 1 указывает, что таксон идентифицирован как структурный ноль в соответствующей группе.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : Кадр данных, представляющий таблицу OTU/SV с таксонами в строках ( rownames ) и образцами в столбцах ( colnames ). Это может быть выходное значение из feature_table_pre_process . Обратите внимание, что это таблица абсолютной численности, не преобразуйте ее в таблицу относительной численности (где суммы столбцов равны 1).meta_data : Фрейм данных переменных. Может быть выходным значением из feature_table_pre_process .struc_zero : Матрица состоит из 0 и 1, где 1 указывает, что таксон идентифицирован как структурный ноль в соответствующей группе. Может быть выходным значением из feature_table_pre_process .main_var : Персонаж. Имя основной интересующей переменной. ANCOM v2.1 в настоящее время поддерживает категориальный main_var .p_adjust_method : Персонаж. Указание метода корректировки значений p для множественных сравнений. По умолчанию – «BH» (процедура Бенджамини-Хохберга).alpha : Уровень значимости. По умолчанию — 0,05.adj_formula : строка символов, представляющая формулу корректировки (см. пример).rand_formula : строка символов, представляющая формулу случайных эффектов в lme . Подробности см. в разделе ?lme .lme_control : список, определяющий контрольные значения для соответствия lme. Подробности см. в разделе ?lmeControl . 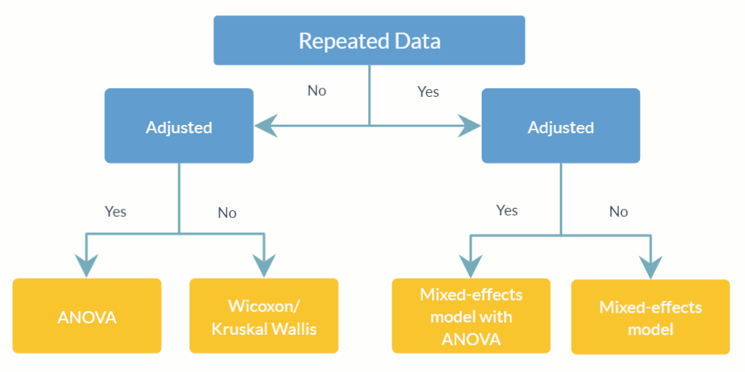
out : Кадр данных со статистикой W для каждого таксона и последующими столбцами, которые являются логическими индикаторами того, является ли OTU или таксон дифференциальной численностью при ряде пороговых значений (0,9, 0,8, 0,7 и 0,6). detected_0.7 обычно используется. Однако вы можете выбрать detected_0.8 или detected_0.9 , если хотите быть более консервативным в своих результатах (меньший FDR), или использовать detected_0.6 , если вы хотите исследовать больше открытий (большая мощность).
fig : Объект ggplot графика вулкана.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Здесь мы хотели бы знать, есть ли какие-то структурные нули на разных уровнях delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Здесь мы хотели бы знать, есть ли какие-то структурные нули на разных уровнях delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Здесь мы хотели бы знать, есть ли какие-то структурные нули на разных уровнях delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )