Быстрая пакетная загрузка файлов BigWig, содержащих данные эпигенных треков и соответствующие последовательности, с использованием графического процессора для приложений глубокого обучения.
Bigwig-loader в основном зависит от библиотеки Rapidsai Kvikio и Cupy, которые лучше всего устанавливать с помощью conda/mamba. Bigwig-loader теперь также можно установить с помощью conda/mamba. Чтобы создать новую среду с установленным bigwig-loader:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderИли добавьте это в файл Environment.yml:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderи обновить:
mamba env update -f environment.ymlBigwig-loader также можно установить с помощью pip в среде, в которой уже установлены библиотека Rapidsai Kvikio и Cupy:
pip install bigwig-loaderМы обернули BigWigDataset в итерируемый набор данных PyTorch, который вы можете использовать напрямую:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Объект набора данных, не зависящий от платформы, можно импортировать из bigwig_loader.dataset . Этот объект набора данных возвращает тензоры купи. Тензоры Cupy привязаны к интерфейсу массива cuda и могут быть преобразованы с нулевым копированием в тензоры JAX или тензорного потока.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Дополнительные примеры см. в каталоге примеров.
Эта библиотека предназначена для загрузки пакетов данных одинаковой размерности, что позволяет сделать некоторые допущения, которые могут ускорить процесс загрузки. Как видно из графика ниже, при загрузке небольшого объема данных pyBigWig работает очень быстро, но не использует пакетный характер загрузки данных для машинного обучения.
В приведенном ниже тесте мы также создали загрузчики данных PyTorch (с set_start_method('spawn')) с использованием pyBigWig для сравнения с реалистичным сценарием, когда на один графический процессор будет использоваться несколько процессоров. Мы видим, что пропускная способность загрузчика данных ЦП не увеличивается линейно с количеством ЦП, и поэтому становится трудно получить необходимую пропускную способность для поддержания насыщения ГП, обучающего нейронную сеть, на этапах обучения.
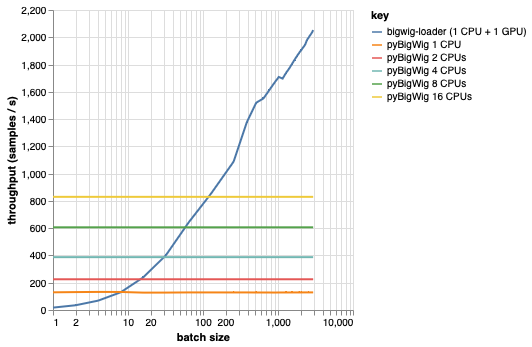
Эту проблему решает bigwig-loader. Это пример использования bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml В этой среде вы сможете запустить pytest -v и убедиться, что тесты пройдены успешно. ПРИМЕЧАНИЕ. Для использования bigwig-loader вам понадобится графический процессор!
В этом разделе описаны шаги, необходимые для добавления новых функций. Если что-то неясно, пожалуйста, откройте тему.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install , чтобы установить крючки предварительной фиксацииТесты находятся в каталоге тестов. Одним из наиболее важных тестов является test_against_pybigwig, который проверяет, что если в pyBigWIg есть ошибка, то она также есть и в bigwig-loader.
pytest -vv .Когда станут доступны программы GitHub с графическими процессорами, мы также хотели бы запустить эти тесты в CI. Но пока вы можете запускать их локально.
Если вы используете эту библиотеку, рассмотрите возможность цитирования:
Ретель, Йорен Себастьян, Андреас Поэльманн, Джош Чиу, Андреас Штеффен и Джорк-Арне Клеверт. «Быстрый загрузчик данных машинного обучения для эпигенетических треков из файлов BigWig». Биоинформатика 40, вып. 1 (1 января 2024 г.): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}