
(c) 2017 Тимоти Беккер и Ван-Пин Ли
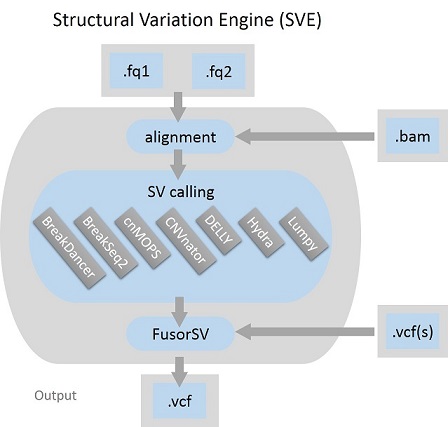
SVE — это механизм выполнения на основе сценариев Python для обнаружения структурных изменений (SV), который может использоваться для любых уровней входных данных, необработанных FASTQ, согласованных BAM или формата вызова вариантов (VCF) и генерирует унифицированный VCF в качестве выходных данных. По своей конструкции SVE по умолчанию состоит из выравнивания, перенастройки и набора современных алгоритмов вызова SV. Это BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra и LUMPY. Также встроен FusorSV, который представляет собой подход к интеллектуальному анализу данных для оценки производительности и объединения наборов вызовов из ансамбля алгоритмов вызова SV.
Пожалуйста, установите ROOT-среду.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeПожалуйста, проверьте файлы заголовков Python2.7 и измените «CFLAGS_FUSOR_SV» в Makefile. Файлы заголовков могут находиться в каталоге «/usr/include/python2.7» и вместо этого использовать «CFLAGS_FUSOR_SV=-I /usr/include/python2.7».
make FusorSV
Или вы можете установить FusorSV с помощью setup.py.
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
В качестве альтернативы предоставляются Dockerfile и образ Docker. Обратите внимание, что sudo может потребоваться для использования докера в зависимости от настроек вашего компьютера.
cd SVE
docker build .
Извлеките образ докера из репозитория.
docker pull wanpinglee/sve
SVE построен на основе /tools/SVE. Проверьте помощь по
/tools/SVE/bin/sve
Короткие чтения в FASTQ будут сопоставлены с данным FASTA, и будет сгенерирован отсортированный BAM.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
Если чтения заданы в формате BAM, функция reign переназначит чтения на формат FASTA и сгенерирует отсортированный BAM. Мы используем SpeedSeq для выполнения перестройки.
bin/sve realign -r <FASTA> <BAM>
Существует семь алгоритмов вызова SV, которые можно использовать для вызова SV. VCF будет создан.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
После вызова каждый образец может иметь несколько VCF в зависимости от того, сколько вызывающих абонентов использовалось. Пожалуйста, соберите VCF образца в папку.
vcfs должен использовать идентификаторы SVE для указания вызывающих абонентов.
| Идентификатор СВЕ | Звонящий |
|---|---|
| 4 | Брейкдансер (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnator (v0.3.3) |
| 11 | ДЕЛЛИ (v2) |
| 14* | ГеномSTRIP |
| 17 | Гидра |
| 18 | комковатый |
| 35 | БрейкСек (v2.2) |
| 0 | Правда (необязательно) |
Примечание*. Из-за проблем с лицензией GenomeSTRIP не встроен в SVE. Однако модель FusorSV по умолчанию способна обрабатывать GenomeSTRIP VCF.
Примеры входных файлов vcf можно организовать следующим образом. Обратите внимание, что vcfFiles является аргументом -i для FusorSV.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Согласно S0.vcf, будет создана новая модель, и VCF будут объединены с помощью новой модели.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
Проект распространяется по лицензии GPL-3.0. Подробную информацию см. в разделе ЛИЦЕНЗИЯ.