อัปเดตล่าสุด: 9/30/2024
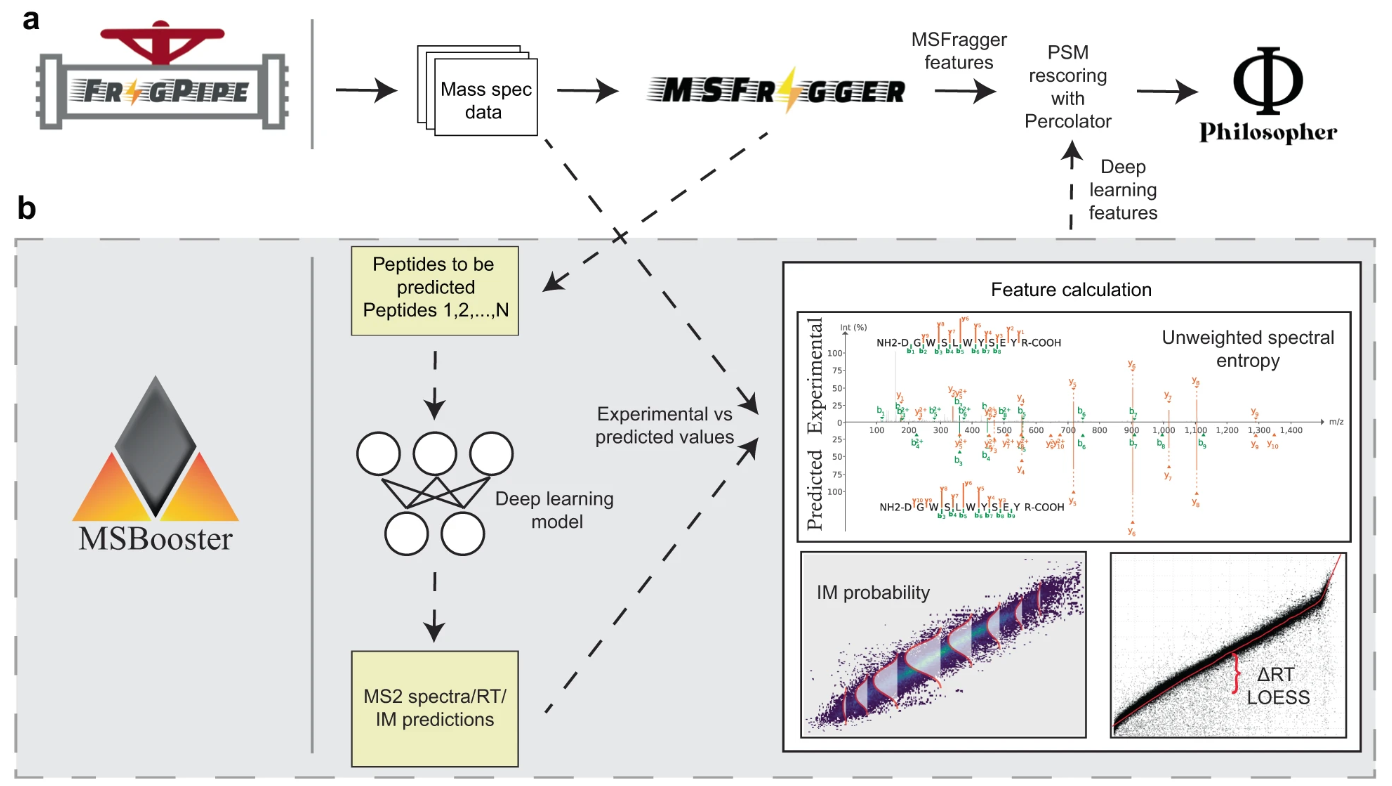
MSBooster เป็นเครื่องมือสำหรับผสมผสานการคาดการณ์สเปกตรัมเข้ากับการจับคู่เปปไทด์สเปกตรัม (PSM) โดยทำคะแนนใหม่ในข้อมูลโปรตีโอมิกส์โครมาโทกราฟีของเหลวจากล่างขึ้นบน แบ่งออกเป็น 4 ขั้นตอนคร่าวๆ ดังนี้
การแยกเปปไทด์จาก PSM ในผลการค้นหา และการจัดรูปแบบสำหรับไฟล์อินพุตของเครื่องทำนายการเรียนรู้เชิงลึก (ML/DL)
การเรียกโมเดลการทำนายและการบันทึกเอาต์พุต
การคำนวณคุณสมบัติ
เพิ่มคุณสมบัติใหม่ให้กับไฟล์ผลการค้นหา
MSBooster เข้ากันได้กับการค้นหาฐานข้อมูลหลายประเภท รวมถึง HLA immunopeptidomics, DDA และ DIA และโปรตีโอมิกส์เซลล์เดียว มันถูกรวมเข้ากับ FragPipe และรวมอยู่ในขั้นตอนการทำงานมากมาย MSBooster ได้รับการพัฒนาโดยคำนึงถึงเครื่องมือ FragPipe อื่นๆ เช่น FragPipe-PDV
MSBooster ได้รับการออกแบบมาเพื่อรองรับรูปแบบไฟล์อินพุตและรุ่นที่หลากหลาย:
| เอาต์พุตแมสสเปกโตรมิเตอร์ |
|---|
| .mzML |
| .มก |
| ไฟล์พีเอสเอ็ม |
|---|
| .เข็มหมุด |
| .pepXML (กำลังดำเนินการ) |
| โมเดลการคาดการณ์ |
|---|
| DIA-NN |
| โมเดลโคอินะ |
MSBooster สามารถทำงานได้ในระบบ Windows และ Linux หากใช้ FragPipe ก็ไม่จำเป็นต้องมีขั้นตอนการติดตั้งอื่นใดนอกจากการติดตั้ง FragPipe MSBooster อยู่ในแท็บ "การตรวจสอบ" เลือกเพื่อเปิดใช้งานคุณสมบัติเวลาการเก็บรักษาด้วย "Predict RT" และคุณสมบัติสเปกตรัม MS/MS ด้วย "Predict spectra" โปรดดูเอกสาร FragPipe สำหรับวิธีเรียกใช้การวิเคราะห์
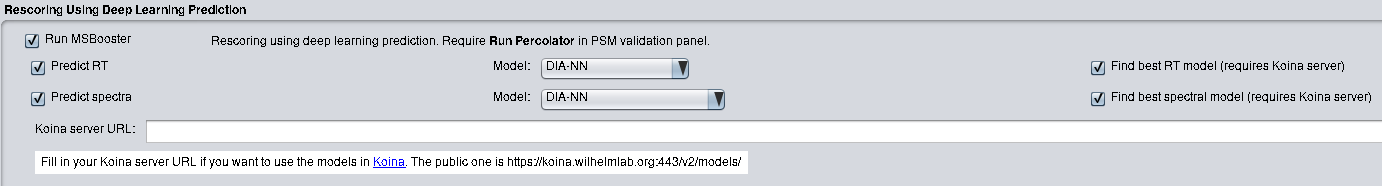
หากใช้ MSBooster แบบสแตนด์อโลนเพื่อรันในบรรทัดคำสั่ง โปรดดาวน์โหลดไฟล์ jar ล่าสุดจาก Releases MSBooster ยังต้องการ DIA-NN สำหรับการทำนาย MS/MS และ RT โปรดติดตั้ง DIA-NN และจดเส้นทางไปยังไฟล์ปฏิบัติการ DIA-NN (เช่น DiaNN.exe สำหรับ Windows, diann-1.8.1.8 สำหรับ Linux)
คุณสามารถรัน MSBooster ได้โดยใช้คำสั่งที่คล้ายกับคำสั่งต่อไปนี้:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
พารามิเตอร์ขั้นต่ำที่ต้องส่งคือ:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
แม้ว่าคุณจะสามารถส่งพารามิเตอร์เหล่านี้ทีละรายการได้ แต่จะง่ายกว่าที่จะวางพารามิเตอร์ไว้ในแต่ละบรรทัดของไฟล์ paramsList โปรดดูที่ msbooster_params.txt สำหรับเทมเพลต
พารามิเตอร์ด้านล่างนี้มีไว้สำหรับการใช้งานทั่วไป พารามิเตอร์เฉพาะของ Koina อยู่ในเอกสารประกอบของ Koina
paramsList (String) : ตำแหน่งไปยังไฟล์ข้อความที่มีพารามิเตอร์สำหรับการรันนี้
fragger (String) : เส้นทางไฟล์ของไฟล์ fragger.params จากการรัน MSFragger MSBooster จะอ่านพารามิเตอร์หลายตัวและปรับพารามิเตอร์ภายในตามพารามิเตอร์เหล่านั้น เช่น ความทนทานต่อข้อผิดพลาดของมวลส่วนและการชดเชยมวล
outputDirectory (String) : ตำแหน่งที่จะส่งออกไฟล์ใหม่
editedPin (String) : MSBooster จะตั้งชื่อไฟล์ใหม่ตามไฟล์ที่ให้ไว้ ตัวอย่างเช่น A.pin จะมีคู่ที่เรียกว่า A_edited.pin หากต้องการเปลี่ยนจากค่าเริ่มต้นเป็น "แก้ไขแล้ว" ให้ระบุสตริงใหม่ที่นี่
renamePin (int) : ว่าจะสร้างไฟล์พินใหม่หรือเขียนไฟล์เก่าใหม่ ค่าเริ่มต้นที่นี่คือ 1 ซึ่งจะไม่เขียนทับ การตั้งค่านี้เป็น 0 จะเขียนทับไฟล์พินเก่า
deletePreds (boolean) : ว่าจะลบไฟล์ที่จัดเก็บการคาดการณ์โมเดลหลังจากเสร็จสิ้นการรันสำเร็จหรือไม่ ตามค่าเริ่มต้น ให้ตั้งค่าเป็นเท็จ ตั้งค่าเป็นจริงหากคุณต้องการลบสิ่งเหล่านี้
loadingPercent (int) : ความถี่ในการรายงานความคืบหน้าของงานโดยใช้ตัวรายงานความคืบหน้า ตามค่าเริ่มต้น ให้ตั้งค่าเป็น 10 หมายความว่าการอัปเดตจะพิมพ์ทุกๆ 10%
numThreads (int) : จำนวนเธรดที่จะใช้ ตามค่าเริ่มต้นจะตั้งค่าเป็น 0 ซึ่งใช้เธรดที่มีอยู่ทั้งหมดลบ 1
splitPredInputFile (int) : ใช้เฉพาะเมื่อการคาดการณ์ DIA-NN ล้มเหลวเนื่องจากข้อผิดพลาดหน่วยความจำไม่เพียงพอ (137) ตามค่าเริ่มต้น ให้ตั้งค่าเป็น 1 แต่คุณสามารถเพิ่มค่านี้เพื่อระบุจำนวนไฟล์ขนาดเล็กที่ไฟล์อินพุต DIA-NN ควรแบ่งออกเป็นจำนวนได้ แต่ละไฟล์จะถูกทำนายตามลำดับ ทำให้ภาระหน่วยความจำง่ายขึ้น
plotExtension (String) : รูปแบบไฟล์ใดที่แปลงควรอยู่ในรูปแบบ png โดยค่าเริ่มต้น และอนุญาตให้ใช้ pdf ได้ด้วย
features (String) : รายการคุณสมบัติที่จะคำนวณ คำนึงถึงขนาดตัวพิมพ์ และคั่นด้วยเครื่องหมายจุลภาคโดยไม่มีช่องว่างระหว่างกัน ค่าเริ่มต้นคือ "predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS"
spectraPredFile (String) : หากคุณนำการคาดการณ์สเปกตรัมเก่ามาใช้ซ้ำ (เช่นจาก DIA-NN หรือ Koina) คุณสามารถระบุตำแหน่งไฟล์ได้ที่นี่
RTPredFile (String) : เหมือนกับ spectraPredFile แต่สำหรับการคาดการณ์ RT
IMPredFile (String) : เหมือนกับ spectraPredFile แต่สำหรับการคาดการณ์ IM
spectraModel (String) : โมเดลการทำนายสเปกตรัมที่จะใช้
rtModel (String) : เหมือนกับ spectraModel แต่สำหรับ RT
imModel (String) : เหมือนกับ spectraModel แต่สำหรับ IM
useSpectra (boolean) : จะใช้คุณสมบัติตามการทำนายสเปกตรัมหรือไม่ ตั้งค่าเป็นจริงตามค่าเริ่มต้น
useRT (boolean) : จะใช้ฟีเจอร์ที่อิงตามการทำนาย RT หรือไม่ ตั้งค่าเป็นจริงตามค่าเริ่มต้น
useIM (boolean) : จะใช้ฟีเจอร์ที่อิงการทำนาย IM หรือไม่ ตั้งค่าเป็นเท็จตามค่าเริ่มต้น
ppmTolerance (float) : ข้อผิดพลาดส่วน ppm ความอดทน (ค่าเริ่มต้น 20ppm)
matchWithDaltons (boolean) : ว่าจะจับคู่ชิ้นส่วนที่คาดการณ์และสังเกตได้ใน Daltons หรือไม่ (เท็จเริ่มต้น)
DaTolerance (float) : จำนวนดาลตันรอบจุดสูงสุดที่คาดการณ์ไว้เพื่อค้นหาจุดสูงสุดของการทดลอง (ค่าเริ่มต้น 0.05)
useTopFragments (boolean) : ว่าจะกรองการทำนายสเปกตรัมไปยังจุดสูงสุดที่มีความเข้มสูงสุด N หรือไม่ (ค่าเริ่มต้นเป็นจริง)
topFragments (int) : ขึ้นอยู่กับจำนวนแฟรกเมนต์ที่คาดการณ์ไว้ที่ควรใช้สำหรับการคำนวณคุณสมบัติ (ค่าเริ่มต้น 20) ใช้เมื่อ useTopFragments เป็นจริงเท่านั้น
removeRankPeaks (boolean) : ตั้งค่าเป็นจริงตามค่าเริ่มต้น ซึ่งจะกรองส่วนต่างๆ ออกจากสเปกตรัมการทดลองเมื่อจับคู่กันแล้ว หากเป็นเท็จ ชิ้นส่วนทดลองสามารถจับคู่ได้ด้วย PSM หลายตัวจากการสแกนครั้งเดียวกัน
useBasePeak (boolean) : ควรใช้ขีดจำกัดล่างกับการคาดการณ์ MS2 เพื่อใช้เฉพาะส่วนที่มีความเข้มสูงกว่าหรือไม่ (ค่าเริ่มต้นเป็นจริง)
percentBasePeak (float) : เปอร์เซ็นต์ที่ส่วนที่มีความเข้มของเปอร์เซ็นต์ของความเข้มสูงสุดของฐานบางส่วนถูกรวมไว้ในการคำนวณความคล้ายคลึงกัน ใช้เฉพาะในกรณีที่ useBasePeak เป็นจริง (ค่าเริ่มต้น 1)
loessEscoreCutoff (float) : การตัดค่าความคาดหวังที่ใช้สำหรับการส่งผ่านครั้งแรกในการรวบรวม PSM สำหรับการสอบเทียบ RT/IM ค่าเริ่มต้นคือ 10^-3.5 หรือประมาณ 0.000316
rtLoessRegressionSize (int) : จำนวน PSM สูงสุดที่ใช้สำหรับการสอบเทียบ RT LOESS (ค่าเริ่มต้น 5000)
imLoessRegressionSize (int) : เหมือนกับ rtLoessRegressionSize แต่สำหรับ IM (ค่าเริ่มต้น 1,000)
minLoessRegressionSize (int) : จำนวน PSM ขั้นต่ำที่จำเป็นในการพยายามสอบเทียบ LOESS RT/IM (ค่าเริ่มต้น 100) หากมี PSM น้อยกว่าจำนวนนี้ ระบบจะใช้การถดถอยเชิงเส้นแทน
minLinearRegressionSize (int) : จำนวน PSM ขั้นต่ำที่จำเป็นในการพยายามสอบเทียบ RT/IM การถดถอยเชิงเส้น (ค่าเริ่มต้น 10) หากมี PSM น้อยกว่าจำนวนนี้ จะไม่มีการพยายามสอบเทียบ
loessBandwidth (String) : รายการแบนด์วิธที่จะลองปรับเทียบ RT/IM LOESS (ค่าเริ่มต้น 0.01,0.05,0.1,0.2) ซึ่งจะต้องคั่นด้วยลูกน้ำโดยไม่มีช่องว่างระหว่างนั้น
regressionSplits (int) : จำนวนการตรวจสอบข้ามที่ใช้สำหรับการสอบเทียบ RT/IM LOESS (ค่าเริ่มต้น 5)
massesForLoessCalibration (String) : มวลสำหรับการเปลี่ยนแปลงมวลที่ควรเหมาะสมกับเส้นโค้งการปรับเทียบของตัวเอง รายการคั่นด้วยเครื่องหมายจุลภาคโดยไม่มีช่องว่างระหว่างนั้น ควรเขียนมวลให้มีจำนวนหลักเท่ากันกับในไฟล์ PIN
loessScatterOpacity (float) : ความทึบของแปลงกระจายในตัวเลขการสอบเทียบ LOESS จาก 0 ถึง 1 (ค่าเริ่มต้น 0.35)
ไฟล์ .pin พร้อมคุณสมบัติใหม่ ตามค่าเริ่มต้น ไฟล์พินใหม่จะถูกสร้างขึ้นโดยลงท้ายด้วย "_edited.pin" คุณลักษณะเริ่มต้นที่ใช้คือ "unweighted_spectral_entropy", "delta_RT_loess" และ "pred_RT_real_units" หากเปิดใช้งานคุณสมบัติการเคลื่อนที่ของไอออน จะมีการรวม "delta_IM_loess" และ "ion_mobility" ไว้ด้วย
spectraRT.tsv และ spectraRT_full.tsv: ไฟล์อินพุตสำหรับโมเดลการทำนาย DIA-NN
spectraRT.predicted.bin: ไฟล์ไบนารีพร้อมการคาดการณ์จาก DIA-NN ที่จะใช้โดย MSBooster สำหรับการคำนวณคุณสมบัติ หากใช้ FragPipe-PDV ไฟล์เหล่านี้จะถูกใช้เพื่อสร้างแผนมิเรอร์ของสเปกตรัมเชิงทดลองและที่คาดการณ์ไว้
MSBooster สร้างกราฟหลายกราฟที่สามารถใช้เพื่อตรวจสอบเพิ่มเติมว่าข้อมูลของคุณเปรียบเทียบกับการคาดการณ์แบบจำลองอย่างไร
โฟลเดอร์ MSBooster_plots:
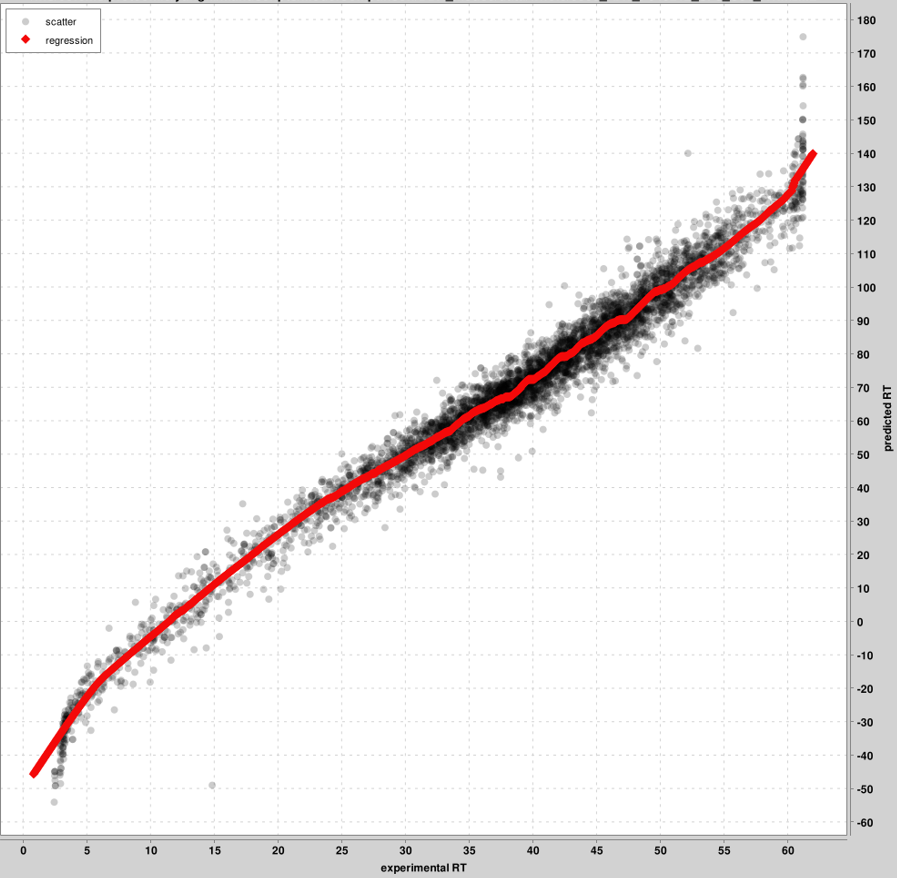
RT_calibration_curves: PSM สูงสุด 5,000 อันดับแรกจะถูกใช้สำหรับการสอบเทียบระหว่างสเกล RT เชิงทดลองและที่คาดการณ์ไว้ PSM ยอดนิยมเหล่านี้จะแสดงอยู่ในกราฟ ไม่ใช่ PSM ทั้งหมด จะมีการสร้างกราฟหนึ่งกราฟต่อไฟล์พิน
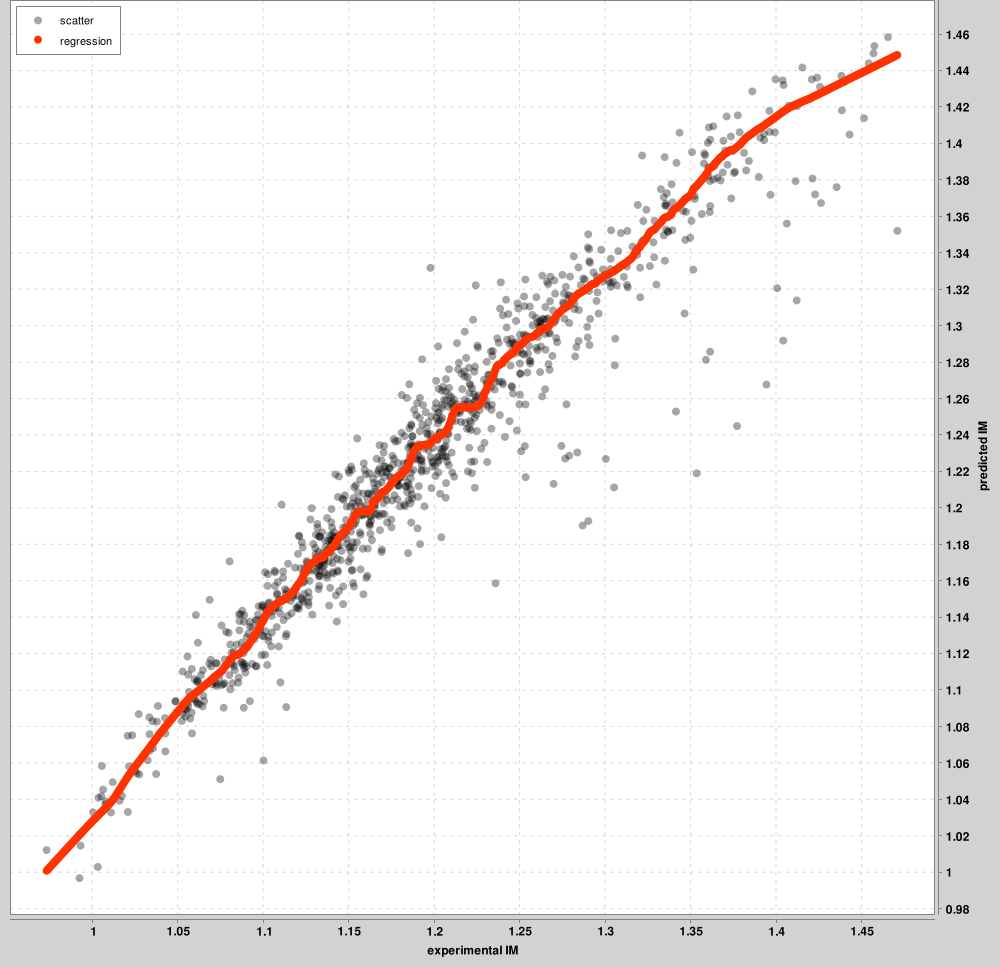
IM_calibration_curves: PSM สูงสุด 1,000 อันดับแรกจะถูกใช้สำหรับการสอบเทียบระหว่างมาตราส่วน IM ทดลองและที่คาดการณ์ไว้ PSM ยอดนิยมเหล่านี้จะแสดงอยู่ในกราฟ ไม่ใช่ PSM ทั้งหมด จะได้เรียนรู้เส้นโค้งที่แยกจากกันสำหรับสถานะการชาร์จแต่ละสถานะ รูปด้านล่างเป็นตัวอย่างของสารตั้งต้นของประจุ 2
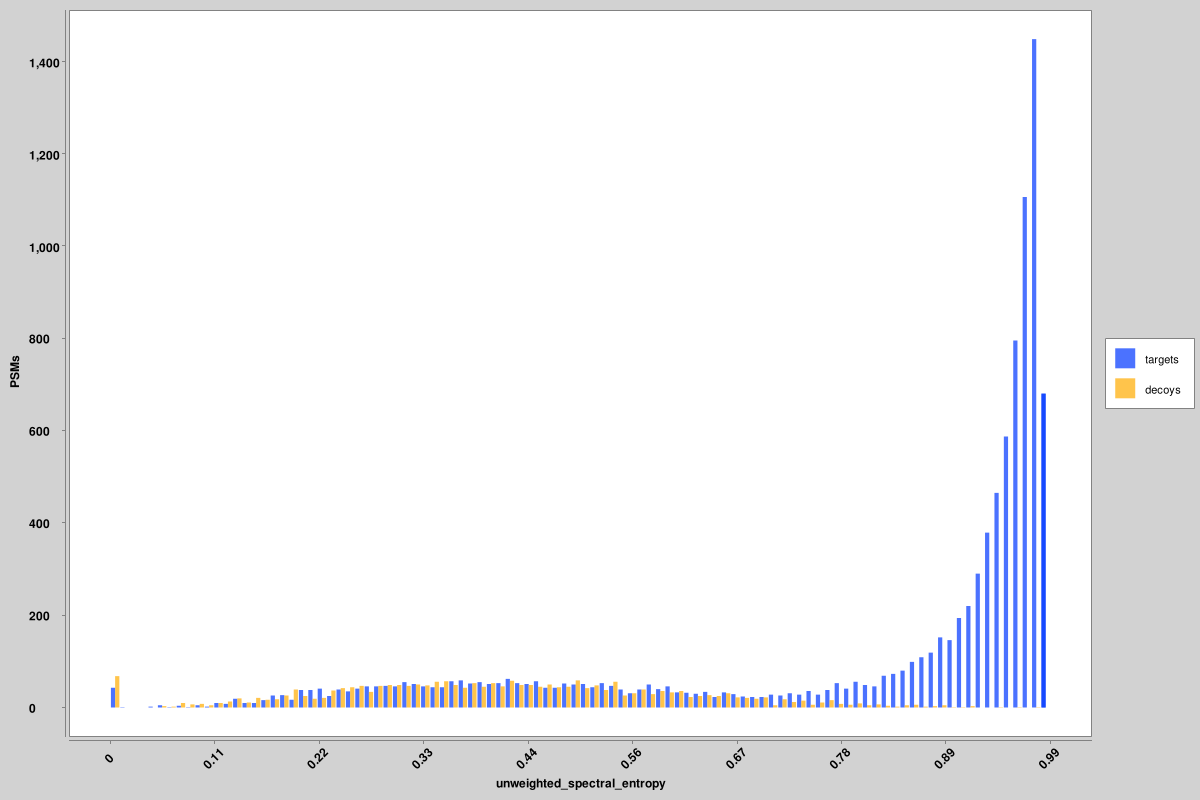
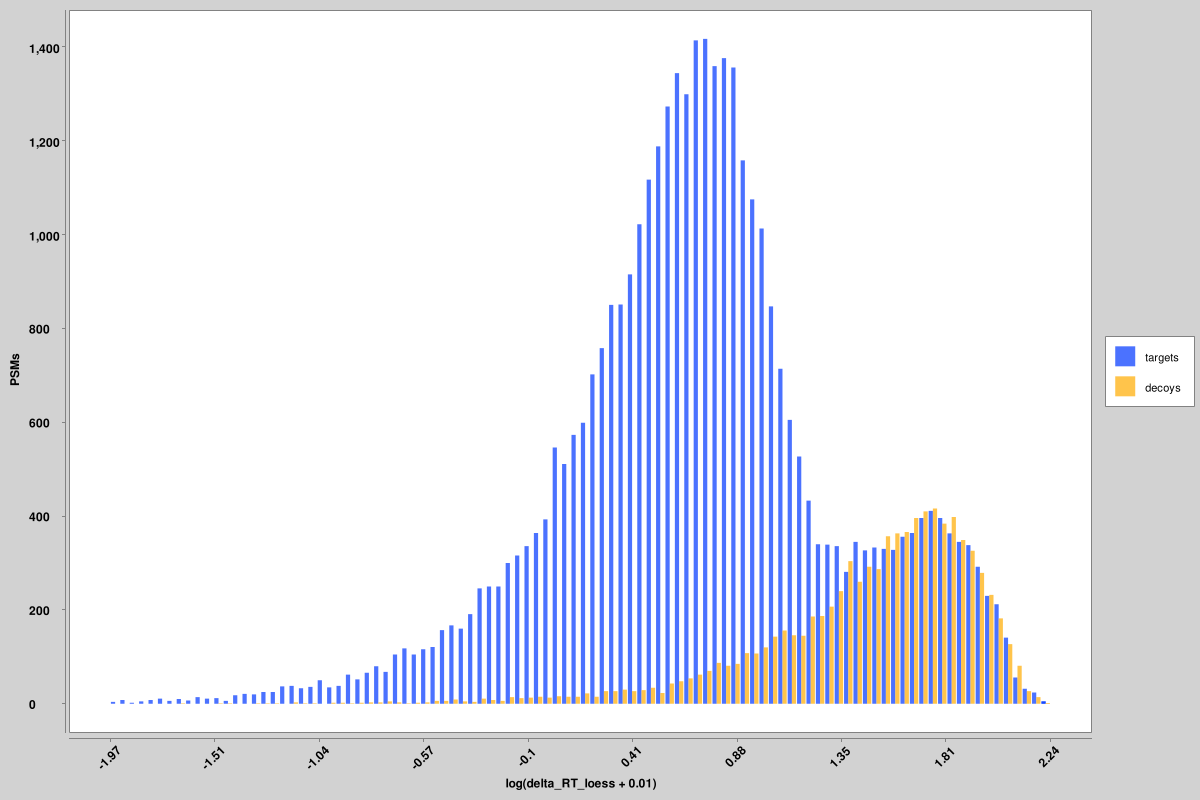
Score_histograms: ฮิสโตแกรมที่ซ้อนทับของเป้าหมายทั้งหมดและหลอก PSM สำหรับแต่ละไฟล์พิน คุณลักษณะบางอย่างได้รับการลงจุดที่นี่ในระดับบันทึกเพื่อให้เห็นภาพที่ดีขึ้นของการกระจายแบบ bimodal ของผลบวกจริงและเท็จ แต่ค่าดั้งเดิมคือค่าที่ใช้ในไฟล์พิน ไม่ใช่เวอร์ชันที่ปรับขนาดบันทึก แสดงให้เห็นว่านี่คือฮิสโตแกรมสำหรับเอนโทรปีสเปกตรัมแบบไม่ถ่วงน้ำหนักและคะแนน delta RT แต่มีการสร้างฮิสโตแกรมที่คล้ายกันสำหรับคุณสมบัติทั้งหมด
ใช้แบบจำลองการทำนายเปปไทด์จาก Koina สำหรับการสร้างฟีเจอร์ MSBooster: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
อ่านคำทำนายจากแบบจำลองใดๆ ผ่านไฟล์ MGF
เอกสารประกอบเกี่ยวกับคุณสมบัติที่ได้รับอนุญาตทั้งหมด และวิธีการควบคุมคุณภาพด้วยเอาต์พุตกราฟิก
โปรดอ้างอิงสิ่งต่อไปนี้เมื่อใช้ MSBooster: https://www.nature.com/articles/s41467-023-40129-9