นี่คือพื้นที่เก็บข้อมูลอย่างเป็นทางการสำหรับ "แบบจำลองเดียวที่จะปกครองพวกเขาทั้งหมด: สู่การแบ่งส่วนสากลสำหรับรูปภาพทางการแพทย์พร้อมข้อความแจ้ง"
เป็นโมเดลการแบ่งส่วนสากลที่เสริมความรู้ ซึ่งสร้างขึ้นจากการรวบรวมข้อมูลที่ไม่เคยมีมาก่อน (ชุดข้อมูลการแบ่งส่วนทางการแพทย์ 3 มิติสาธารณะ 72 ชุด) ซึ่งสามารถแบ่งกลุ่ม 497 คลาสจาก 3 รูปแบบที่แตกต่างกัน (MR, CT, PET) และ 8 บริเวณร่างกายมนุษย์ โดยได้รับแจ้งจากข้อความ (กายวิภาค คำศัพท์)
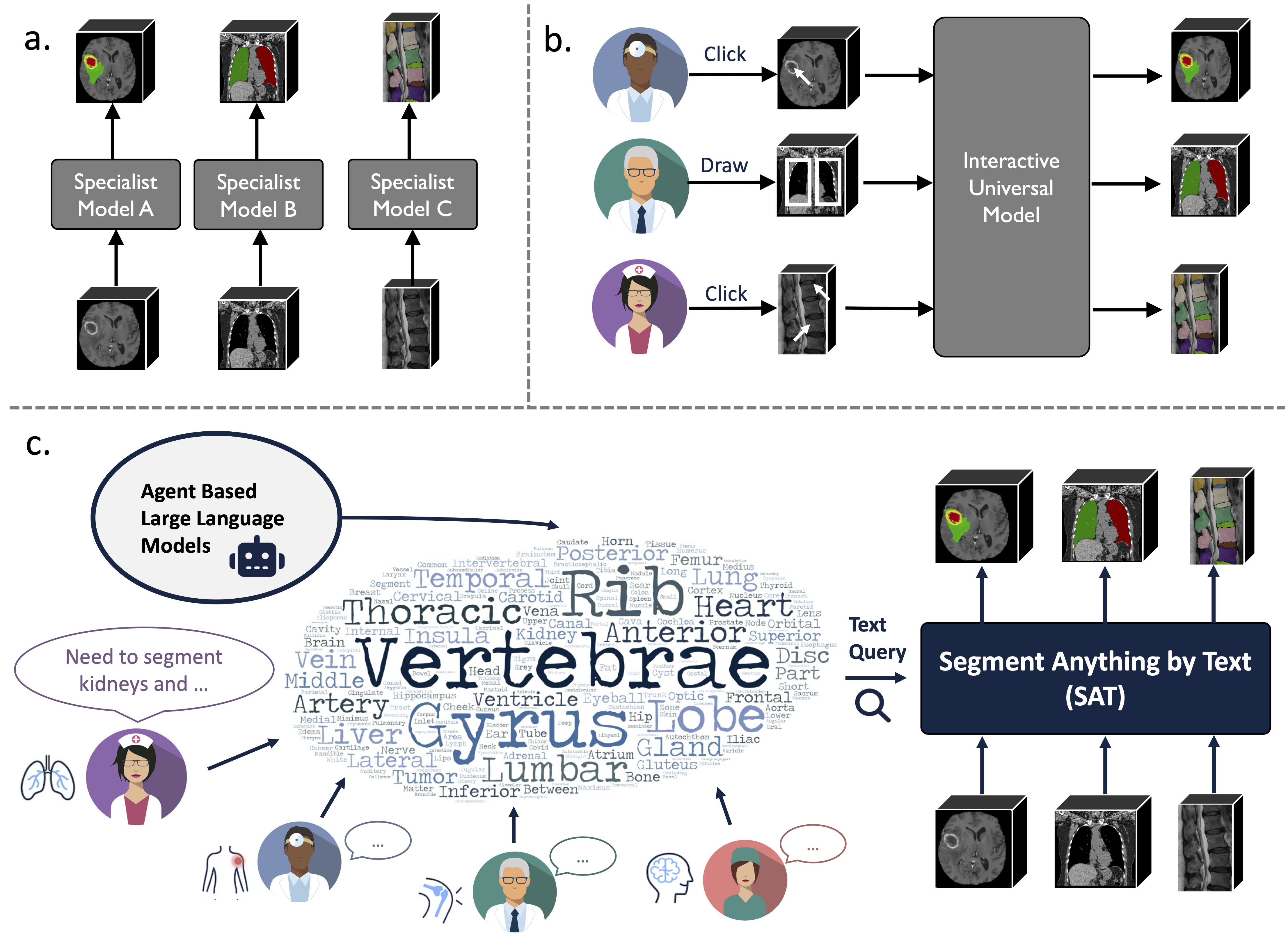
อาจมีประสิทธิภาพและมีประสิทธิภาพมากกว่าการฝึกอบรมและการปรับใช้โมเดลเฉพาะทางต่างๆ ค้นหาข้อมูลเพิ่มเติมบนเว็บไซต์หรือกระดาษของเรา
2024.08 ? จาก SAT และแบบจำลองภาษาขนาดใหญ่ เราสร้างชุดข้อมูลการตีความ CT หน้าอก 3 มิติแบบครอบคลุมขนาดใหญ่และมีคำแนะนำตามภูมิภาค ประกอบด้วยการแบ่งส่วนระดับอวัยวะสำหรับ 196 หมวดหมู่ และรายงานแบบหลายรายละเอียด โดยแต่ละประโยคจะยึดตามการแบ่งส่วนที่เกี่ยวข้อง ตรวจสอบได้ที่กอดเฟซ
2024.06 ? เราได้เผยแพร่โค้ดเพื่อสร้าง SAT-DS ซึ่งเป็นคอลเลกชันของชุดข้อมูลการแบ่งเซ็กเมนต์สาธารณะ 72 ชุด ประกอบด้วยรูปภาพ 3 มิติมากกว่า 22K, มาสก์การแบ่งเซ็กเมนต์ 302K และคลาส 497 รายการจาก 3 รูปแบบที่แตกต่างกัน (MRI, CT, PET) และบริเวณร่างกายมนุษย์ 8 แห่ง เราสร้าง SAT นอกจากนี้เรายังมีลิงก์ดาวน์โหลดทางลัดสำหรับชุดข้อมูล 42/72 ซึ่งได้รับการประมวลผลล่วงหน้าและจัดทำแพ็กเกจโดยเราเพื่อความสะดวกของคุณ พร้อมสำหรับการใช้งานทันทีเมื่อดาวน์โหลดและแตกไฟล์ ตรวจสอบ repo นี้เพื่อดูรายละเอียด
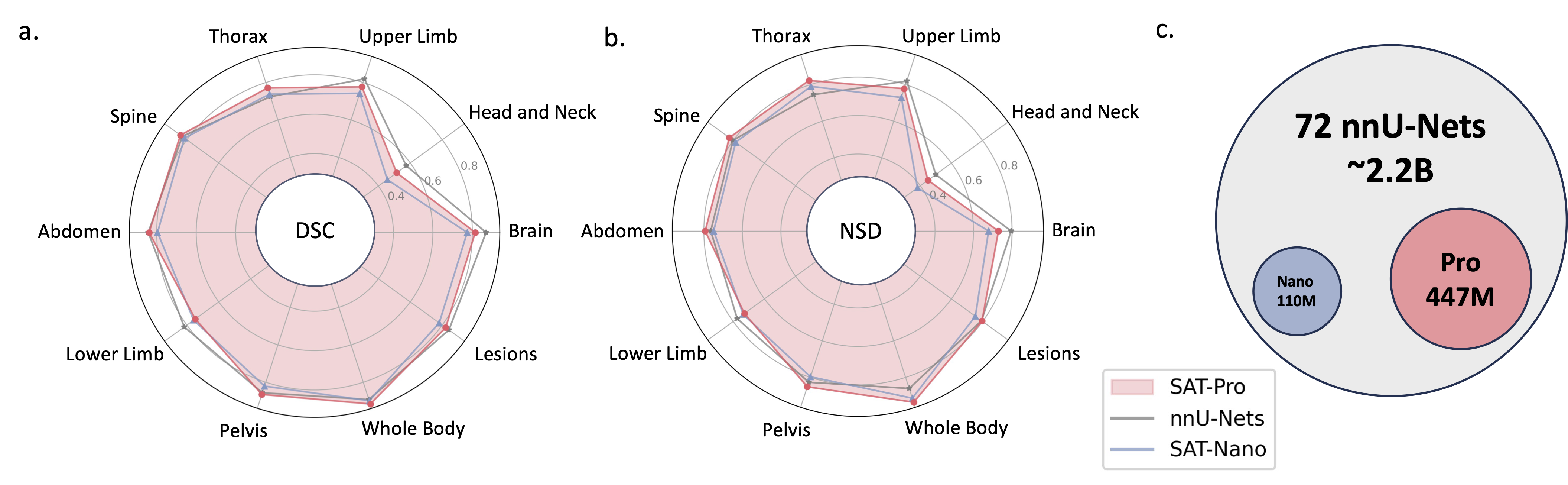
2024.05 ? เราฝึก SAT เวอร์ชันใหม่ด้วยขนาดโมเดลที่ใหญ่ขึ้น ( SAT-Pro ) และชุดข้อมูลมากขึ้น ( 72 ) และตอนนี้รองรับ 497 คลาสแล้ว! นอกจากนี้เรายังต่ออายุ SAT-Nano และเปิดตัว SAT-Nano บางรูปแบบ โดยอิงตามแบ็คโบนภาพที่แตกต่างกัน (U-Mamba และ SwinUNETR) และตัวเข้ารหัสข้อความ (MedCPT และ BERT-Base) สำหรับรายละเอียดเพิ่มเติมเกี่ยวกับการอัปเดตนี้ โปรดดูเอกสารใหม่ของเรา
การใช้งาน U-Net ขึ้นอยู่กับเวอร์ชันที่กำหนดเองของสถาปัตยกรรมเครือข่ายแบบไดนามิก เพื่อติดตั้ง:
cd model
pip install -e dynamic-network-architectures-main
ข้อกำหนดสำคัญอื่นๆ:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
คุณต้องติดตั้ง mamba_ssm หากคุณต้องการ SAT-Nano รุ่น U-Mamba
S1. สร้างสภาพแวดล้อมตาม requirements.txt
เอส2. ดาวน์โหลดจุดตรวจสอบ SAT และ Text Encoder จาก Huggingface
S3. เตรียมข้อมูลเป็นไฟล์ jsonl ตรวจสอบการสาธิตใน data/inference_demo/demo.jsonl
image (เส้นทางไปยังรูปภาพ), labe (ชื่อของเป้าหมายการแบ่งส่วน), dataset (ชุดข้อมูลที่กลุ่มตัวอย่างเป็นสมาชิก) และ modality (ct, mri หรือ pet) จำเป็นสำหรับแต่ละตัวอย่างต่อกลุ่ม รูปแบบและคลาสที่ SAT รองรับสามารถพบได้ในตารางที่ 12 ของบทความนี้
orientation_code (การวางแนว) คือ RAS ตามค่าเริ่มต้น ซึ่งเหมาะสมกับภาพส่วนใหญ่ในระนาบแนวแกน สำหรับรูปภาพในระนาบทัล (เช่น การตรวจกระดูกสันหลัง) ให้ตั้งค่านี้เป็น ASR รูปภาพอินพุตควรมีรูปร่าง H,W,D รหัสกระบวนการข้อมูลของเราจะทำให้รูปภาพอินพุตเป็นมาตรฐานในแง่ของการวางแนว ความเข้ม ระยะห่าง และอื่นๆ สามารถดูรูปภาพที่ประมวลผลสำเร็จสองรูปได้ใน demoprocessed_data ตรวจสอบให้แน่ใจว่าการทำให้เป็นมาตรฐานนั้นทำอย่างถูกต้องเพื่อรับประกันประสิทธิภาพของ SAT
S4. เริ่มต้นการอนุมานด้วย SAT-Pro ?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d คือขนาดแบตช์ของแพตช์รูปภาพอินพุต และจำเป็นต้องปรับตามหน่วยความจำ GPU (ตรวจสอบตารางด้านล่าง) --max_queries แนะนำให้ตั้งค่าให้ใหญ่กว่าคลาสในชุดข้อมูลการอนุมาน เว้นแต่หน่วยความจำ GPU ของคุณจะถูกจำกัดมาก
| แบบอย่าง | ขนาดแบตช์_3d | หน่วยความจำจีพียู |
|---|---|---|
| SAT-Pro | 1 | ~34GB |
| SAT-Pro | 2 | ~ 62GB |
| SAT-นาโน | 1 | ~24GB |
| SAT-นาโน | 2 | ~36GB |
S5. ตรวจสอบ --rcd_dir สำหรับเอาต์พุต ผลลัพธ์จะถูกจัดเรียงตามชุดข้อมูล สำหรับแต่ละกรณี จะพบรูปภาพอินพุต ผลการแบ่งส่วนแบบรวม และโฟลเดอร์ที่มีการแบ่งส่วนของแต่ละคลาส เอาต์พุตทั้งหมดจะถูกจัดเก็บเป็นไฟล์ nifiti คุณสามารถเห็นภาพได้โดยใช้ ITK-SNAP
หากคุณต้องการใช้ SAT-Nano ที่ได้รับการฝึกอบรมบนชุดข้อมูล 72 ชุด เพียงแก้ไข --vision_backbone เป็น 'UNET' และเปลี่ยน --checkpoint และ --text_encoder_checkpoint ตามลำดับ
สำหรับ SAT-Nano รุ่นอื่นๆ (ฝึกกับชุดข้อมูล 49 ชุด):
UNET-ของเรา: set --vision_backbone 'UNET' และ --text_encoder 'ours' ;
UNET-CPT: ตั้งค่า --vision_backbone 'UNET' และ --text_encoder 'medcpt' ;
UNET-BB: ตั้งค่า --vision_backbone 'UNET' และ --text_encoder 'basebert' ;
UMamba-CPT: ตั้งค่า --vision_backbone 'UMamba' และ --text_encoder 'medcpt' ;
SwinUNETR-CPT: ตั้งค่า --vision_backbone 'SwinUNETR' และ --text_encoder 'medcpt' ;
การเตรียมตัวก่อนเริ่มการฝึกอบรม:
sh/ เพื่อเริ่มกระบวนการฝึกอบรม ยกตัวอย่าง SAT-Pro: sbatch sh/train_sat_pro.sh
สิ่งนี้ยังจำเป็นต้องสร้างข้อมูลทดสอบตาม repo นี้ด้วย คุณอาจอ้างถึงสคริปต์สเลม sh/evaluate_sat_pro.sh เพื่อเริ่มกระบวนการประเมิน:
sbatch sh/evaluate_sat_pro.sh
หากคุณใช้รหัสนี้เพื่อการวิจัยหรือโครงการของคุณ โปรดอ้างอิง:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}